今天在浏览R包网站的时候又发现一个好包——broom包。
这个包干嘛的了?
查看R包介绍,这个包可以将统计结果转换为数据框格式,比如说逻辑回归模型、Cox回归模型的结果,然后可以方便的输出来。
看到这里,是不是觉得很惊喜!
回归模型是临床研究中最常用的统计方法之一,如果手动提取数据结果的话肯定费时又费力,现在可以使用这个包来快捷方便的导出结果数据。
下面来学习下这个包。
1. 安装和加载R包
可以直接从CRAN上安装,很顺利。
install.packages("broom") # 安装R包
library(broom) # 加载R包
library(survival) # cox回归模型需要
2. 加载演示数据集
使用colon数据集进行演示。
data(colon) # 加载数据集
str(colon) # 查看数据集结构
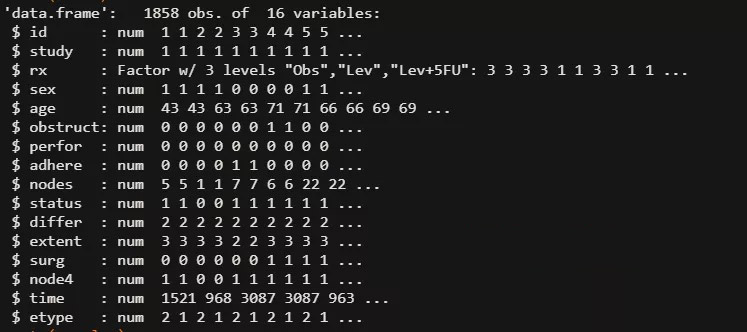
查看数据集介绍,可以知道有些变量是分类变量,但是加载进来的数据集变量还是数字型,我们处理一下。
mycolon <- colon %>% # 创建新数据集 新变量
transmute(time,
status,
Age = age,
Sex = factor(sex, levels = c(0,1),
labels = c("Female", "Male")),
Obstruct = factor(colon$obstruct),
Differ = factor(colon$differ),
Extent = factor(colon$extent),
Adhere = factor(colon$adhere),
Perfor = factor(colon$perfor),
Surg = factor(colon$surg),
Nodes = nodes,
Rx = factor(colon$rx))
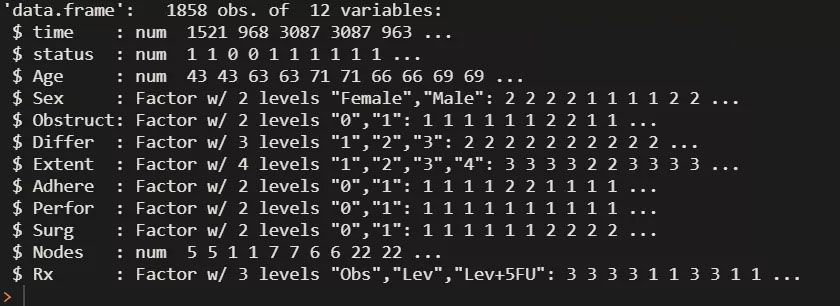
如上代码所示,我将colon数据集中的部分变量转换了因子,并新建了一个数据集mycolon,下面使用mycolon数据集进行演示。
3. 拟合模型
本篇推文演示逻辑回归模型和Cox回归模型的结果怎么快速导出,毕竟这两种模型是最常用的,也是很实用的。
先拟合下这两个模型。
3.1 拟合Cox回归模型
使用survival包的coxph()函数拟合下Cox回归模型。
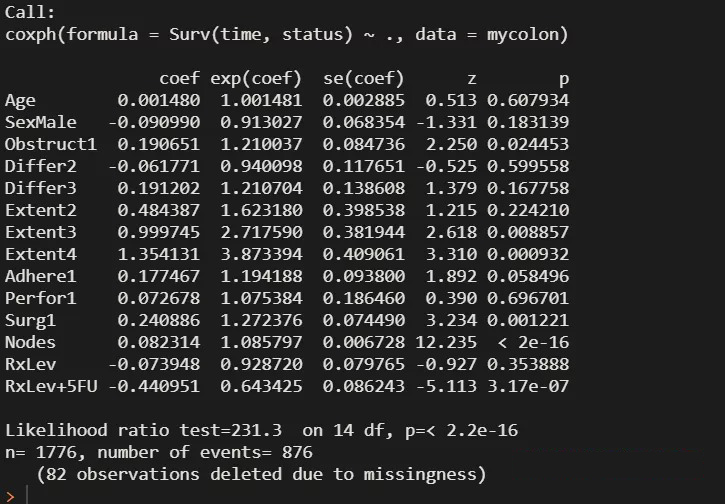
coxphmodel <- coxph(Surv(time, status) ~ ., mycolon)
coxphmodel # 查看模型结果
~符号后的.表示使用数据集中的全部变量纳入回归模型。
3.2 拟合逻辑回归模型
使用glm()函数来拟合逻辑回归模型。
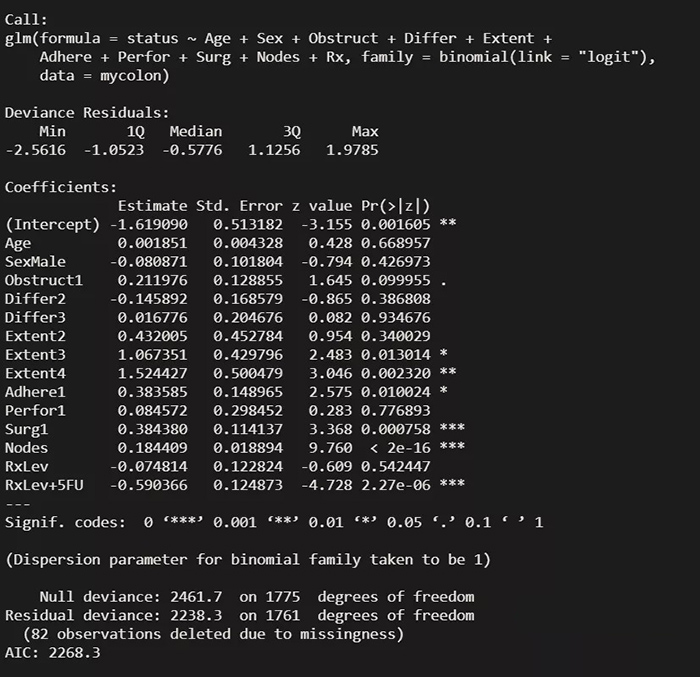
glmmodel <- glm(status ~ Age + Sex + Obstruct + Differ + Extent + Adhere + Perfor + Surg + Nodes + Rx,
binomial(link="logit"), data = mycolon)
summary(glmmodel) # 查看模型结果
4. broom包怎么用?
首先这个包有三种不同的整理结果的方法。
4.1 tidy()函数
这个函数主要用来整理模型的统计结果,比如说系数和P值,并构建成一个数据集。
我们整理下Cox模型的统计结果,直接将拟合模型对象放入tidy()函数中即可。
coxphtable <- tidy(coxphmodel)
coxphtable
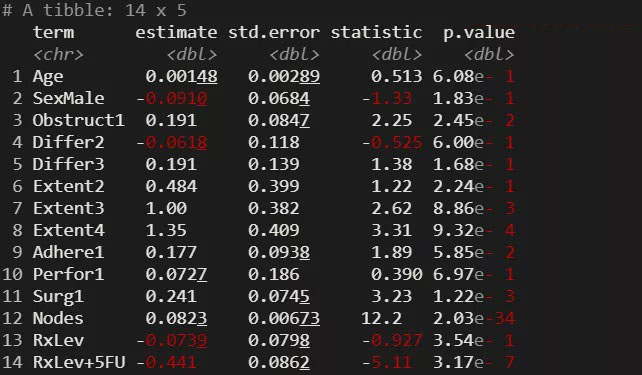
如上所示,上面的数据框里已经是整洁的数据结果了。
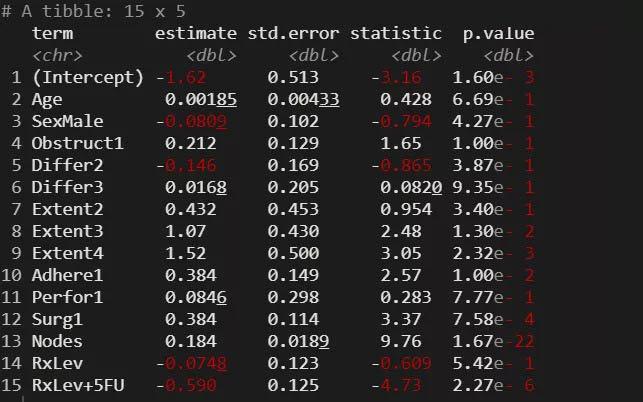
逻辑回归模型结果的整理跟Cox模型是一样的。
glmtable <- tidy(glmmodel)
glmtable
4.2 augment()函数
这个函数主要在建模的原始数据集中添加一些列变量,比如说预测、残差等。
glmaug <- augment(glmmodel, data = mycolon)
glmaug
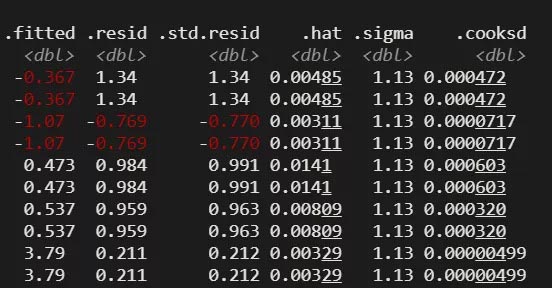
这些是在建模数据集中增加的列变量。
4.3 glance()函数
主要整理模型的其他统计结果,比如说R²,校正R²,残余标准差等。
glmgla <- glance(glmmodel)
glmgla
# A tibble: 1 x 8
# null.deviance df.null logLik AIC BIC deviance df.residual nobs
# <dbl> <int> <dbl> <dbl> <dbl> <dbl> <int> <int>
# 1 2462. 1775 -1119. 2268. 2350. 2238. 1761 1776
5. 导出结果
上面的模型已经整理成了数据集的形式,可以直接使用openxlsx包的write.xlsx()函数将结果导出为Excel文件。
library(openxlsx) # 加载R包
write.xlsx(coxphtable, "coxphtable.xlsx")
# 前面是R中的对象,后面是保存在工作目录的文件名称
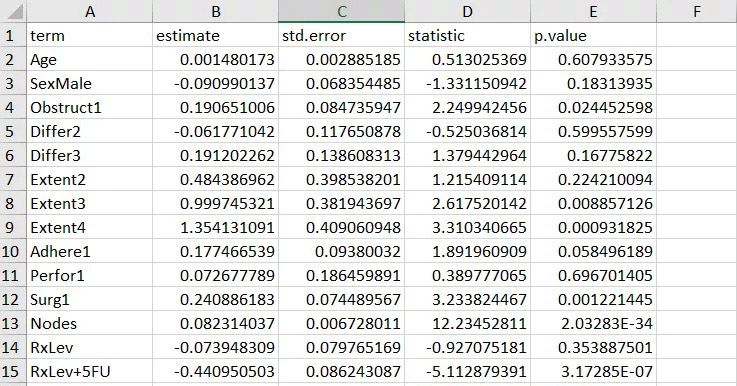
write.xlsx(glmtable, "glmtable.xlsx")
# 前面是R中的对象,后面是保存在工作目录的文件名称
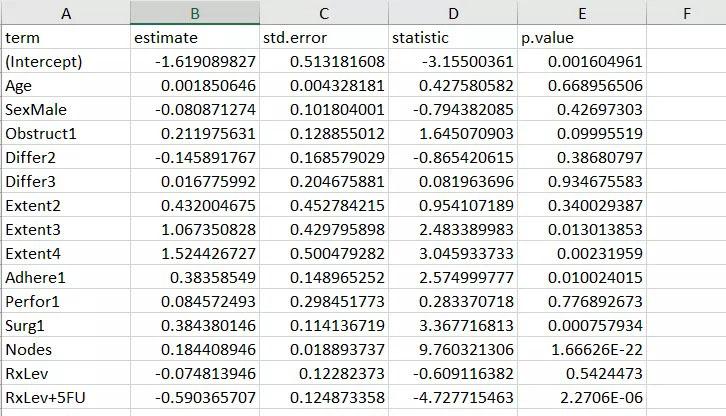
如上图所示,就将逻辑回归模型和Cox回归模型的结果输出来了。
其他结果的导出跟上面一样的。
最后,除了导出逻辑回归和Cox回归的结果外,假设检验如t.test、cor.test和wilcox.test的结果也可以使用这个包导出。
这个包有兴趣的可以查看帮助文件再仔细学习学习。
参考资料
survival包帮助文件 broom包帮助文件 openxlsx包帮助文件














