背景介绍
自2003年人类基因组草图公布以来,基因组测序技术取得了巨大的进步,多种大规模平行测序(MPS)平台也如雨后春笋般迅猛增长。其中,全基因组测序(Whole Genome Sequencing,WGS)可无偏颇地检测各类基因突变,以提供整个基因组的全面视图,也是目前临床及基础研究中常用到的技术之一。
多年来,大规模平行测序市场一直由Illumina等寡头垄断,大大限制了国内相关产业的发展。为改变这一现状,近年来,以华大智造为代表的国内基因测序企业在国产测序仪研发、生产方面作出了不懈努力,也取得了一定的进步。华大智造在短短几年内相继推出了拥有完全自主知识产权、具有国际先进水平的测序仪,例如MGISEQ-200、MGISEQ-2000、DNBSEQ-T7等。该系列国产测序仪的优势是在保证相同reads读长和产量的同时节省成本,以DNBSEQ-T7为例,其在单次运行产生6TB测序数据的背景下,成本低至4.5美元/ GB。此外,华大智造仍在持续推出一系列新的MPS设备,并且期望在提供高质量测序数据的前提下,努力实现数据产出更快、价格更低。
目前 ,华大智造的测序仪已经被成功应用于多个领域的研究。
2021年4月17日,来自韩国的研究团队在Genes & Genomics杂志在线发表了题为“Comparison between MGI and Illumina sequencing platforms for whole genome sequencing”的研究文章,分析比较了华大智造MGISEQ-2000和DNBSEQ-T7平台与Illumina的NovaSeq 6000平台在全基因组测序层面的性能。通过多方面评估,研究人员发现各测序平台在片段大小分布、基因覆盖率和表达变异检测方面的表现都很相似,表明了MGISEQ-2000和DNBSEQ-T7平台的性能表现已经达到了国际领先水平。

文章发表在Genes & Genomics
主要研究内容
研究人员选择了Illumina的NovaSeq 6000和华大智造的MGISEQ-2000和DNBSEQ-T7,以评估MGISEQ-2000和DNBSEQ-T7测序仪器在全基因组测序上的性能表现。同时,利用这三个平台对来自韩国肺癌患者的正常和肿瘤组织进行测序,并对产生的数据做了平行比较。
通过评估三个平台原始数据的质量发现,在质量控制层面,三个平台均表现出相似的质量和基因组覆盖深读。三个平台中,Q20都大于99.7%,同时,GC含量也类似,约为41%。
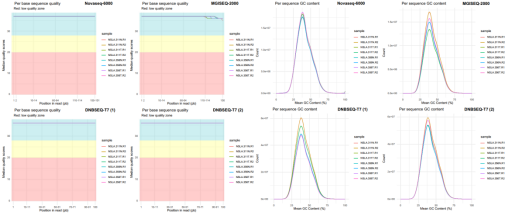
图1. 原始测序数据质量的比较,图片来源:Genes & Genomics
研究团队利用BWA-MEM方法将全基因组序列对比到参考基因组。结果显示,所有样品的覆盖率均在99.9%以上。
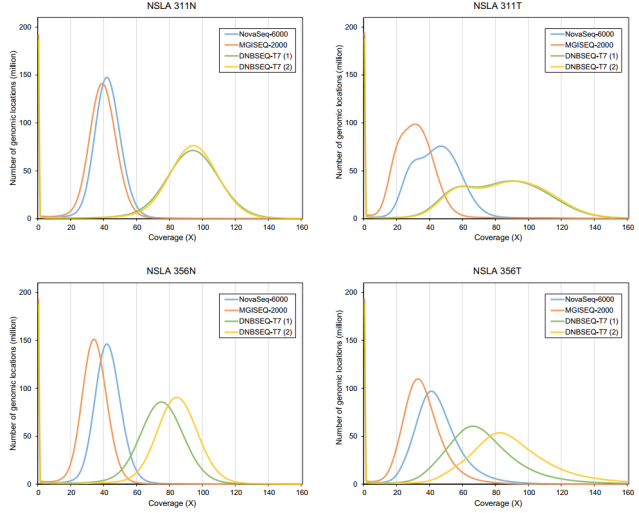
图2. 测序平台的覆盖率比较,图片来源:Genes & Genomics
此外,研究人员利用相同的分析方法分析了三个平台的SNVs和Indels,结果显示,NovaSeq 6000与MGISEQ-2000测序结果最相似,重叠比率最高 (每个样本分别为92.0%和93.0%)。
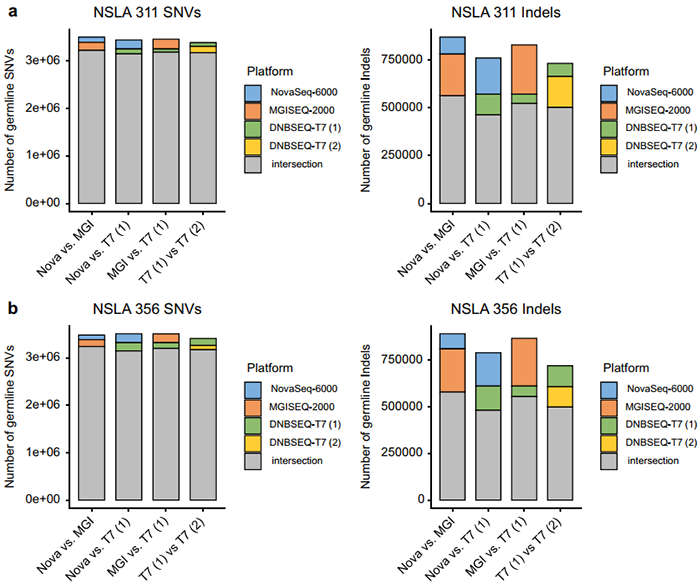
图3. SNVs和Indels检测的比较,图片来源:Genes & Genomics
为了确定仅在一个平台上被检测到的种系变异是否为真正的变异,而不是由于平台本身产生的测序误差,研究人员分析了低分数的种系变异,并设定当该变异仅在一个平台上出现,那么就被认为是无效的。结果发现,NovaSeq 6000测序数据中的无效变异比例约为2.54%,MGISEQ-2000为0.45%。
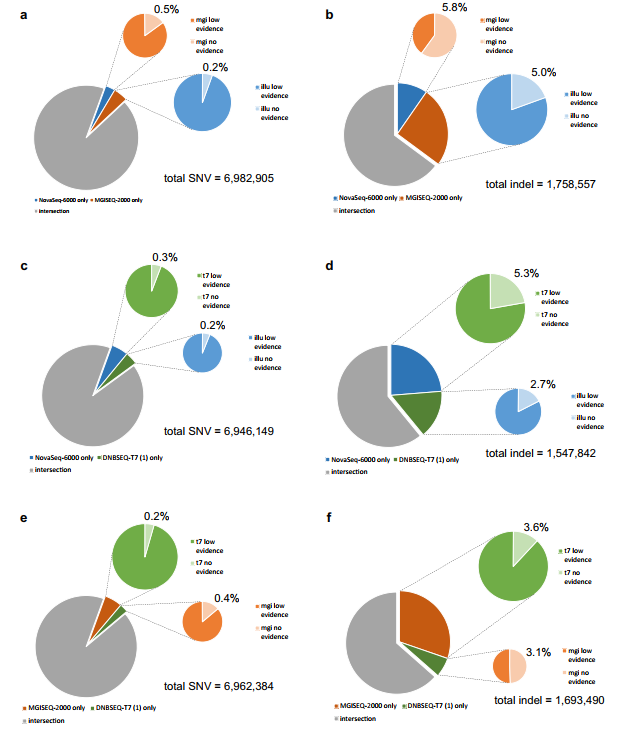
图4. 无效突变体比例的分析,图片来源:Genes & Genomics
研究总结
该研究对三种测序平台在全基因组测序层面进行了比较分析,通过多方面的数据支撑,研究团队认为上述三个平台都提供了高质量的测序数据和足够的基因组覆盖率,均适用于全基因组测序分析。就变异体检测层面,MGISEQ-2000与NovaSeq 6000性能最相似。此外,与NovaSeq 6000相比,DNBSEQ-T7还能够检测到更多的8 bp以上的indel。总之, NovaSeq 6000、MGISEQ-2000和DNBSEQ-T7三个平台的测序性能具有高度的一致性。
参考资料
[4] Zhang, X., Zhong, H., Li, Y. et al. Sex- and age-related trajectories of the adult human gut microbiota shared across populations of different ethnicities. Nat Aging 1, 87–100 (2021). https://doi.org/10.1038/s43587-020-00014-2














