随着单细胞测序技术的发展,产生了越来越多的单细胞组学数据,包括基因表达谱、染色体可及性、DNA 甲基化等等。如何将各种组学整合在一起,消除批次间差异,成为构建单细胞细胞多组学蓝图,对单细胞数据进行下游分析的一项重大挑战。现有方法主要有 Seurat 和 Harmony,这两种方法并非针对不同组学数据的整合而开发的,且都需要对待整合的数据重新进行从头分析。
2021 年 4 月 19 日,加州大学 Joshua 实验室在 Nature Biotechnology 杂志上发表文章 Iterative single-cell multi-omic integration using online learning,开发了一种新的高效单细胞多组学数据整合算法 —— 在线 iNMF(在线 intergrative non-negative matrix factorization, 在线非负矩阵分解)。该算法创新性在单细胞数据分享中引入「在线学习」思想,可分批次处理持续产生的单细胞数据,实现在笔记本电脑上构建单细胞多组学图谱。图片来源:Nature Biotechnology
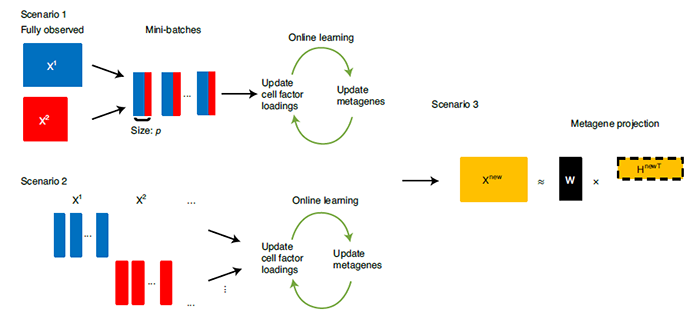
该方法可以在三种情境下整合单细胞多组学数据,第一种是已有完整大数据集,算法将其拆分成多个小数据集分别计算出元基因(metagene)和细胞负荷(cell factor loading),适合所有单细胞数据当前可用的情况;第二种是陆续产生新数据的情况,在线算法将实时根据新输入的数据进行参数更新,适合不断整合新测序的细胞;第三种是不根据新数据更新原基因,而是根据已有参考集得出的元基因,计算新数据的细胞因子负荷,适合根据大型精选参考图谱快速查询数据集。图片来源:Nature Biotechnology由于在线 iNMF 是在原先的批量 iNMF 算法基础上,引入「在线学习」开发的新算法,研究人员首先比较了在线 iNMF 与批量 iNMF 的性能差异。在训练数据集与测试数据集上,在线 iNMF 均表现出更快的收敛速度。并且在单批次数据量较小的情况下,在线 iNMF 算法的迭代速度对批次大小变化不敏感。图片来源:Nature Biotechnology
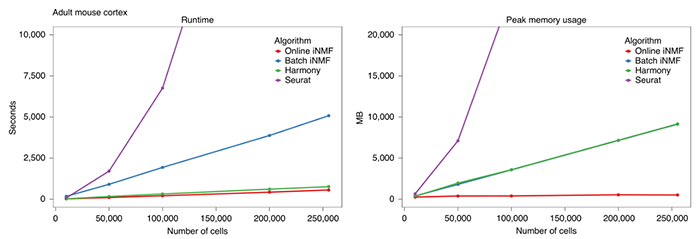
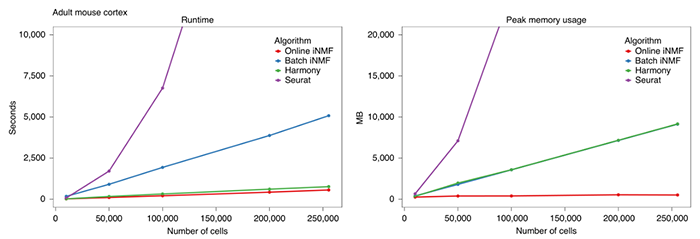
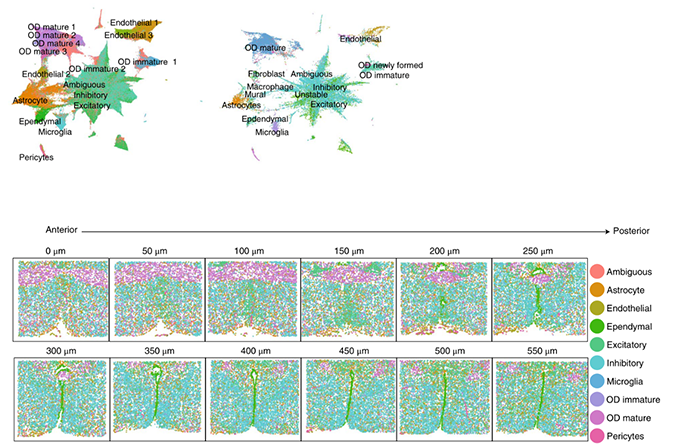
为了验证在线 iNMF 的性能,研究人员将之与现有的数据整合算法进行比较。最近一项研究将 14 种现有的单细胞数据整合方法进行比较后发现,Harmony, Seurat 和 LIGER(iNMF 为该方法的一种扩展)这三种方法在不同数据集中综合来看,具有最优的数据对齐与细胞聚类表现。将在线 iNMF 与批次 iNMF、Harmony 和 Seurat 进行对标比较,并模拟拆分数据以及不同样本的实验数据进行验证。结果显示,在线 iNMF 在速度大幅提高并且占用内存减少的情况下,表现出与这些方法相似或者更优的数据对齐与聚类性能。并且随着数据量的增大,这种优势变得更加明显。图片来源:Nature Biotechnology在线 iNMF 算法可对齐不同批次 / 组学的数据研究人员在普通笔记本电脑中实现了一百多万细胞的单细胞测序数与空间转录组数据(Slide-seq、MERFISH)的整合。空间转录组数据能够提供带有空间位置信息的基因表达谱数据,然而基因捕获量不如单细胞测序数据。通过整合这两种组学数据,可以获得更高深度的空间全转录组数据。研究人员利用第三类场景下的在线 iNMF 算法,解析空间组数据在单细胞测序数据上的细胞因子负荷,能够获取单细胞数据中不同细胞簇在空间上的分布情况。通过整合分析结果能够看出,空间转录组数据与单细胞数据的细胞聚类结果非常相似,并且细胞簇在空间位置的分布与已知下丘脑结果非常吻合,从而验证了在线 iNMF 能够有效区分不同位置上富集的不同类型细胞。图片来源:Nature Biotechnology在线 iNMF 算法的另一项优势在于,在场景二下,可以允许新数据出现时,基于原有数据的分析结果,更新元基因矩阵,而不需要重新访问已经处理过的细胞。在涉及多个小组的大型研究项目中,存在异步生成的实验数据,此功能对这类任务尤其有用。在文章中,研究人员通过一个包含四个组学(scRNA-seq,snRNA-seq,snATAC-seq 及 snmC-seq)的单细胞多组学数据集对此功能进行了演示。通过依次整合数据集中的不同组学,可以看到 UMAP 图中的细胞聚类结果在不断迭代优化。最终四个组学整合产生的数据对齐分数为 0.786,k 近邻批次效应测试分数为 0.324。相比之下,Seurat 仅整合 scRNA-seq 与 snATAC-seq 数据,得到的数据对齐分数为 0.481,k 近邻批次效应测试分数为 0.231。此外经测试发现,在线 iNMF 算法对具有数据整合的顺序具有极强的鲁棒性。在线学习算法最大的特点是能够在新数据到来时,在不重复计算的前提下与已有处理结果进行合并。此功能对大型分布式协作工作尤其有用。在未来将有越来越多如 BRAIN Initiative,Human Body Map 及 Human Cell Atlas 的大规模单细胞多组学图谱项目,在此类项目中研究人员需要不断整合新的测序数据以构建全面的细胞图片,因而在线 iNMF 的迭代式数据整合方法将会越来越能够发挥用处。1. Gao, C. , and J. D. Welch . Iterative Refinement of Cellular Identity from Single-Cell Data Using 在线 Learning. 2020.2. Stuart, T. , et al. "Comprehensive Integration of Single-Cell Data." Cell 177.7(2019):1888-1902.e21.3. Stuart, T. , and R. Satija . "Integrative single-cell analysis." Nature Reviews Genetics (2019).4. Korsunsky, I. , et al. "Fast, sensitive and accurate integration of single-cell data with Harmony." Nature Methods 16.4(2019):1-8.5. Tran, Htn , et al. "A benchmark of batch-effect correction methods for single-cell RNA sequencing data." Genome biology 21.1(2020).