2021年4月5日,房刚课题组在Nature Methods发表文章Discovering multiple types of DNA methylation from bacteria and microbiome using nanopore sequencing,开发了新方法NanoDisco,利用纳米孔测序同时检测多种细菌DNA甲基化motifs,并利用细菌DNA甲基化多样性增强菌群分析清晰度。
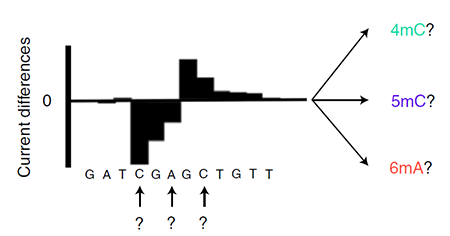
1. 对纳米孔测序检测DNA甲基化的深入理解: 迄今为止 >4000株被破译的细菌的表观遗传组几乎完全是基于PacBio三代测序平台的。此平台对细菌中的6mA和4mC motifs有非常高的可靠度和灵敏度,而对5mC的检测效率不佳。另一种三代测序技术,(Nanopore 纳米孔测序)已在人类基因组5mC检测中逐渐广泛使用,但是现有的技术大多专门针对于CpG 中的5mC检测。房刚课题组通过对大量细菌5mC motif的分析发现不同序列motif中的5mC在纳米孔测序中的信号有很高的异质性 (图1);同样,6mA和4mC信号也存在很高的异质性。也就是说,现有的针对CpG 5mC(或少量具体序列)的纳米孔测序检测方法并不能有效适用于更广泛的DNA甲基化检测,即无法有效的检测细菌中常见的三种DNA甲基化。图1:不同序列motif中的6mA, 4mC 和 5mC在纳米孔测序中的信号有很高的异质性。2. De novo(无须先验知识)发现细菌三种DNA甲基化的新方法。细菌DNA甲基化分析的第一步是de novo找出全部methylation motifs。大家比较熟悉的是大肠杆菌中广泛存在的G6mATC, C5mCWGG。平均来说,每个细菌菌株有三个不同的methylation motifs, 但多的有20多个,比如幽门杆菌。对一个未知的motif, 需要识别出具体甲基化类型(6mA, 4mC, 或5mC)并找到被甲基化的碱基 (图2)。考虑到细菌DNA甲基化的一些特性,新方法NanoDisco的设计使用了一个多标签分类的机器学习架构。经过大量的验证,NanoDisco可以可靠的de novo找到细菌和菌群中的6mA, 4mC, 5mC motifs和其中的甲基化位点。作者认为这一功能会大大帮助更广泛的、更完整的研究细菌DNA甲基化。的确,从实际细菌和菌群样本中,文章还发现很多细菌其实存在多个5mC motifs,远超出之前研究的估计,从而创造很多新的研究机遇。
图2:新的方法NanoDisco同时解决了利用纳米孔测序de novo发现DNA甲基化motifs遇到的两个根本问题:1)识别出甲基化类型(methylation typing);2)找到motif中具体被甲基化的碱基(fine mapping)
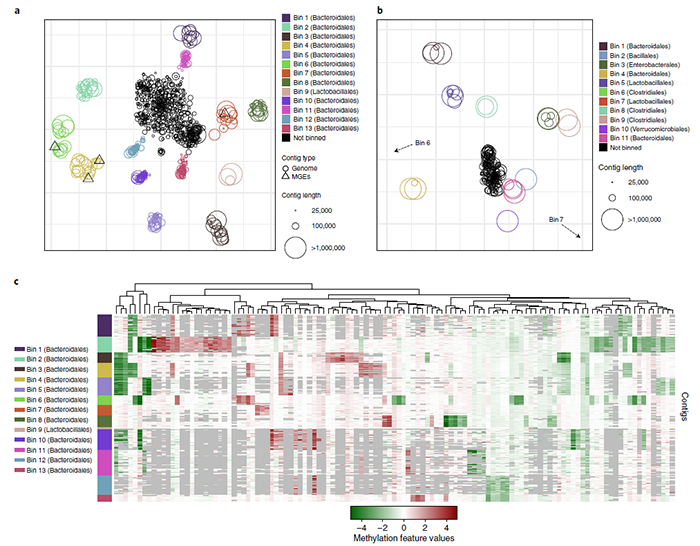
3.利用细菌DNA甲基化多样性增强菌群分析。房刚团队在2017年巧妙利用细菌DNA甲基化多样性作为天然条形码 (natural epigenetic barcode) 来区分基因组高度相似的细菌种和菌株,从而高清晰度的分析菌群。当时的研究是基于PacBio测序平台的,所以只能有效利用细菌中6mA和4mC 甲基化。这项新的研究中开发的新方法有效处理了PacBio测序和纳米孔测序的一些本质区别,从而实现利用三种甲基化 (6mA, 4mC, 5mC) 更好地区分菌群中不同的细菌(图3)、关联可移动原件(Mobile Genetic Elements, 比如质粒)和其宿主、以及检测宏基因组组装中的错误。
图3:同时利用三种细菌DNA甲基化多样性作为天然条形码(natural epigenetic barcode)进行高清晰度的宏基因组分析
文章还对两个常见的三代测序技术进行比较:PacBio的三代测序技术不擅长在多样序列motif中检5mC,但对于细菌6mA和4mC motifs的检测,PacBio测序还是优于纳米孔测序的,即两个平台仍是互补关系;研究者应当根据具体问题选择测序平台。文章还强调NanoDisco侧重于motif层面的细菌甲基化分析而非单个甲基化位点的分析,因为对于后者来说纳米孔测序的精准度在不同motifs之间的高低差别很大。https://github.com/fanglab/nanodiscohttps://nanodisco.readthedocs.io/en/latest/overview.htmlhttps://nanodisco.readthedocs.io/en/latest/faq.html (当中值得一提的是:作者强调该方法专注于细菌和菌群的分析,并不建议其在真核生物中使用)
课题组网页:http://fanglab.bio/
https://doi.org/10.1038/s41592-021-01109-3