在过去的十年里,单细胞测序技术的蓬勃发展深刻地扩展了我们对于基础生物学的理解。目前,单细胞转录组测序在一次实验中就可以检测数万个细胞,在解析细胞异质性和鉴定新型细胞亚群方面具有独特的优势。
虽然说单细胞基因组测序亦可为细胞间异质性和基因组不稳定性等问题提供了新的视角,对于生物学和医学同样非常很重要,不过,当前的单细胞基因组测序主要依赖全基因组扩增的方法,而这种方法的普及目前仍被拷贝数变异检测的低精度、低保真度以及测序成本所阻碍。
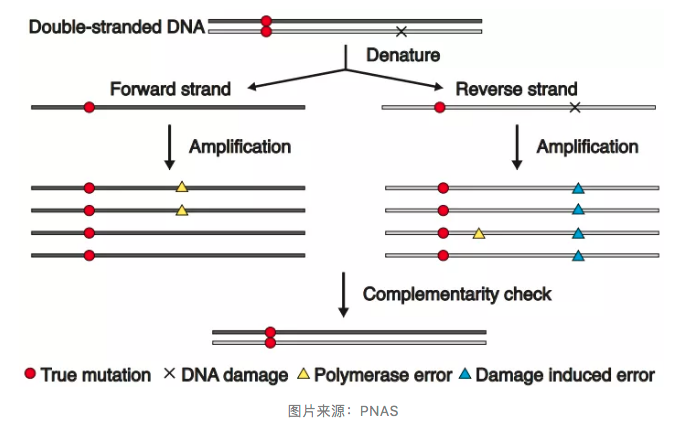
单细胞中单核苷酸变异 (SNVs) 的测定一直存在假阳性的难题,这种假阳性主要由两方面原因造成:首先,用于扩增的聚合酶会产生错误。体外 DNA 合成时,对于大多数 DNA 聚合酶来说,碱基替换的错误率为 10-4 到 10−6。这表明人类 60 亿碱基对的基因组在第一个扩增周期就可以产生数千假阳性。第二, 细胞裂解和扩增过程中外力因素造成的 DNA 损伤,以及活细胞内自然发生的损伤,两者均可被 DNA 聚合酶错误识别并造成假阳性。
为了解决上述难题,北京大学生物医学前沿创新中心主任谢晓亮团队等在 PNAS 在线发表了题为 Accurate SNV detection in single cells by transposon-based whole-genome amplification of complementary strands 的文章。
该研究报道了一种全基因组扩增方法 ——Multiplexed end-tagging amplification of complementary strands (META-CS),该方法能够在单管反应中分别标记和放大 DNA 序列,并准确地从单个细胞中识别出 SNV,几乎消除了所有假阳性,达到了迄今为止最高的精度。

研究内容
META-CS 的原理及主要操作步骤
META-CS 是基于该团队之前开发的全基因组扩增方法 —— Multiplexed end-tagging amplification (META),虽然 META 这种基因组线性扩增的方法在检测拷贝数目变异和单核苷酸变异上的准确度都优于以前的 MALBAC,DOP-PCR 等方法,但仍有改善空间。
考虑到一个真正的 SNV 必须在 DNA 两条链的相同位置,而聚合酶错误和 DNA 损伤都只发生在 DNA 的一条链上,因此,可以通过测序后检查两条链的互补性来过滤掉假阳性。正是基于该理念,研究人员对 META 进行了优化并开发了 META-CS。
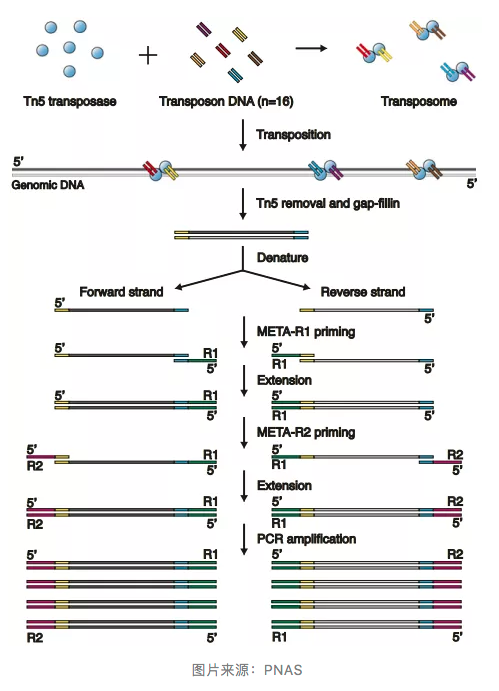
简单地说,META-CS 首先利用 Tn5 转座子对单细胞基因组 DNA 进行片段化,然后每个片段用 16 个独特的转座子序列中的 2 个随机标记,以作为后续反应的启动位点。这种多重转座子序列的使用提供了片段特异性条码,并减少了与分子内发夹结构相关的扩增损失。
接下来,DNA 片段被打开,然后用两个连续的聚合酶延伸反应预扩增两条单链,以获得链特异性标记。每一轮聚合酶延伸反应后,用外切酶去除多余的引物。
最后,从原始 DNA 的正义链和反义链分别扩增出来的产物,可以通过将它们比对到参考基因组的位置进行区分。
这样,通过两条链的信息以确定 SNV,精准度显著提高。同时,META-CS 的整个过程是在一个试管中完成的,并且可以很容易地扩大到多孔板,操作方便,简单易行。
META-CS 准确性的验证
为评价 META-CS 检测 SNV 的准确性,研究人员将一个人类单倍体细胞系 (eHAP) 的单个祖先细胞克隆扩展 5 天至数百个细胞,然后用口吸法获得 10 个单细胞,用 META-CS 的方法进行扩增并测序。其余的细胞被用于另一个 8 天的第二次扩增,然后再提取细胞的 DNA 信息,以代表祖先细胞的基因组,用于比对分析。
首先,他们确定了鉴别一个 SNV 所需的最低测序 reads 数为 4,并且 SNV 检测的数量并没有随着测序 reads 的变化而发生较大改变,这表明 META-CS 能够在 4 个测序 reads 的情况下测量突变率,大大节约成本。
随后,在 eHAP 培养第 5 天的单细胞基因组数据中,每个细胞可以检测到 70±25 个 SNVs。在这 95 个被检测到的 SNV 中,有 63 个为 C > A 类型的转换。这与此前类似研究的报道基本一致。
为了验证这种 C > A 主导的突变谱,研究人员对剩下的继续培养的细胞进行扩增和测序。在相同的检测阈值下,他们发现平均每个细胞 231±65 个 SNV,这表明第 13 天的细胞确实比第 5 天积累了更多的突变。然而,第 13 天细胞的突变谱几乎与第 5 天细胞相同 (皮尔逊相关系数 R = 0.995)。后续单个人类精子的基因组数据同样验证了 META-CS 鉴别 SNV 的准确性。
人类神经元中 SNVs 的检测
在确定了 META-CS 的准确性之后,研究人员将其应用于人类神经元的研究上。
长期以来,人们认为体细胞 SNVs 在成熟神经元中积累,并被推测在衰老和神经退化中发挥作用。然而,对单个神经元的研究尚不成熟,为了更准确地描述神经元的突变模式,他们从三个死后个体的脑组织中单个前额叶皮层的神经元进行全基因组扩增测序。
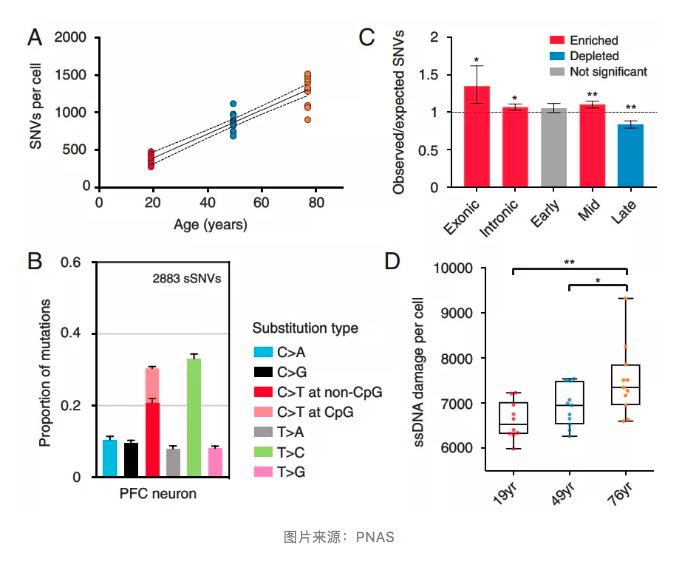
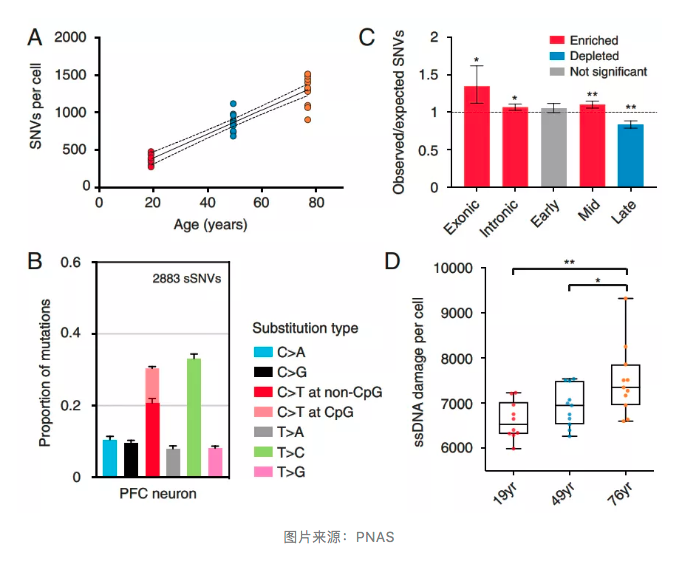
结果发现,来自 19 岁供者的样本可以在每个细胞共鉴定出 379±66 个 SNV,而这一数字在 49 岁供者为每细胞 871±123 个,76 岁供者为每细胞 1304±202 个。很显然,该结果支持了神经元中 SNVs 随着年龄的增长而逐渐累积的前期报道。
进一步的研究发现,神经元的 SNVs 主要为 T > C 和 C > T 转换。此前已有研究表明,人类新型突变主要在复制后期区域富集,这可能是由于 DNA 复制后期单链 DNA 的积累所致。不过,非分裂性的神经元的突变在复制后期区域较少,主要富集在转录区域,这也支持了成熟神经元中的部分 SNVs 由转录相关的 DNA 损伤导致的假设。
人外周血单个核细胞中 SNVs 的检测
当前,随着癌症免疫治疗的迅速发展,对免疫细胞基因组变异的研究也将成为必要。因此,研究人员利用 META-CS,对 53 个来自健康男性供体的外周血单个核细胞进行单细胞全基因组扩增测序。
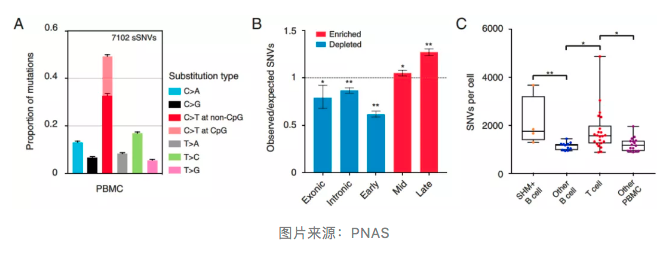
分析结果显示,平均每个细胞鉴定出 1494±721 个 SNVs。外周血单个核细胞的突变谱主要由 C > T 跃迁组成,与造血干细胞谱有很强的相关性。此外,与上述神经元的发现相反, 外周血单个核细胞 SNVs 的基因组在后期复制区域中富集。
此外,B 细胞在成熟过程中发生体细胞高突变 (SHM),这一过程将引入免疫球蛋白基因的高突变率来进行抗体反应。在扩增测序的 15 个 B 细胞中,他们鉴定了 4 个 SHM+ B 细胞,与之前的认知类似,SHM+ B 细胞比其他 B 细胞有更高的 SNV 频率。
更有趣的是,在排除 4 个 SHM+ B 细胞后,其他的 B 细胞和外周血单个核细胞中 SNV 的数量显著低于 T 细胞,其中的内在机制还有待进一步的研究。
研究总结
综上所述,该报道推出了 META-CS 这一单细胞全基因组扩增方法,与传统单细胞全基因组扩增方法相比,META-CS 有四个主要优势:
首先,META-CS 并不局限于具有杂合单核苷酸多态性 (SNPs) 的二倍体细胞,同样适用于单倍体细胞或非整倍体的癌细胞;
其次,该方法非常稳健,单细胞扩增的成功率约为 90%。
第三,整个反应在单管内完成,简化了实验程序,减少了样品损失;
最后,使用 META-CS,只需要 4 条 reads 就可以识别突变,这大大降低了测序成本。
总之,该方法其利用双链 DNA 中两条 DNA 链的互补性来实现准确的 SNV 鉴定,并成功应用于人体多种细胞类型中,这将扩展我们对包括发育、衰老、疾病 (如癌症) 等相关的基础生物学的理解。
通讯作者简介
谢晓亮,生物物理化学家,传奇般的「四院院士」(美国国家科学院院士、美国国家医学院院士、美国艺术与科学院院士、中国科学院外籍院士)。
2010 年主持创办北京大学生物动态光学成像中心「BIOPIC」(现已更名为生物医学前沿创新中心),担任主任;2016 年组建北京大学未来基因诊断高精尖创新中心,担任主任。2018 年 7 月,他全职回国,担任北京大学李兆基讲席教授。2019 年 11 月,他担任北京大学理学部主任。他多次获得国际大奖,包括「阿尔伯尼生物医学奖」,美国化学会「 Peter Debye 物理化学奖」,美国生物物理学会「 Founders 奖」等。(作者介绍来自北大官网)
参考文献
文章来源:丁香学术













