2021年2月17日,来自加州大学尔湾分校(UC Irvine)的聂青教授和Maksim Plikus教授在Nature Communications杂志上发表了题为Inference and analysis of cell-cell communication using CellChat的研究论文(第一作者为金锁钦博士)。基于单细胞数据,研究人员结合系统生物学建模和机器学习的方法,开发了一个高效且方便使用的细胞通讯网络推断和分析的计算工具CellChat,以实现对复杂细胞通讯网络的精确推断、系统定量与比较分析、直观可视化。该研究也创建了一个高质量的配体与受体相互作用的数据库CellChatDB,开发了一个在线交互平台(http://www.cellchat.org/),用于方便获取CellChatDB和探索细胞通讯图谱 (Cell-Cell Communication Atlas)。
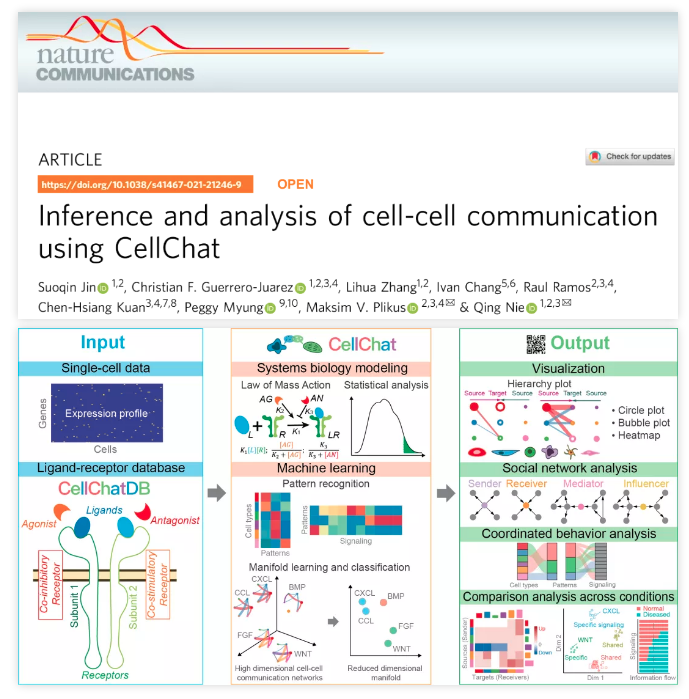
准确地表示配体与受体等信号分子的相互作用对于预测具有生物意义的细胞通讯很重要。研究人员创建了一个经文献验证的配体与受体相互作用的数据库CellChatDB。与其他数据库相比,CellChatDB不仅考虑了具有多个亚基的配体和受体形成的异聚复合物,也考虑了其他协同因子(例如促进剂、拮抗剂)的调控作用。前面这一点对于许多生长因子、细胞因子等(例如BMP、TGF、WNT、Interleukin、Interferon)来说尤其重要,因为配体-受体的相互作用通常涉及多个亚基的结合,亲和力由亚基的特定组合决定【3】。后面这一点对于一些信号通路(例如BMP、TGF、WNT)很重要,因为协同因子可以正、负调控配体与受体的亲和力【4】。
利用系统生物学建模方法,研究人员定量地描述不同细胞类型之间的通讯概率,并结合统计分析进一步推断具有生物意义的显著相关的细胞通讯,从而系统地构建不同细胞类型之间的通讯网络。为了能够直观地展示细胞间的自分泌和旁分泌信号,研究人员提出了一个新的细胞通讯网络的可视化方法,称为分层图。CellChat具有强大的可视化功能,根据不同的分析目的,提供了不同的可视化方法,例如圆形图、气泡图、热图、和弦图等。
为了解析复杂的细胞通讯网络,研究人员灵活运用网络生物学、模式识别和流形学习的建模方法,为揭示关键的信号通路以及细胞通讯的组织原理和生物模式提供了一套数学框架和高效的分析方法。具体地,CellChat可以快速而有效地进行以下计算分析:(1)对于给定的一个细胞通讯网络,推断出不同细胞类型在细胞通讯中扮演的主要作用,例如哪些细胞类型是主要发送和接收信号的,哪些是主要的信号介导子以及具有驱动影响作用;(2)推断不同细胞类型、不同信号通路及其之间的协同作用,从而有助于揭示细胞通讯的组织原理;(3)对推断的细胞通讯网络从功能和拓扑两个方面进行分类,便于根据具有已知生物学功能的信号通路来推断与之相似的信号通路的生物功能;(4)对不同时刻、不同生物条件等多个数据集进行细胞通讯网络的比较分析,揭示细胞通讯的动态变化、共有和特异的信号通路等。针对这些不同的定量分析,CellChat提供了各种各样的直观可视化方法,使得用户可以快速方便地获得关键的分析结果和结论。
研究人员将CellChat工具成功应用于小鼠和人的皮肤发育、伤口愈合和皮肤疾病等单细胞转录组数据集,不仅揭示了很多已经被生物学实验验证的信号通路,同时也揭示了未被文献报道的细胞通讯调控机理。例如,CellChat的计算分析预测了新生小鼠皮肤毛囊早期发育中调控真皮凝结(dermal condensate)形成的两个信号通路Edn3-Ednrb和Pros1-Axl,进一步Maksim Plikus实验室利用RNAscope原位杂交技术在E14.5天小鼠皮肤的相关细胞中验证了这两个信号通路中相关基因的共表达,揭示了小鼠毛发生长的潜在信号调节机制。
综上,针对单细胞数据的细胞通讯分析,该研究提供了一个高质量的配体与受体相互作用的数据库CellChatDB,开发了一个方便使用、定量分析和可视化功能强大、精确性高的细胞通讯网络推断、分析和可视化的R软件包CellChat, 为细胞通讯的研究提供了新的方法、工具和资源。CellChatDB包含2,021对相互作用关系,其中48%涉及异聚体,这些配体与受体对被划分为229个功能相关的信号通路,可以从不同层次研究细胞通讯。据悉,CellChat工具目前已被不少研究人员应用于自己实验室新产生的单细胞数据,研究细胞通讯的机制,特别是在不同条件下细胞通讯的动态变化。
原文链接:
https://www.nature.com/articles/s41467-021-21246-9
参考文献













