确定染色体结构、核体、染色质状态和基因表达之间的关系是研究核组构(nuclear organization)的首要目标。当前的主要研究手段是基于测序的基因组学方法和显微技术,如Hi-C和SPRITE在绘制基因组内染色体间相互作用方面已卓有成效,最近报道的通过Oligopaint和DNA FISH可以实现对单个细胞中的多个DNA位点完成成像【1-3】。以上这些研究已经表明,染色体组构在单细胞水平上乃至单个细胞内两个等位基因之间都是高度异质的。为了进一步在单细胞水平上了解这些组构原理,我们需要一种集成工具来对细胞中的染色体和核体等结构进行成像。
2021年1月27日,来自美国加州理工学院的蔡龙团队在Nature杂志上发表了一篇题为“Integrated spatial genomics reveals global architecture of single nuclei”的文章,在这项研究中,作者开发了DNA seqFISH+技术,并结合多重免疫荧光和RNA-seqFISH的空间多模式方法,对小鼠胚胎干细胞内的染色体结构、核体、染色质状态和基因表达情况进行分析。

首先,在RNA seqFISH(转录组)和DNA seqFISH+(基因组)荧光通道中,研究人员通过以1Mb分辨率标记了2460个基因座,并在随后的sequential IF(组蛋白修饰和亚核结构)荧光通道中以25kb分辨率在20条染色体的每一条上针对60个连续位点获取额外的1200个基因座的成像信息(见图1),经过不断的优化组合方案以及在共聚焦显微镜上对免疫荧光和DNA-FISH图像的多轮杂交比对,研究人员对单个细胞核的整体架构进行综合分析。
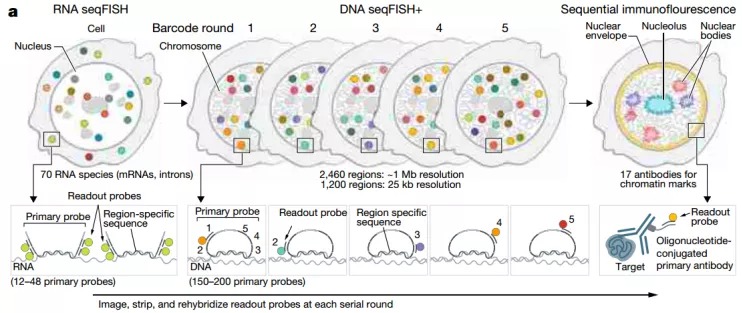
图1. DNA seqFISH+ 结合RNA seqFISH结合以及sequential IF的实验示意图
从整合的多重免疫荧光和DNA seqFISH+数据中,研究人员系统地计算了每个DNA位点和最近的免疫荧光体素(immunofluorescence voxel)的物理距离。此外,通过计算每个DNA位点在免疫荧光标记表面300 nm范围内的时间分数来生成“染色质图谱”。研究人员发现,在单细胞水平上,许多DNA位点在大部分细胞中始终接近特定的免疫荧光标记,例如,在77.2%的细胞中,Pou5f1基因座似乎更接近H3K9ac外部,而在85.2%的细胞中,Eef2更接近核散斑。同一染色体上不同免疫荧光标记的固定位点的存在进一步限制了染色体组构,如染色体4包含与异染色质标记H3K9me3相关的固定位点和核散斑蛋白SF3A66的固定位点。也就是说,尽管单细胞染色体结构和核体位置是可变的,但也同样具备一些不变的特征,提示单个细胞核组织性的存在。
基于这些组合的染色质谱将单独的体素(voxel, 即3D像素)划分为12个主要簇,同时也为每个DNA位点分配一个区(见图2)。其中,一些基因组具有特征性的区域相关性,例如Pou5f1与活性区域2和区域1-2及区域2-3交界相关。研究人员进一步发现,转录活性位点往往出现在RNAPII密集区的表面而非中心,也会出现在活性区与混合区(2-3区)的交界面上。考虑到mRNAs的寿命通常只有几小时,大多数基因可能不会动态地定位到活性区域进行转录,因而可以推断大多数基因很可能预先定位在这些区域或界面上,而这一过程可能由潜在的表观遗传状态或其他因素来决定。
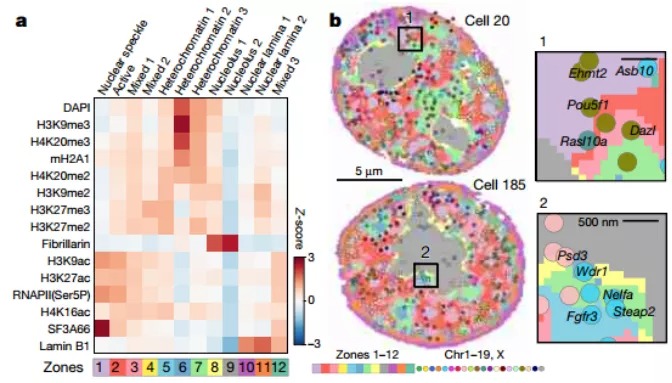
图2. 不同区域染色质标记的差异富集以及z平面对核区及DNA loci的重构
除了上述发现之外,研究人员还观察到细胞核内免疫荧光信号的整体强度在单个细胞之间也显示出明显异质性,免疫荧光数据的聚类分析也显示基于整体染色质修饰水平的至少七种不同状态。那么染色质状态、mRNA表达和染色体组织的异质性在代际间是稳定的还是动态的呢?研究人员通过克隆分析实验加以验证,如果克隆相关细胞具有相似的分子状态,那么这些状态可能在代际之间具有相对的稳定性。为此,研究人员以10:1的比例在GFP阳性小鼠的ES细胞(10)中播种未标记的小鼠ES细胞(1),培养24或48小时(约2-4代)。在24小时的时间点,集落内大多数细胞的总mRNA和染色质特征高度相关,在48小时时间点也保持一定的相关性。需要注意的是,单个免疫荧光标记物,如mH2A1和H3K27me3的动力学在集落内高度相关,但在集落间不相关,提示这些染色质特征至少可在三到四代之间遗传。与此相反,如H3K9ac,无论在一个集落内或集落间均不表现相关性,提示这些特征在代际之间发生快速波动。
以上是这项研究的主要发现。这篇文章的通讯作者,来自加州理工大学的华人科学家蔡龙研究员表示,“我们发现许多DNA位点位于细胞的核体或凝聚物表面,这是非常令人惊讶的,因为它们沿着2D表面排列,而非3D空间的填充”。
蔡龙研究员补充说,“在这篇文章之前,我们认为染色体在单个细胞中是随机排列的。正因为我们可以在同一细胞中同时成像3000个DNA位点和许多抗体,才可以观察到,几乎在所有细胞中,许多位点都以确定的方式与核体相关。举例来说,我们发现Chr4的一部分与异染色质的外表面相关,另一部分与核散斑的外表面相关,而这种关系几乎存在于所有细胞中。那么我们可以说单细胞中的染色体实际上有许多不变的特征,而并非高度随机的结构”。
最后,蔡龙研究员提到,“在物理学中,“invariances”和“equations of motion”之间有着一一对应的关系,因此“invariances”在概念上也一样重要。在生物学中,我们没有方程,因为系统和参数空间是高度多样性的,寻找守恒量是我们能做的最好的事情。在进化中找到守恒量也是非常重要的。同样地,使用单细胞工具,在看似随机的单细胞中发现保守的特征在概念上是有趣的”。
原文链接:
https://doi.org/10.1038/s41586-020-03126-2
参考文献
1. Lieberman-Aiden, E. et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science 326, 289–293 (2009).
2. Quinodoz, S. A. et al. Higher-order inter-chromosomal hubs shape 3D genome organization in the nucleus. Cell 174, 744–757 (2018).
3. Beliveau, B. J. et al. Versatile design and synthesis platform for visualizing genomes with Oligopaint FISH probes. Proc. Natl Acad. Sci. USA 109, 21301–21306 (2012).













