今天跟大家分享的是2020年4月发表在J. Cell. Physiol.(IF:5.546)杂志上的一篇文章”Development and validation of a novel epigenetic signature for predicting prognosis in colon cancer”.在文章中作者建立了一种表观遗传学特征,该特征可用于将结肠癌患者分为两组,其复发风险显著不同。该生物标志物有助于识别需要强化随访和积极治疗干预的患者。
一.研究背景
评估结肠癌患者复发的风险很大程度上取决于肿瘤淋巴结转移(TNM)分期。然而,由于肿瘤的异质性,对于具有相同TNM分期的患者,一贯的治疗策略可能会导致差异的临床结果。研究者一直在寻找敏感的生物标志物来鉴定具有高复发风险的患者。
二.分析流程
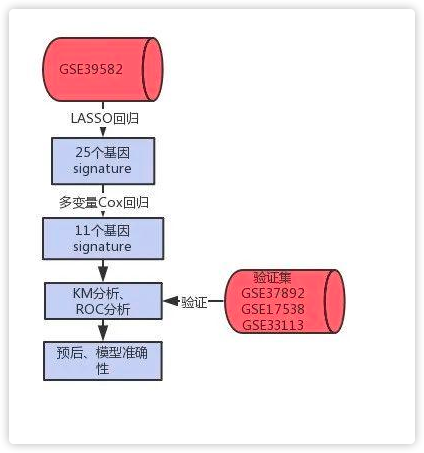
三.结果解读
1.准备GEO数据集
本研究共纳入919例患者,作者将557例来自GSE39582组的患者以1:1的比例随机分为训练集和内部验证集。将GSE37892,GSE17538和GSE33113集合并为验证集,来验证风险评分签名的预后价值。
2.从训练集中发展表观遗传学特征
将来自GSE39582组的患者按1:1随机分为训练集和内部验证集后,作者对调控表观遗传相关基因的表达进行单变量Cox回归分析,并鉴定出70个基因。
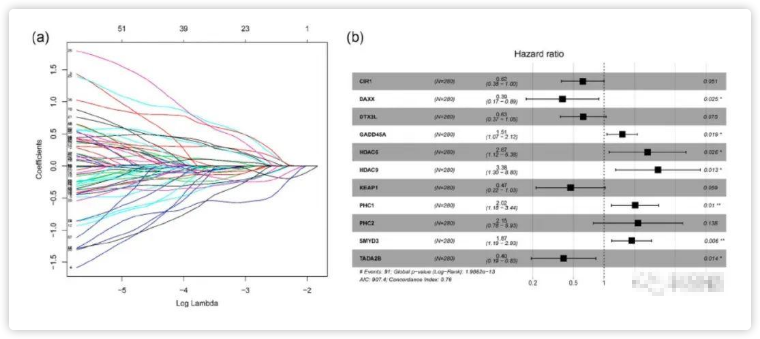
接着进行了LASSO回归分析来进一步缩小范围,并鉴定出25个基因(图 1a)。
然后作多变量Cox回归分析,鉴定出11个基因与结肠癌患者预后显著关联(图1b)。
3.11个mRNA signature在训练集中的预后价值
根据risk score中位数作为临界值,将训练集中的患者分为高风险组和低风险组。
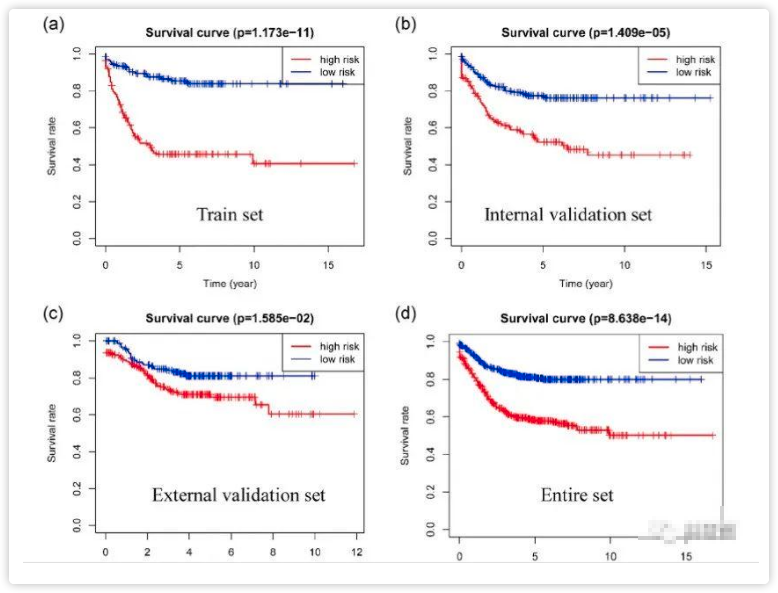
图2a:与低风险组患者相比,高风险组患者的中位RFS显著缩短。
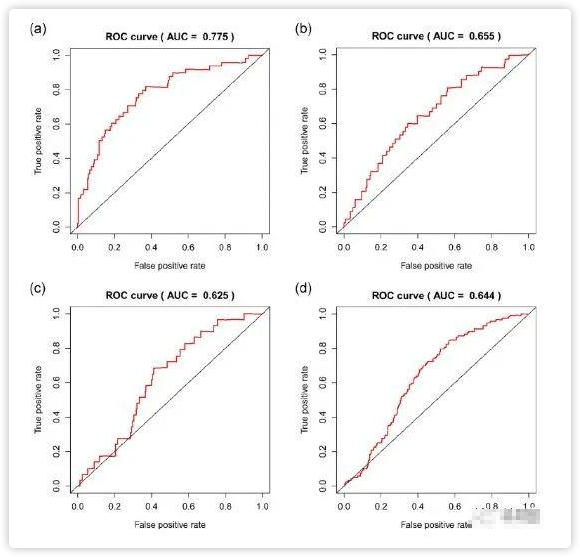
图3a:接着在训练集中进行了ROC分析来评估11个基因signature的预后准确性。
4.在内部、外部验证集和全队列中验证11基因签名的预后预测价值
图2b:在内部验证集中,与低风险组相比,高风险组患者的RFS同样较差。
图2c-d:在外部验证集和全队列中也得到了类似的结果。
接着,同样分别进行ROC分析来验证预后准确性(图 3b-d)。
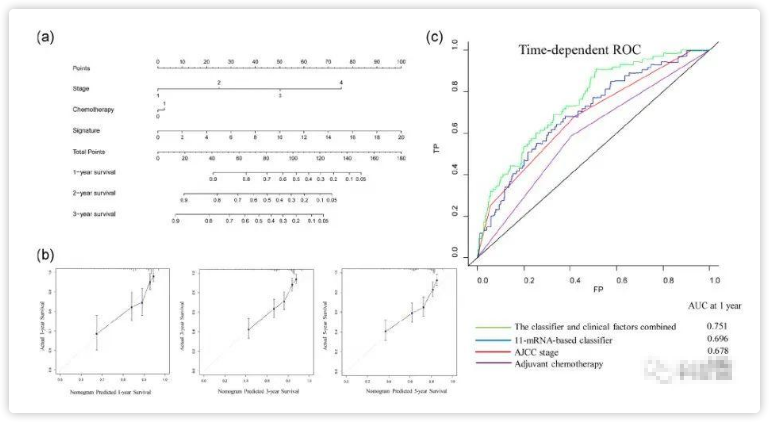
5.11个基因signature的独立预后价值
表1:多变量Cox分析表明,11个基因signature也是独立预后因素。
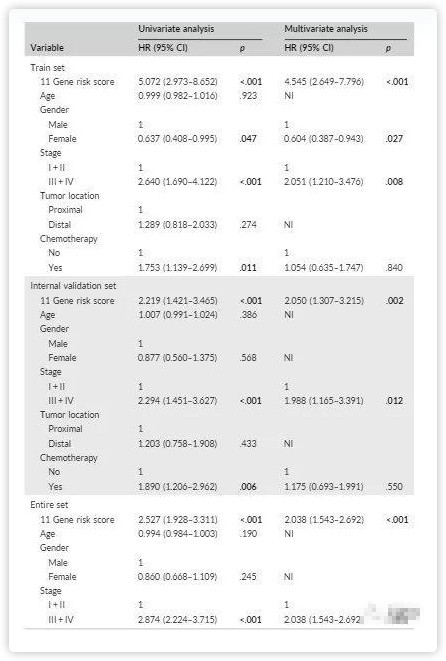
图4:K-M分析显示,在患有II期或III期疾病的患者以及接受或不接受化疗的患者中,高风险组的患者RFS同样较差。
在患有II期或III期疾病的患者以及接受或不接受化疗的患者中,11个基因signature仍是强有力的预后因素。
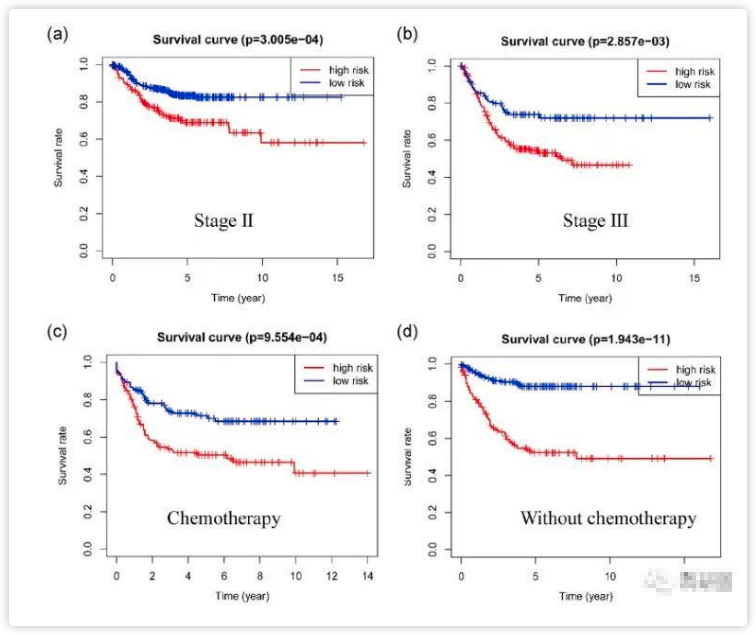
6.开发用于预测结肠癌RFS的列线图
为了预测结肠癌患者的RFS,开发了整合表观遗传调控基因特征,病理分期和辅助化疗的列线图(图 5a-b)。
图5c:1年期的时间依赖性ROC显示,整合后具有更好的预后预测准确性(AUC=0.751)。
小结













