今天和大家分享的是20年2月发表在Aging-US (IF:4.831)杂志上的一篇文章,“Development and validation of a RNA binding protein-associated prognostic model for lung adenocarcinoma”,作者从TCGA数据库获取了肺腺癌(LUAD)的RNA测序数据,并确定了正常组织和癌组织之间差异表达的RNA结合蛋白(RBPs),通过一系列生物信息学分析系统地研究了这些RBPs的表达和预后价值。八个RBPs(IGF2BP1,IFIT1B , PABPC1,TLR8, GAPDH, PIWL4, RNPC3,和lzC3H12C)被鉴定为与预后相关的关键基因,并用于构建预后模型。
一、 研究背景
RNA结合蛋白(RBP)在维持细胞的生理平衡中起着重要作用,尤其是在发育过程和应激反应期。尽管已知RBP参与多种疾病的发生和发展,但RBP在肿瘤发展中的作用仍然很少。许多报道表明RBP在肿瘤中异常表达,影响蛋白质翻译,并参与了癌变。因此,确定肺腺癌(LUAD)中差异表达的RBPs将促进我们对肺癌进展的分子机制的了解,并为诊断和预后提供潜在的生物标志物。
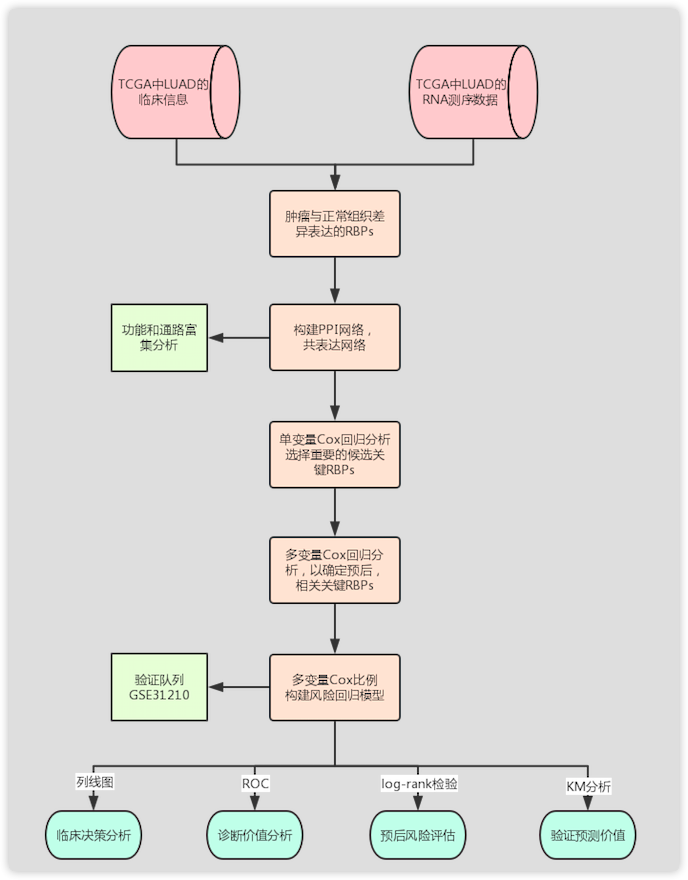
二、 分析流程
三、 结果解读
1.在肺腺癌(LUAD)患者中鉴定差异表达的RBPs
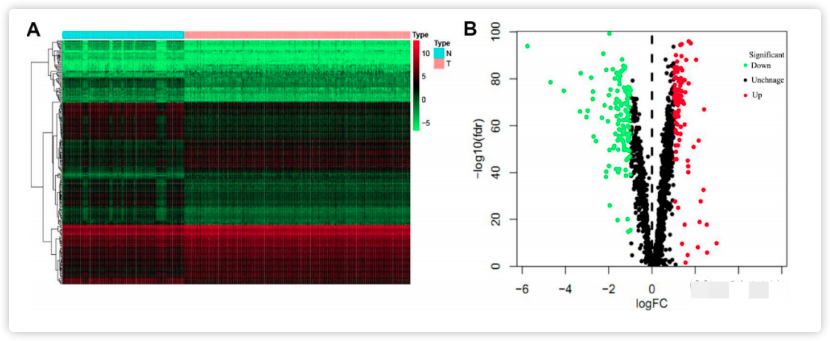
作者对从TCGA数据库中获取了肺腺癌数据库,包含524个肿瘤样本和59个正常肺组织样本。用Limma包分析差异RBPs,分析中总共包括1542个RBP,符合本研究筛选标准有223个RBPs( P <0.05,| log2FC| > 1.0),其中包括101个上调的RBPs和122个下调的RBPs。
图1.肺腺癌中差异表达的RBPs,热图(A);火山图(B)
2.差异表达的RBPs的GO和KEGG通路富集分析
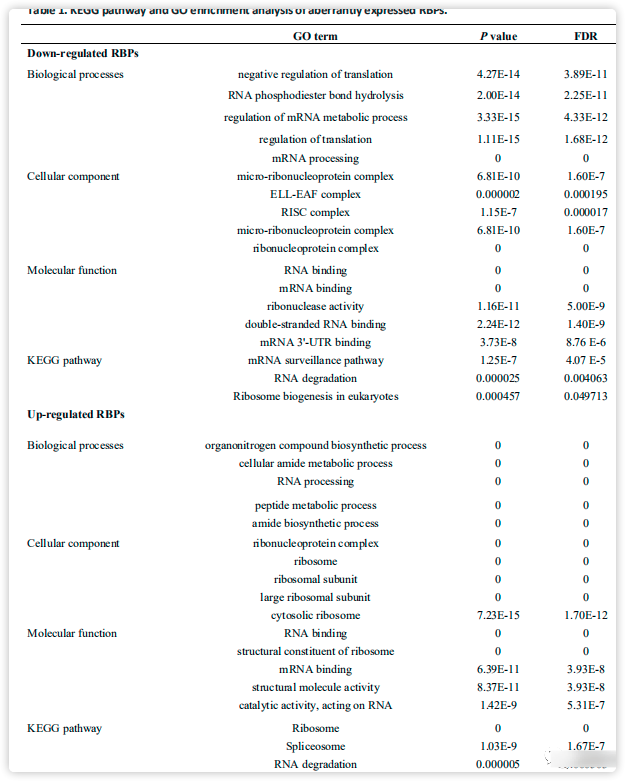
用Webgestalt(http://www.webgestalt.org/)进行GO和KEGG富集分析,下调的差异表达RBPs和上调的差异表达RBPs在生物过程,细胞组成,分子功能中富集情况和参与的KEGG通路如表1所示。作者发现,在真核生物中,下调的差异RBPs主要在mRNA监控途径、RNA降解和核糖体生物发生中富集,而上调的RBPs在核糖体、剪接体和RNA降解中显著富集。
表1.异常表达RBPs的GO和KEGG通路富集分析
3.蛋白质相互作用(PPI)网络构建和关键模块选择
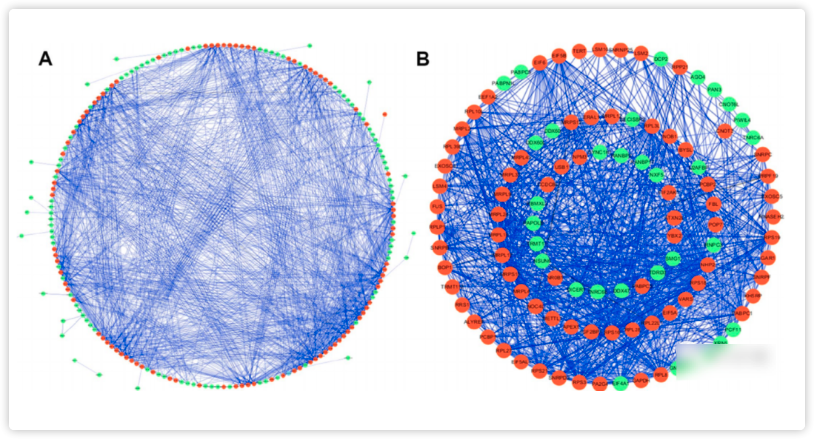
细胞中很多核心分子过程都是通过蛋白质分子构成的复合体完成的,所以作者使用STRING数据库对差异RBPs的蛋白相互作用进行分析,并导入Cytoscape中可视化PPI网络,包括了197个nodes和1484条edges(图2.A); 接着作者使用分子复杂检测(MCODE)插件得到关键模块和基因,并对其进行富集分析,发现了他们存在于很多重要的途径。图2.B是第一个关键模块,共107个节点,用绿色来表示下调的RBPs,红色表示上调的(图2.B)。
图2.蛋白质-蛋白质相互作用网络和模块分析
4.选择与预后相关的RBPs
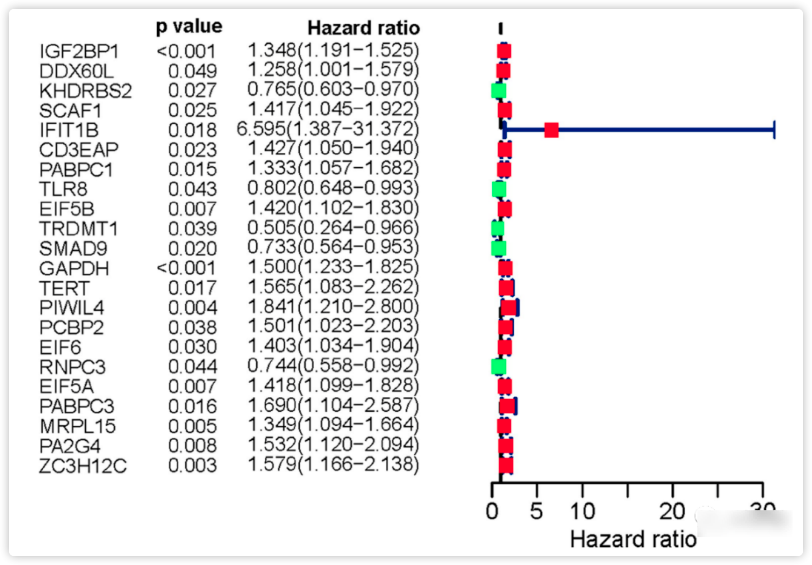
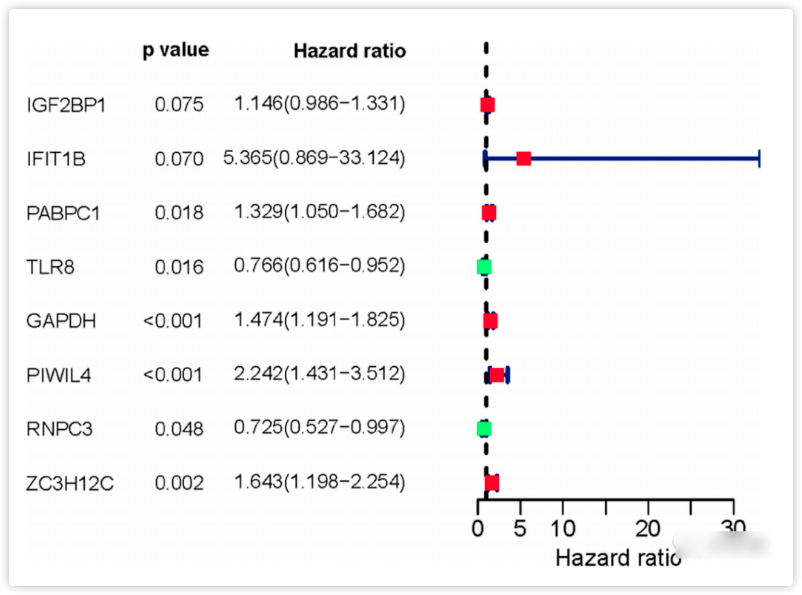
上一步,作者从PPI网络中总共鉴定了197个关键的不同表达的RBP。为了找到对预后影响最大的分子,先进行了单因素Cox回归分析,获得了22个候选RBPs(图3)。接着多因素Cox回归分析了这22个预后相关的候选RBPs,确定了8个关键RBPs是LUAD患者的独立预测因子(图4,表2)。
图3.单因素Cox回归,用于识别训练集中的关键RBPs
图4.多因素Cox回归分析识别与预后相关的关键RBPs
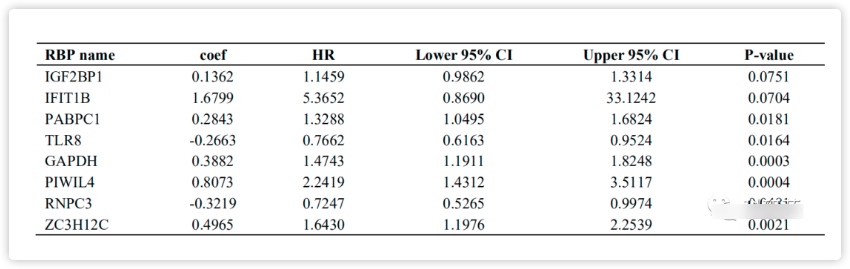
5.风险评分模型的构建与分析
根据上面的表2,计算了每个患者的风险评分:Risk score= (0.1362×Exp IGF2BP1)+(1.6799×Exp IFIT1B)+(0.2843× Exp PABPC1)+(-0.2663×Exp TLR8)+(0.3882×Exp GAPDH1)+(0.8073×Exp PIWIL4)+(-0.3219× Exp RNPC3)+(0.4965×Exp ZC3H12C)
构建完风险模型后,作者进行了生存分析以评估它的预测能力。根据中位风险评分将458名LUAD患者被分为低风险和高风险组。结果表明,高风险亚组患者比低风险亚组的OS差,在KM图下还加了每个时间点的例数(图5.A)。为了进一步评估预后能力,进行了ROC分析,ROC曲线下面积(AUC)为0.775(图5.B),诊断效果中等。图5.C中绘制了风险因子关联图,显示了低风险和高风险得分人群的RBP表达和生存状态,在下图中是按照预测风险值排序的患者与生存时间之间的关系,其中,绿点代表活着的患者,红色代表过世的患者,从图中可以看出,高风险人群的死亡人数明显高于低风险人群(右边的红点更多)。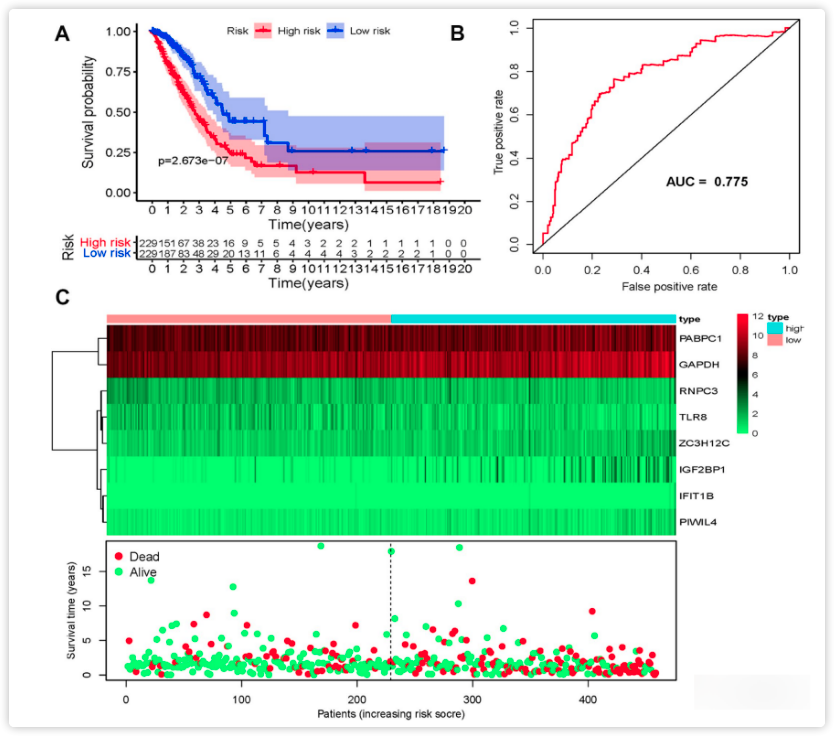
图5.TCGA队列中的八个基因预后模型的风险评分分析。低风险和高风险亚组的生存曲线(A);ROC曲线,用于根据风险评分预测(B);表达热图和生存状态散点图(C)。
此外,作者还评估了在其他LUAD患者队列中具有相似预后价值的八种RBPs预测模型,用GSE31210数据集作为验证队列。在GSE31210队列中,高风险评分的患者的OS也较低风险评分的患者差(图6A-6C),AUC比训练集的结果还要好。这些结果表明该预测模型具有较好的敏感性和特异性。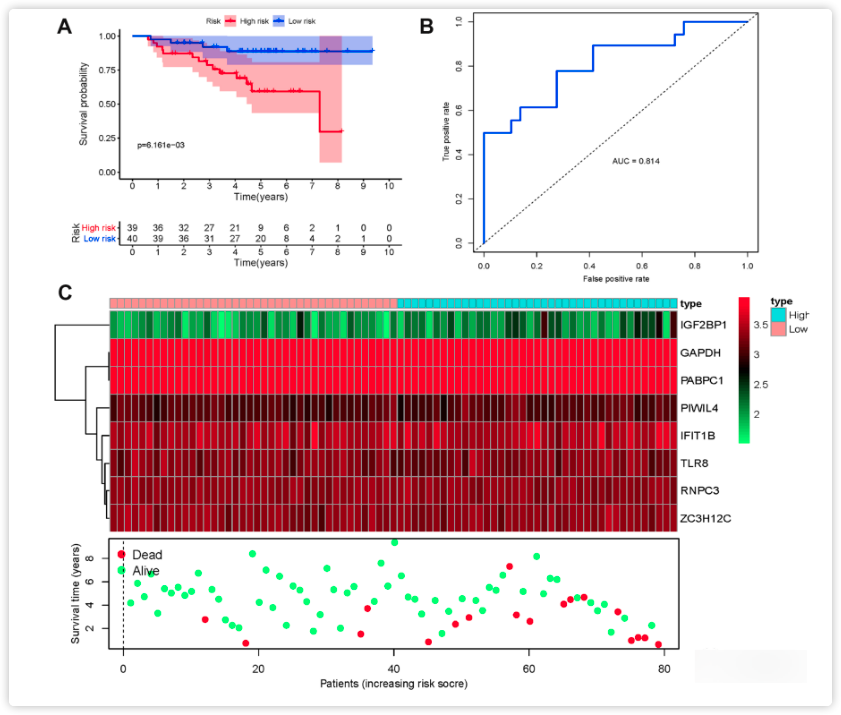
图6.GSE31210队列中的八个基因的预后模型的风险评分分析
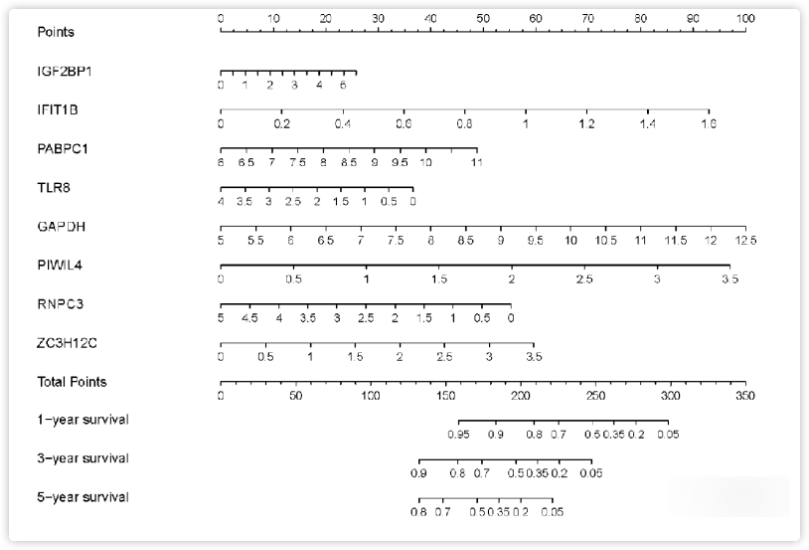
6.基于八个关键RBPs的列线图的构造
基于多因素Cox分析的结果,作者利用列线图Nomogram来对回归可视化,根据每项得分计算LUAD患者的估计生存率。对每个自变量取值,在该点作一条垂直于Points轴的直线,交点即代表该自变量取值下的评分,计算每个患者各个自变量对应的points,加起来就是total points. Total Points轴上找到该患者总分对应的点,画一垂直线到生存概率轴上,交点即为该患者的1年,3年或5年生存概率。
图7. TCGA队列中用于预测LUAD患者的1、3和5年OS的列线图
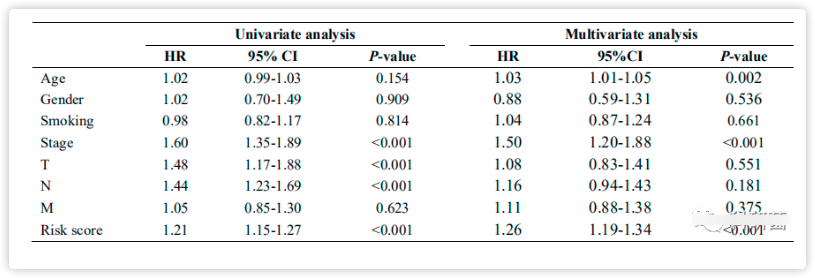
此外,通过进行COX回归分析评估了TCGA中的LUAD患者不同临床特征的预后意义。结果显示,LUAD患者的肿瘤分期,原发肿瘤部位,局部淋巴结受累和危险评分与OS相关(P < 0.001)。但是,通过多元回归分析仅发现年龄,肿瘤分期和风险评分是与OS相关的独立预后因素。
表3.不同临床参数的预后价值
7.验证关键RBPs的预后价值和表达
为了进一步探讨LUAD中8个的关键RBPs预后价值,使用Kaplan Meier生存分析了其中6个RBPs,对数秩检验的结果表明,六个RBPs与LUAD患者的OS相关(图9)。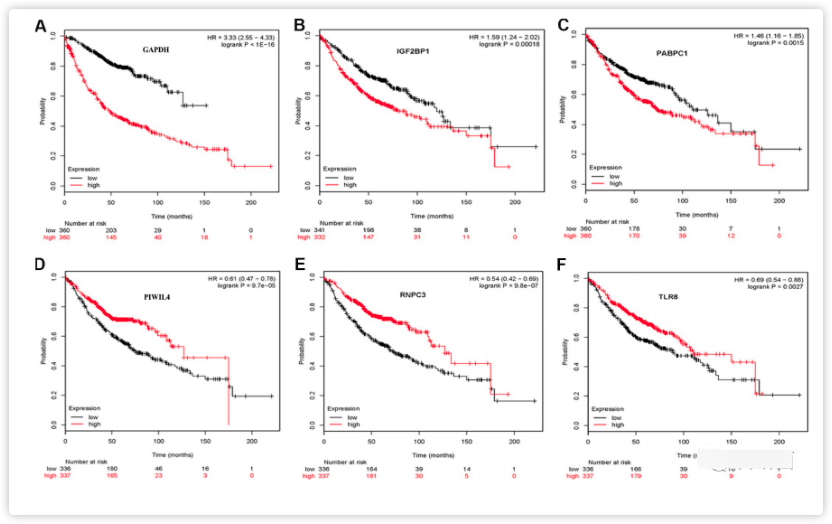
图9.Kaplan Meier验证RBPs在LUAD中的预测价值
为了进一步确定这些关键RBPs在LUAD中的表达,作者使用了人类蛋白质图谱数据库(Human Protein Atlas database http://www.proteinatlas.org/)的免疫组化结果,与正常肺组织相比,肺癌中IGF2BP1,PABPC1和GAPDH显著增加。但是,肺癌组织中TLR8,PIWIL4和ZC3H12C的抗体染色水平相对降低。此外,IFIT1B的蛋白质表达在肿瘤和正常肺组织之间没有显著差异(图10)。这一步提示读者,如果缺乏自己的临床样本,或许也可以在相应数据库中找到免疫组化的结果来验证自己的观点。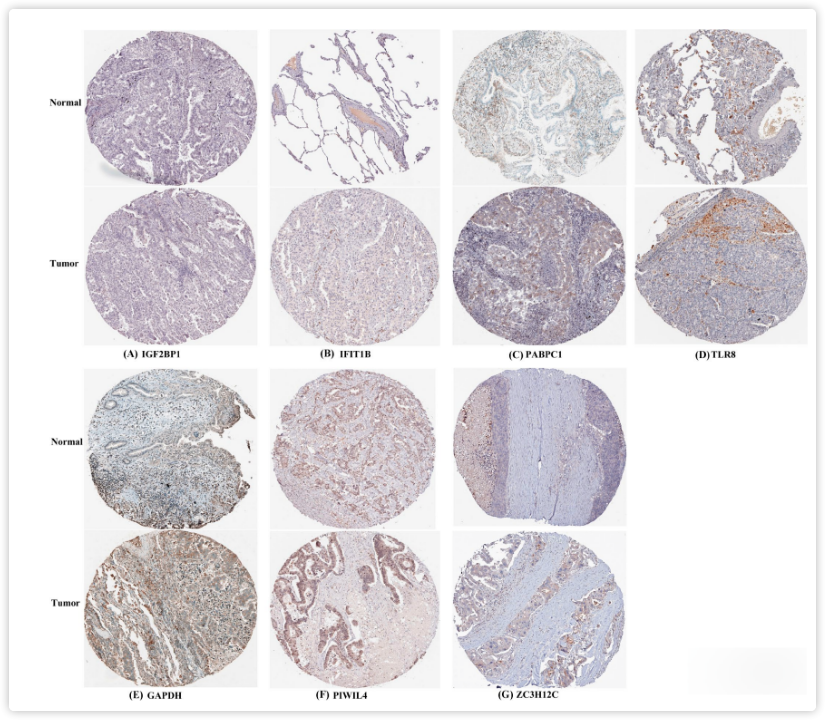
图10.使用HPA数据库验证LUAD和正常肺组织中关键RBPs的表达
小结 本篇文章中根据来自TCGA的LUAD数据,鉴定了223个在肿瘤和正常组织之间表达不同的RBPs。系统地分析了相关的生物学途径,构建了这些RBPs的共表达网络和PPI网络。此外,作者还进行了关键RBP的单变量Cox回归分析,生存分析,多因素Cox回归分析和ROC分析,以进一步探索其生物学功能和临床意义。最后基于八个与预后相关的关键RBPs基因构建了预测LUAD预后的风险模型。这些发现可能有助于开发新颖的生物标志物,用于LUAD患者的诊断和预后。