肺部微生物群的生物失调可能与肺部疾病的发展有关。然而,肺部微生物群和肺癌之间的相互作用仍然不清楚。今天我们来分享一篇文章,看看肺部微生物群在肺肿瘤中可能的意义~
“Characterization of the lung microbiome and exploration of potential bacterial biomarkers for lung cancer”
摘要
肺部微生物群的生物失调可能与肺部疾病的发展有关。然而,肺部微生物群和肺癌之间的相互作用仍然不清楚。该研究的目的是评估和比较肺癌和良性肺部疾病之间在微生物分类和衍生功能方面的差异。对样本α多样性和β多样性、肺微生物群组成的分类状况、肺微生物群功能预测等方面的开展了研究。结果表明,肺癌患者和良性肺部疾病患者的肺部微生物群存在差异,某些细菌可能具有预测肺癌的潜力。更进一步推测肺微生物组可能具有作为细菌生物标志物和肺癌治疗新靶点的潜力,值得进一步探索。
相关概念
一.微生物多样性研究相关知识:
1.多样性指数:是指物种多样性测定。
主要有三个空间尺度:α多样性,β多样性,γ多样性。
每个空间尺度的环境不同测定的数据也不相同。
α多样性: 主要关注局域均匀生境下的物种数目,因此也被称为生境内的多样性(within-habitatdiversity)。
群落生态学中研究微生物多样性,通过单样品的多样性分析(α多样性)可以反映微生物群落的丰度和多样性,包括一系列统计学分析指数估计环境群落的物种丰度和多样性。
β多样性: 指沿环境梯度不同生境群落之间物种组成的的相异性或物种沿环境梯度的更替速率也被称为生境间的多样性(between-habitatdiversity),控制β多样性的主要生态因子有土壤、地貌及干扰等。
β多样性意义:
①它可以指示生境被物种隔离的程度;
②β多样性的测定值可以用来比较不同地段的生境多样性;
③β多样性与α多样性一起构成了总体多样性或一定地段的生物异质性。
群落生态学中研究微生物多样性,β多样性是对不同样品/不同组间样品的微生物群落构成进行比较分析。
γ多样性: 描述区域或大陆尺度的多样性,是指区域或大陆尺度的物种数量,也被称为区域多样性(regionaldiversity)。控制γ多样性的生态过程主要为水热动态,气候和物种形成及演化的历史。
群落生态学中研究微生物多样性,γ多样性分析是指α多样性与β多样性相结合的分析。
2.α多样性指数
计算菌群丰度(Community richness)的指数
Chao指数,chao指数越大,说明该样本物种数比较多。
计算菌群多样性(Communitydiversity)的指数
Shannon指数,Shannon值越大,说明群落多样性越高。
二.principal coordinate analysis (PCoA):
主坐标分析,是一种研究数据相似性或差异性的可视化方法,通过一系列的特征值和特征向量进行排序后,选择主要排在前几位的特征值,PCoA 可以找到距离矩阵中最主要的坐标,结果是数据矩阵的一个旋转,它没有改变样品点之间的相互位置关系,只是改变了坐标系统。通过PCoA 可以观察个体或群体间的差异。
可以通过分析坐标轴中样本和样本之间的距离直观地看到2个样本或2组样本之间的菌群差异性。若2个样本或2组样本之间的直线距离较近,则表示这2个样本或2组样本的菌群差异性较小;相反,若2个样本或2组样本之间的直线距离较远,则表示它们之间菌群差异性较大。
三.KEGG 通路富集分析
KEGG PATHWAY数据库是一个手工画的代谢通路的集合。基因通路富集分析就是在一组基因或蛋白中找到一类过表达的基因或蛋白。通过基因通路富集分析,可以初步分析基因可能参与的生物学过程或者信号通路。
四.GINI(基尼系数)
Gini系数是国际上通用的,用以衡量一个国家或地区居民收入差距的常用指标。所以,运用在模型中是评价模型的区分能力的指标。
Gini<0.3,模型不太能接受;
0.3<=Gini<0.35,模型区分能力一般,模型有优化空间;
0.35<=Gini<=0.5,模型区分能力比较满意;
Gini>0.5,Gini越高越有过拟的可能性。
五.随机森林模型
随机森林是机器学习中的一种常用方法,随机森林顾名思义,是用随机的方式建立一个森林,森林里面有很多的决策树组成,随机森林的每一棵决策树之间是没有关联的。在得到森林之后,当有一个新的输入样本进入的时候,就让森林中的每一棵决策树分别进行一下判断,看看这个样本应该属于哪一类(对于分类算法),然后看看哪一类被选择最多,就预测这个样本为那一类。
背景
在过去,人们通常认为肺是一个无菌的环境。然而,高通量测序技术发现肺存在许多细菌群落,包括变形菌、厚壁菌门和拟杆菌门。越来越多的证据表明,肺部微生物群的改变与肺部疾病之间存在联系,如哮喘、慢性阻塞性肺病、特发性肺纤维化(IPF)、囊性纤维化和肺癌。
已知一些特定的微生物与癌症有关。例如,幽门螺杆菌与胃癌的高风险有关;核梭杆菌的数量与结直肠癌患者的预后有关。而微生物组和肺癌之间的联系仍然相对不清楚。尽管以前的一些研究已经注意到支气管肺泡灌洗液、肺组织、痰、唾液和粪便样本中特定微生物群的变化可能与肺癌有关,但这些研究产生了一些相互矛盾的结果。此外,有研究发现,乳杆菌在治疗小鼠肺癌方面显示出效果,这意味着调节微生物可能影响肺癌的治疗。因此,文章进一步探索微生物群与肺癌之间的关系,并且探究其相互作用的潜在机制。
以前的一项研究表明,个体的微生物群可以保持长期稳定,这突出了微生物群作为诊断方法和治疗靶点的潜力。由于唾液和痰液样本容易受到口腔微生物群的干扰,并且通常很难从晚期肺癌患者身上获得肺组织样本,因此一些专家认为,BALF是一种可行的肺部微生物群取样方法,因为BALF微生物群不会受到口腔污染的干扰。为了验证以前的研究,并提供关于肺微生物群如何影响肺癌患者和良性肺部疾病个体的更具体的理解,该研究旨在进一步应用基于16S rRNA的二代测序来研究BALF样本中肺微生物群的分类和衍生功能特征的差异。
方法
1.研究共招募了54名患者,其中32名患者病理诊断为肺癌,22名患者诊断为良性肺部疾病作为对照组。记录所有参与者的人口统计学和临床数据,包括年龄、性别、体重指数(BMI)、吸烟史、吸烟指数、肿瘤标志物、病理类型和肿瘤分期。
2.用十六烷基三甲基溴化铵/十二烷基硫酸钠法从BALF样品中提取基因组DNA。
3.通过16S rRNA扩增子测序进一步分析。
4.α多样性的计算方法是使用ASVs, Chao1, Shannon指数进行测量。β多样性是使用布雷-柯蒂斯度量、未加权均匀性和加权均匀性对稀疏数据进行测量的。应用非参数多元方差分析评估组间的多样性差异。基于布雷-柯蒂斯度量、未加权均匀分布和加权均匀分布,进行主坐标分析。在根据ASVs数据预测KO丰度,并根据LEfSe 的LDA评分估计差异富集的KEGG途径。使用随机森林模型来估计每个不同属的重要性,并根据ROC特征曲线中的AUC值来评估预测肺癌的能力。
5.Wilcoxon秩和检验用于对组间分类丰度数据(出现率> 10%,平均丰度> 0.5%)进行假设检验,以获得基于不同级别分类丰度表的P值,并根据需要通过BenjaminiHochberg校正对P值进行调整,以进行多次检验。微生物数据和临床参数之间的关系用斯皮尔曼相关试验进行评估。
结果
临床样本基线特征
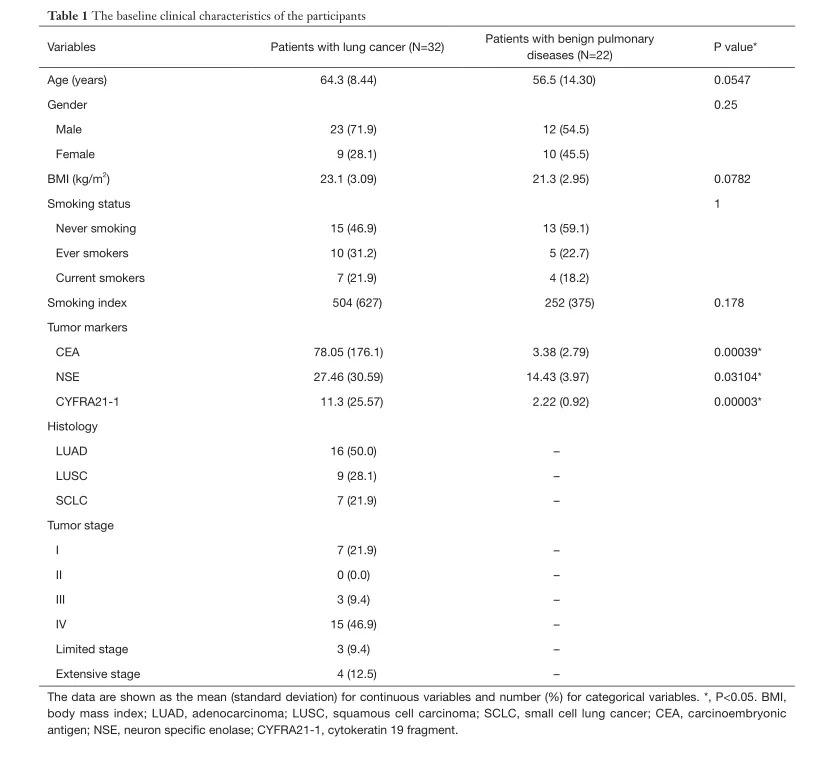
表1 显示了54名参与者的人口统计学和临床数据,包括32名肺癌患者和22名良性肺部疾病患者。
从表中可以发现:两组在年龄、性别、BMI、吸烟状况和吸烟指数方面没有显著差异。而在肺肿瘤标志物(CEA,NSE,CYFRA21-1)方面存在显著差异。患者临床特征(包括年龄、性别、体重指数和吸烟)的差异可能会影响单个细菌群落,这两组患者在这些因素上没有统计学上的显著差异,故而最大限度地避免了对最终微生物群测序的干扰。
肺癌和对照组之间的α和β多样性
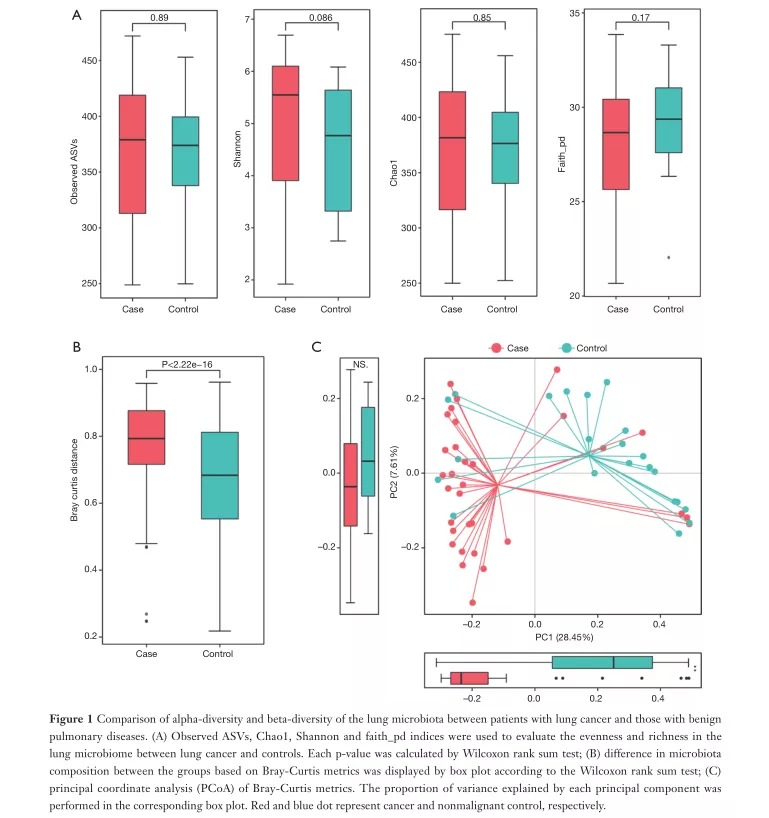
Fig1.探究了肺癌患者和良性肺部疾病患者肺微生物群α多样性和β多样性的差异。
A.通过ASV、Chao1、Shannon和faith_pd指数来评估肺癌组和对照组之间的肺微生物组的均匀度和丰富度(α多样性)。从图中可以发现:四个P值均大于0.05,故肺癌患者与良性肺部患者的肺部微生物组的丰富度和平均度是相似的。
B.根据威尔科克森秩和检验,使用布雷-柯蒂斯度量组间微生物群落组成的差异(β多样性)。发现:P<0.05,故两组的β多样性差异显著。
C.根据布雷-柯蒂斯度量进行了主坐标分析。两组样本之间的直线距离较远,表示肺癌患者和良性肺部疾病患者之间菌群差异性较大。
肺微生物群组成的分类状况
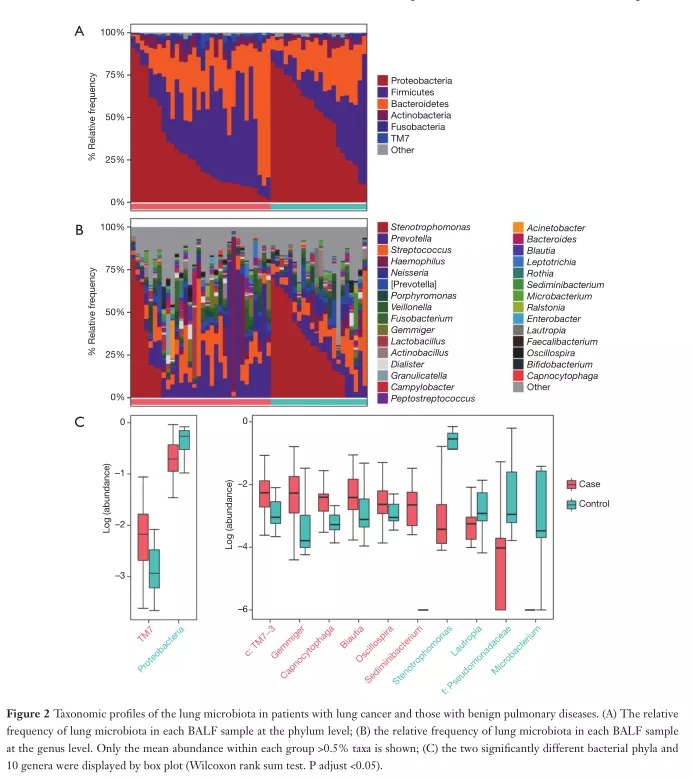
Fig2.探究了肺癌患者和良性肺部疾病患者肺微生物群的分类状况差异。根据两组BALF样品中微生物群落的相对丰度,进行了门和属水平的分类和分析。
A.展示了每个BALF样本中肺微生物群在门水平上的相对丰度。可以发现:变形菌门、厚壁菌门和拟杆菌门在癌症组和对照组中都是最常见的。而且在两组中放线菌、梭杆菌和TM7的平均丰度均大于0.5%。
B.展示了每个BALF样本中微生物群落在属水平的相对丰度。可以发现:嗜食单胞菌、普雷沃氏菌、链球菌、嗜血杆菌和奈瑟氏球菌在癌症组和对照组中都是最常见的。
C.两个明显不同的细菌门和10个属用箱线图来显示。
与良性肺部疾病相比,肺癌患者TM7的相对丰度显著增加(调整P=0.005634),蛋白细菌显著减少(调整P=0.01388)。
肺癌患者中有6个属(c:TM7-3、囊胞吞菌属、沉降杆菌属、革兰氏阴性菌属、布拉氏菌属和示波螺旋菌属)比良性肺部疾病患者更丰富(校正P<0.05),有四个属(微杆菌属、狭营养单胞菌属、劳托亚属和假单胞菌科)丰度低于良性患者。
预测肺微生物群的功能
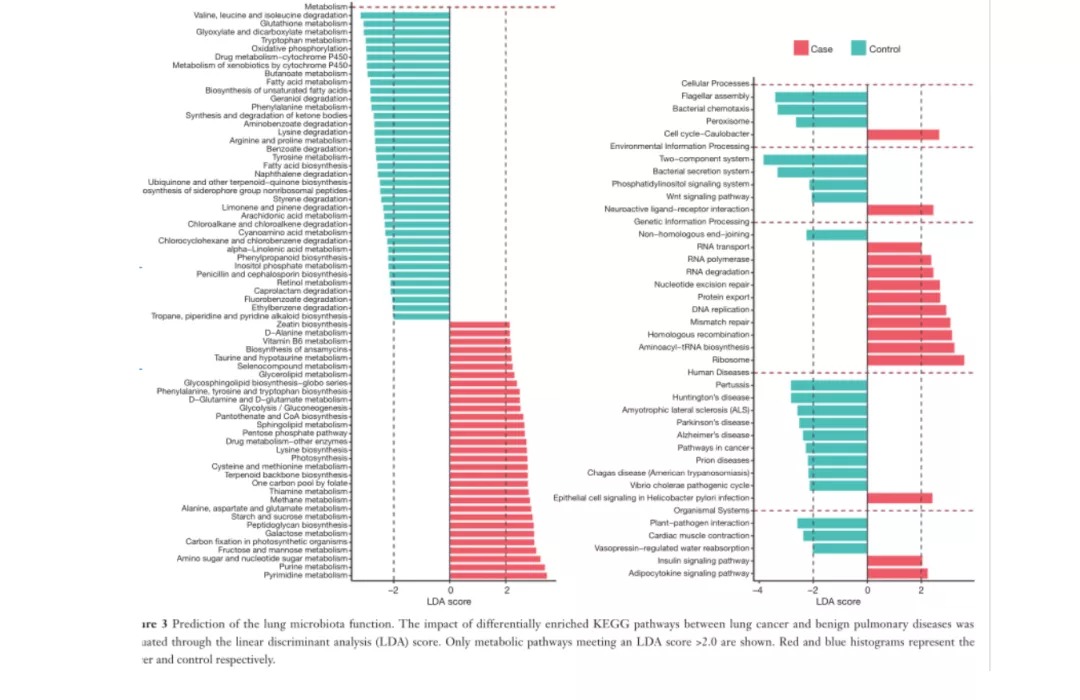 Fig3.预测了肺微生物群功能。通过线性判别分析(LDA)评分评估肺癌和良性肺部疾病之间差异富集的KEGG通路的影响。仅显示符合LDA评分> 2.0的代谢途径。
Fig3.预测了肺微生物群功能。通过线性判别分析(LDA)评分评估肺癌和良性肺部疾病之间差异富集的KEGG通路的影响。仅显示符合LDA评分> 2.0的代谢途径。
从图中可发现:在肺癌组和对照组中分别有46和57条差异途径。肺癌患者BALF样本中的微生物群有明显的核糖体代谢、嘧啶代谢和嘌呤代谢途径的代谢行为 。良性肺部疾病患者的样本中,有双组分系统、鞭毛装配和细菌分泌系统明显过多。
肺癌患者潜在的细菌生物标志物
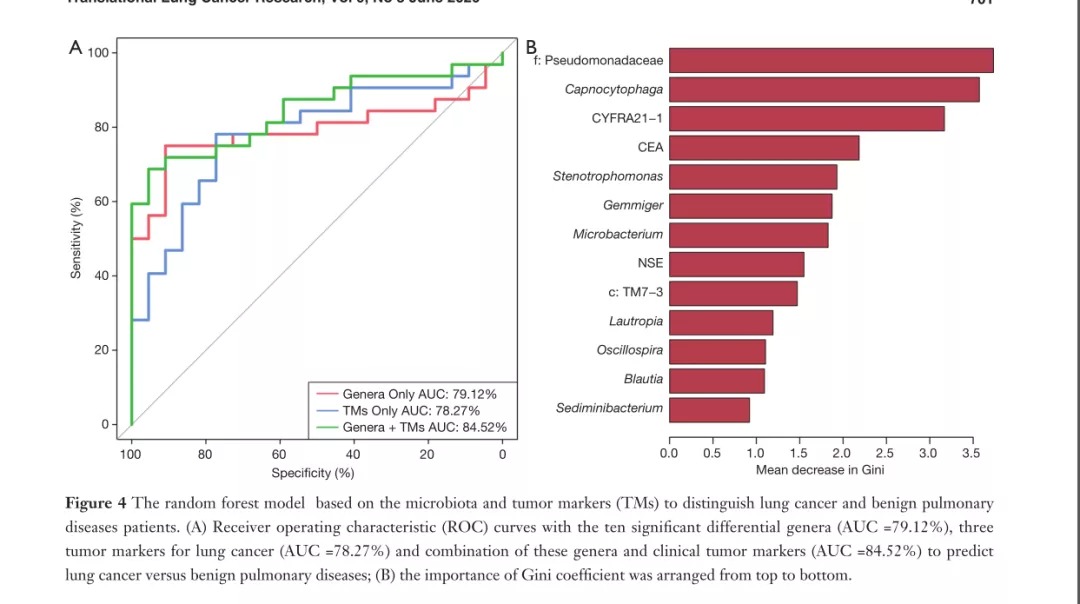 Fig4.基于微生物群和肿瘤标志物(TMs)的随机森林模型来区分肺癌和良性肺部疾病患者。
Fig4.基于微生物群和肿瘤标志物(TMs)的随机森林模型来区分肺癌和良性肺部疾病患者。
A图:对比了三个组:Genera组:十个具有显著差异属(AUC =79.12%)、TMs组:三个肺癌肿瘤标志物(AUC =78.27%)、Genera+TMs组:十个显著差异属和临床肿瘤标志物的组合(AUC =84.52%)的检测良恶性肺部疾病的效能,通过ROC曲线可以发现:Genera+TMs组:十个显著差异属和临床肿瘤标志物的组合的AUC =84.52%最高,表示其检测效果最好。
B图:展示了各个标志物的Gini系数。从图中可发现:与临床肿瘤标记物相比,假单胞菌科和囊胞吞菌属的Gini系数较高,且区分能力比较满意。
接着,作者还对微生物标志物(嗜囊细胞性细菌、沉降性细菌和c:TM7-3)和肿瘤标志物(CEA和CYFRA21-1)进行了相关性分析,分析发现:二者呈正相关且显著相关。
讨论
结论:
1.肺癌患者和良性肺部疾病患者之间菌群差异性较大,在微生物群落的整体结构中观察到分化的趋势。
2.肺癌患者和非恶性肺部疾病患者的样本中微生物群落的丰富度和多样性没有显著差异。
3.肺癌患者的样本中,TM7和c:TM7-3的相对丰度显著升高。
4.肺癌患者的核糖体和嘧啶代谢途径明显丰富。
5.细菌和临床肿瘤标志物的组合可以更好地预测肺癌。
局限:
1.本研究登记的患者数量不够大,肺癌患者没有按组织学亚型或不同阶段进行分类,可能存在异质性。
2.在没有验证队列的情况下,使用特定细菌来区分肺癌和良性肺部疾病可能会导致假阳性值和不可靠性。
3.本研究是横断面研究,未对微生物组之间相互作用的机制做进一步探讨。
参考文献:Cheng C, Wang Z, Wang J, Ding C, Sun C, Liu P, Xu X, Liu Y, Chen B, Gu B. Characterization of the lung microbiome and exploration of potential bacterial biomarkers for lung cancer. Transl Lung Cancer Res. 2020 Jun;9(3):693-704. doi: 10.21037/tlcr-19-590. PMID: 32676331; PMCID: PMC7354118.













