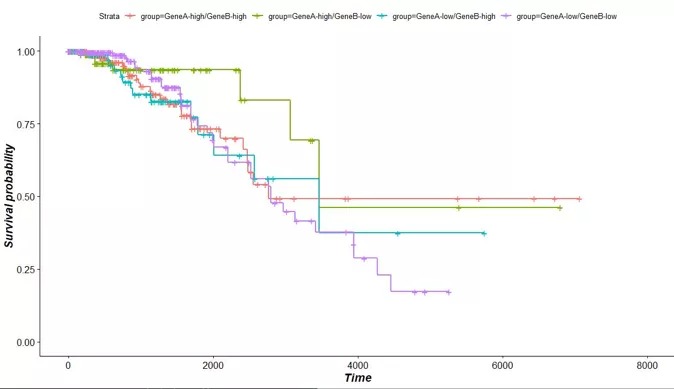
RTCGA是一系列根据数据类型分离的包,相当于要先下载这些离线数据R包之后再直接从离线数据包里面获取TCGA的所有数据。最新的版本可以加载下图所有的包,可谓是非常强大了。

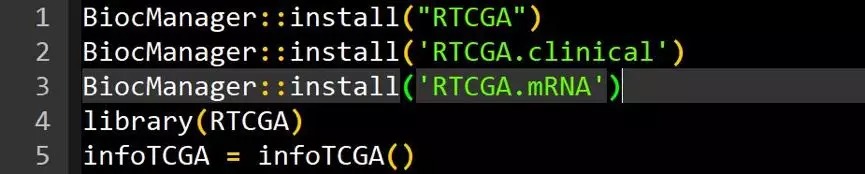
infoTCGA看到各种肿瘤的各种数据那是相当的丰富。
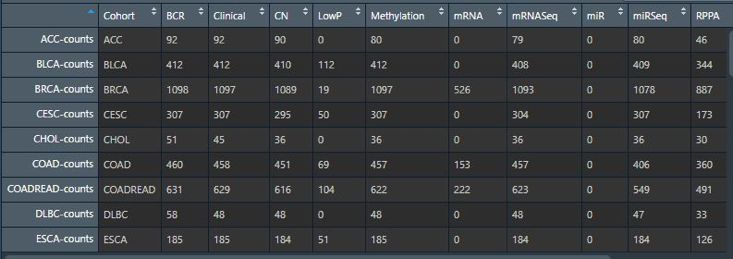
二、从TCGA数据库上获取乳腺癌 的临床数据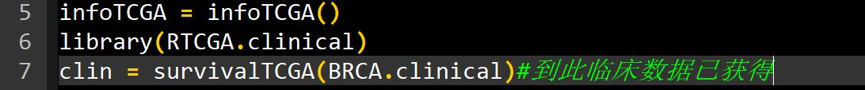
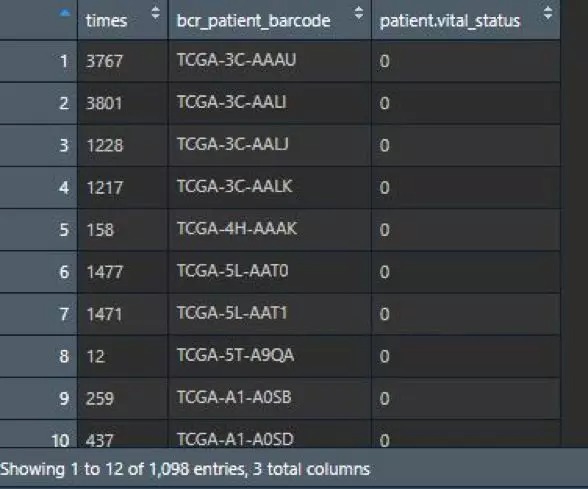
至此便获得了乳腺癌的临床数据。从图可以看出该数据集一共有1098个样本。
三、从TCGA数据库上获取乳腺癌 相关基因的表达数据并整合
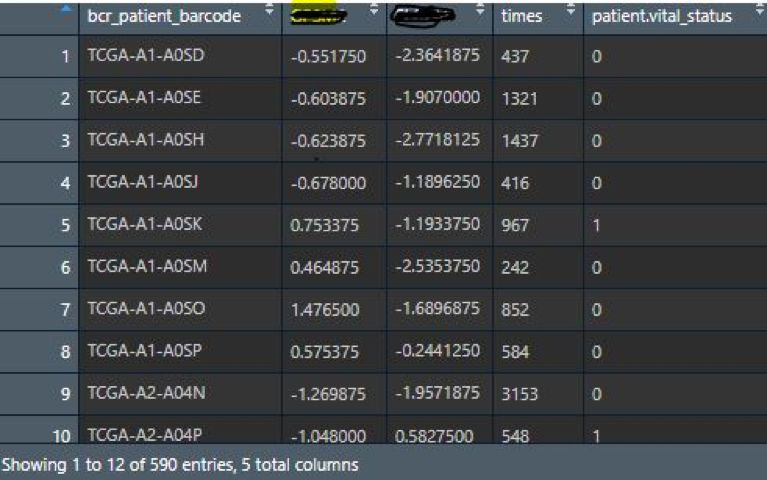
此时得到的exprSet如图:

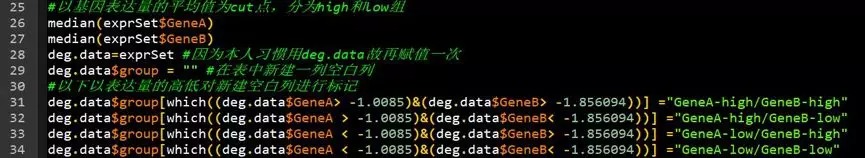
下面便是全文的关键。如何分组仁者见仁智者见智。我这里新建了一列用来标记基因表达高低。应该是比较通俗易懂的方法。
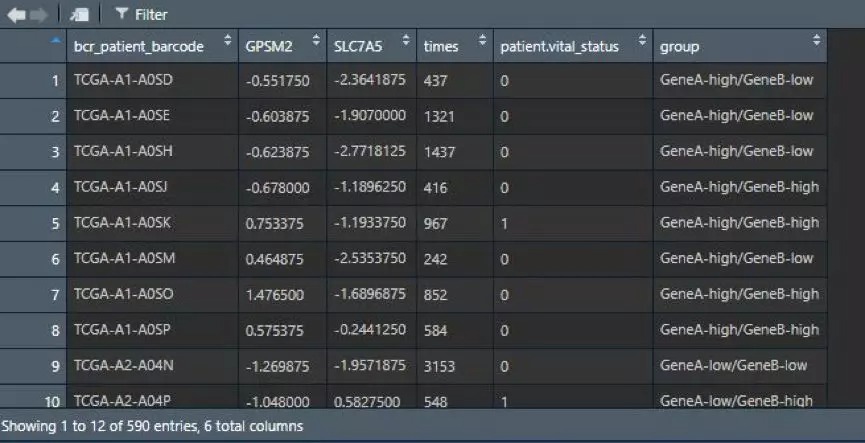
此时由于整合的缘故,数据框只剩590个样本。如图,最后一列为分组的标志。
最后就是常规的ggsurvplot画图。小伙伴们也可以用help命令仔细阅读该包的使用方法,做出你所需要格式的图片,这里就不再赘述。
至此,双基因的生存曲线就画完了。总的来说,双基因的生存曲线和单个基因的差别不大,重要是分组的思路。














