对于组学的数据,我们经常在得到一组的候选的数据之后,往往下一步经常都会去做富集分析来了解这些目标数据的具体功能。由于现在有各种各种各样的组学,比如转录组,蛋白质组,miRNA等等。因此我们就需要基于不同的组学使用不同的工具进行富集。这样的话,我们就需要掌握不同数据库的时候。今天就来给大家介绍一个可以对不同组学数据来进行富集分析的数据库:
GeneTrail(http://genetrail.bioinf.uni-sb.de/epi_upload.html)
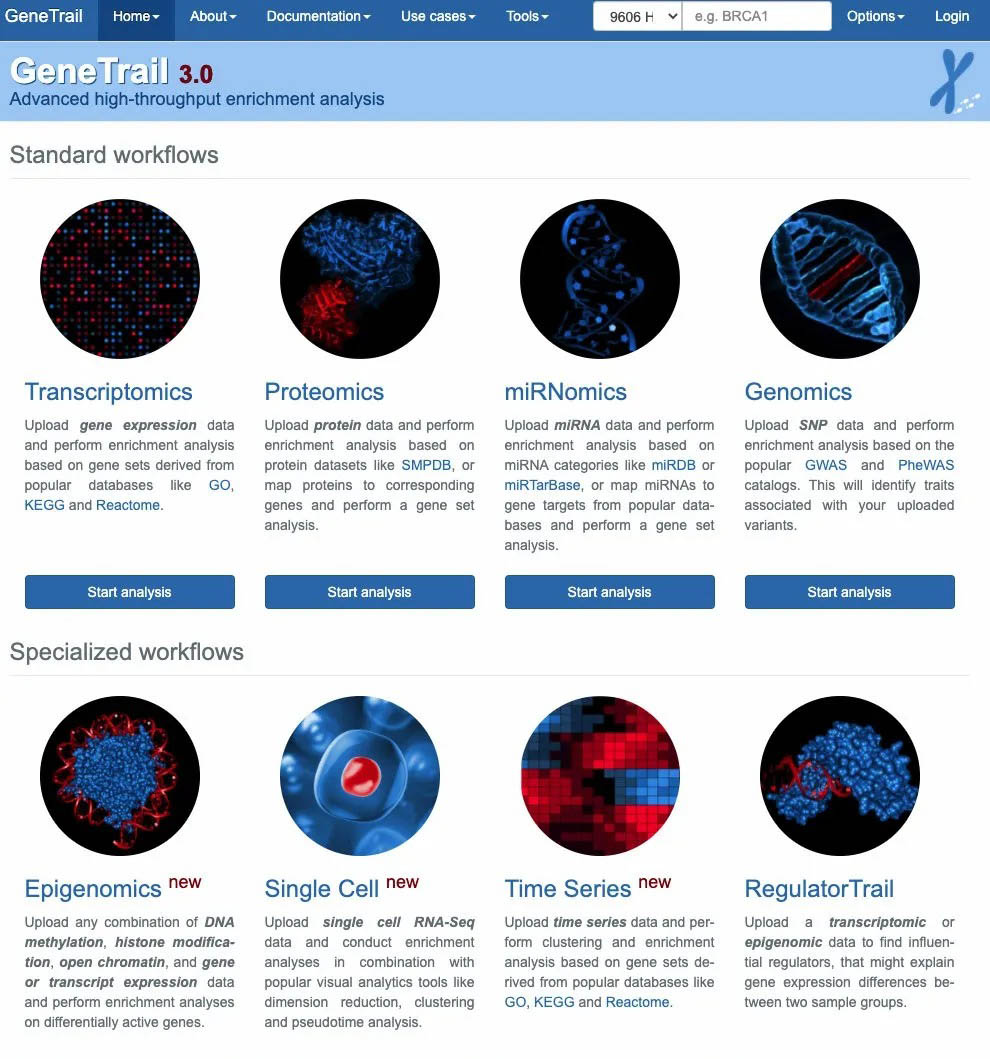
这个数据库可以对四种不同的组学进行分析:1) 转录组;2)蛋白质组; 3)miRNA组学;4)基因组(SNP);
同时也提供了四种其他组学的分析流程:1)表观遗传组学;2)单细胞组学;3)时间序列;4)基因调控组学
2.富集背景数据库介绍
我们之前对于富集分析有一个简单的介绍。本质上富集分析还是要基于不同功能注释的数据库来进行分析。因此了解分析的背景数据库可以更方便我们了解分析的结果
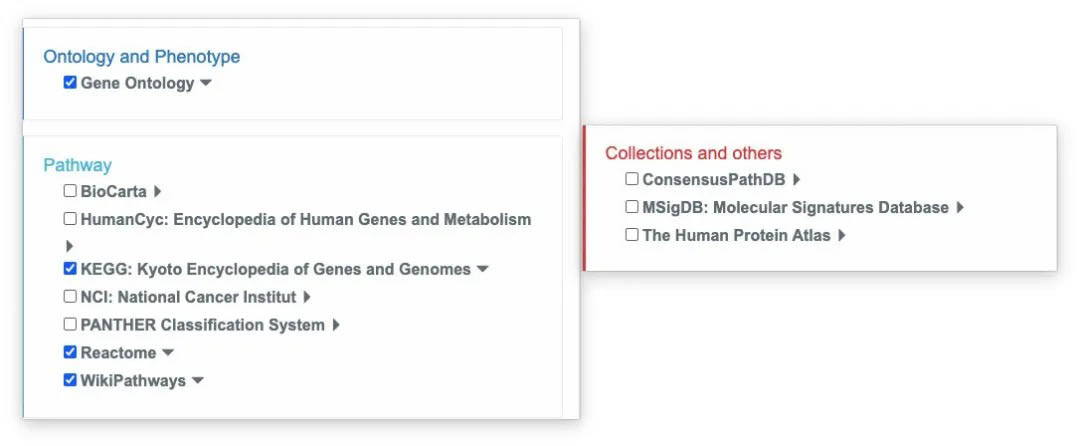
转录组数据:对于转录组数据而言,很多注释数据都是给予转录组的,所以我们可以进行多个数据库的功能注释,其中包括GO数据库;通路相关数据库基因组位置注释;调控标靶数据库;其他注释数据库;细胞标志物。

蛋白质组:对于蛋白的注释数据库就没有那么多了。基本的分析数据库就只有:GO分析、通路分析、其他数据库。
miRNA:关于miRNA功能的注释,主要是基于miRDB以及miRTarBase两个数据库的注释来的。
PS:miRNA功能富集的数据库随着miRNA研究的增多也越多越多。我们之前也介绍一个适用miRNA做GSEA分析的数据库: miRNA富集分析数据库
SNP:关于SNP注释的数据库就比较少了,主要还是集中在GWAS以及pheWAS两个数据库
数据库操作
由于数据库操作方式都类似,我们就选择最普遍适用的转录组的选择来进行演示。
3.1 数据上传
对于数据的上传,数据库主要分成了三种上传模式:
GSE号输入:如果我们要分析的是GSE数据库的数据,可以直接输入GSE号码即可直接进行分析
文件上传:我们可以上传自己想要分析的文件来进行富集分析。主要注意的是,这类的文件需要是txt格式的,同时是以tab为分隔符
文本复制:除了上传也可以直接把想要分析的内容复制到网站上面

另外对于数据的富集分析,我们之前介绍过,目前的主要的算法还是分ORA以及GSEA两种。这两种对于数据的上传要求是不一样的。如果我们只是想有ORA分析。只需要上传基因名即可。如果我们要做GSEA分析的话,则需要上传基因名+相对于的基因排名。同样的如果我们有原始数据,数据库可以基于原始数据,自己来分析进而进行富集分析。
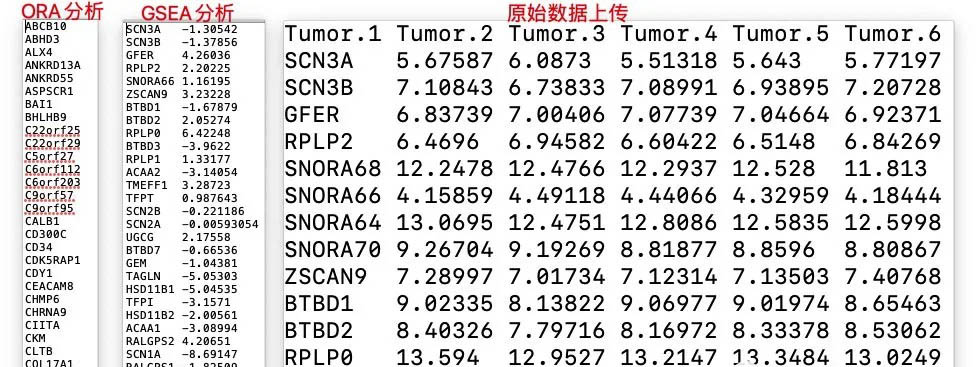
由于可以选择原始数据,所以我们就这里就上传原始数据来进行分析。对于原始数据的要求,第一列一定是基因名,第一行则一定是样本的分组信息。
3.2 数据分析
在上传的原始数据之后,我们需要对样本定义进行,哪些是control,哪些是case。
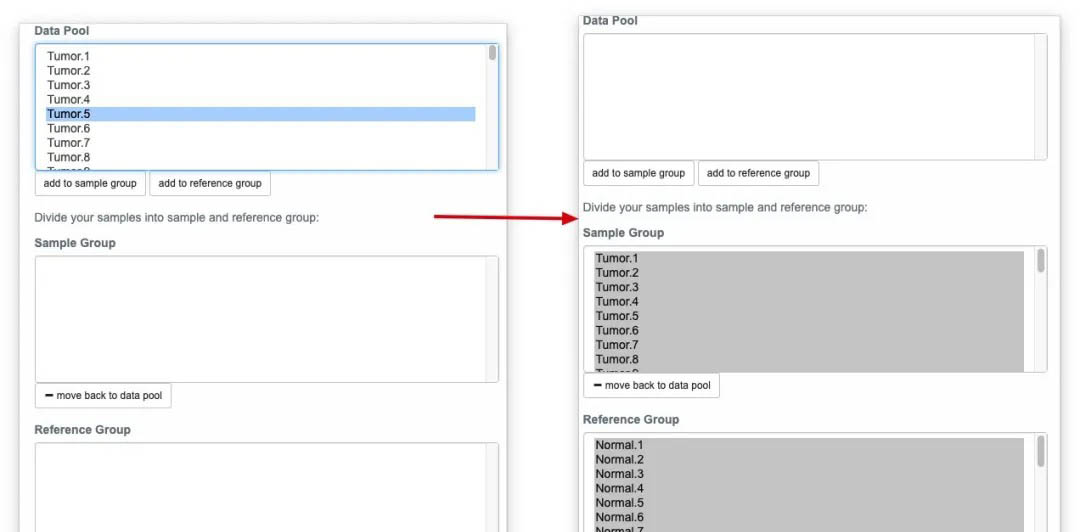
3.3 选择分析的算法
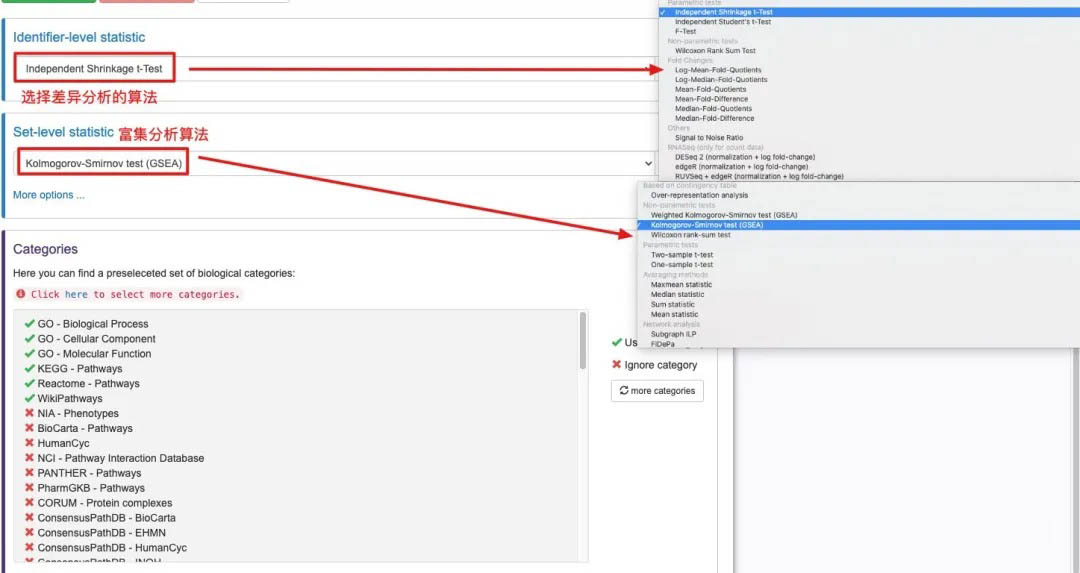
在定义好分组之后,就需要来定义对于分组进行什么样的差异分析同时对于差异的结果进行什么样的富集分析以及富集分析的数据库是什么。
3.4 结果展示
对于不同分析的算法结果展示是不一样的。对于GSEA而言,现在是可以看到一个汇总的图片
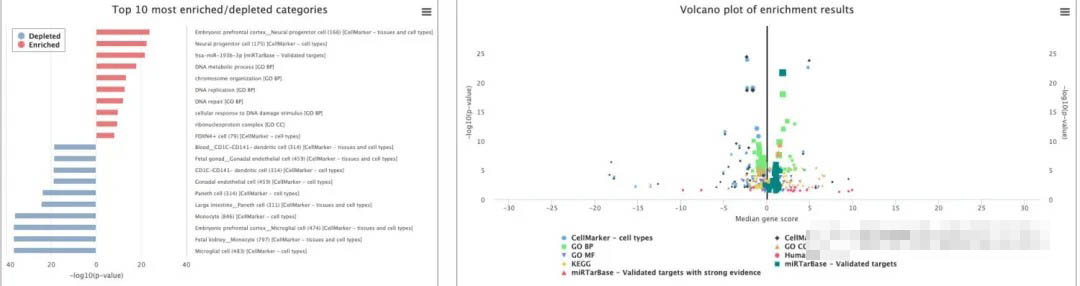
同样的,对于具体的某一个条目,我们可以看到具体的结果以及GSEA图片
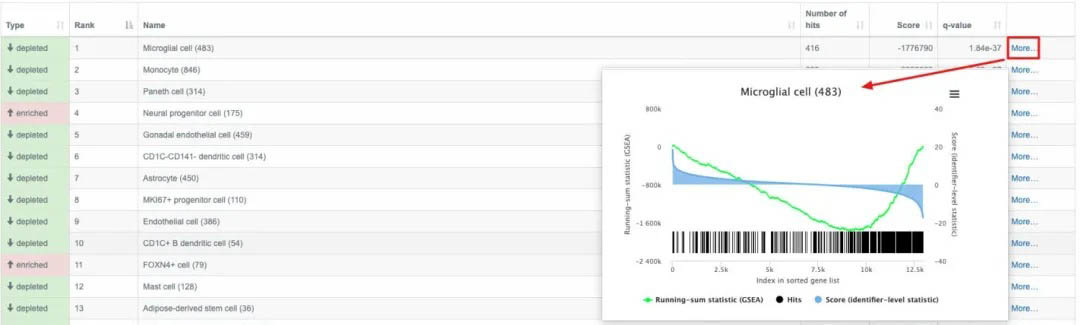
另外,相较于其他数据库而言,这个数据库的富集分析,还提供了一个反富集的选项。即我们在之前的分析当中是可以知道候选基因主要作用于哪个通路。这里可以反向的分析出某一个基因和哪些通路有关。这样就方便我们来进行筛选了。
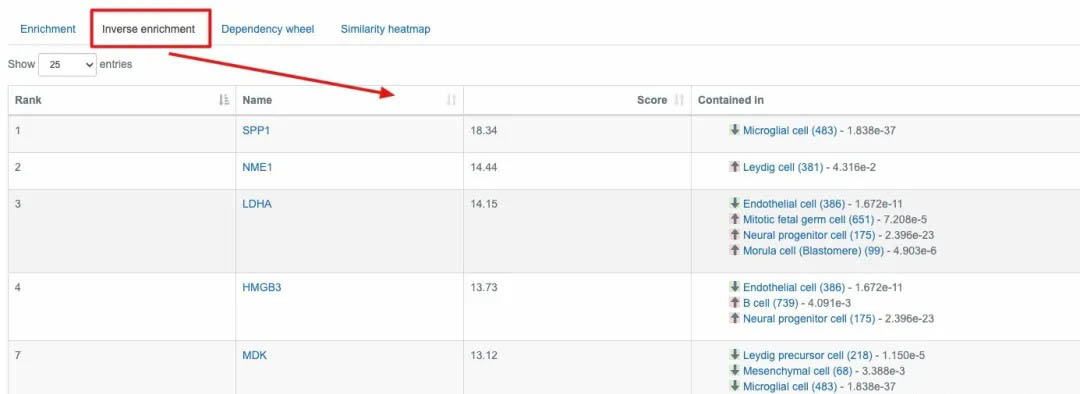
使用场景
由于数据库提供了多个组学的分析,所以对于任何组学想要做富集分析的需求,都可以使用。另外数据库提供了上传原始数据来进行分析的选项。所以方便不会做差异的同学进行GSEA分析。














