数据库背景介绍
首先这里有一个挺有意思的地方,我们如果在必应搜索“EBI数据库“显示出来的官方网址点击进入后会出现以下这个界面
相信一开始不了解EBI数据库架构的小伙伴们会发懵,所以晨曦在这里解释以下:
EBI(European Bioinformatics Institute)是指欧洲生物信息学研究所,是非盈利性学术组织EMBL的一部分。
也就是可以这样理解,EBI类似于NCBI是一个大型机构,所以里面会有很多类型的子数据库,如果我们直接在浏览器里键入EBI,大概率会转移到总数据库这里,我们这里进入scRNA-seq数据库的方法有两个
第一:直接输入以下网址进入
Home < Single Cell Expression Atlas < EMBL-EBI
第二:通过总数据库进入(分为三个步骤)
步骤一

步骤二
步骤三
那么讲解完如何进入这个数据库以后,我们来浏览一下这个数据库的数据规模
这个数据库最近更新是在2021年5月16日。
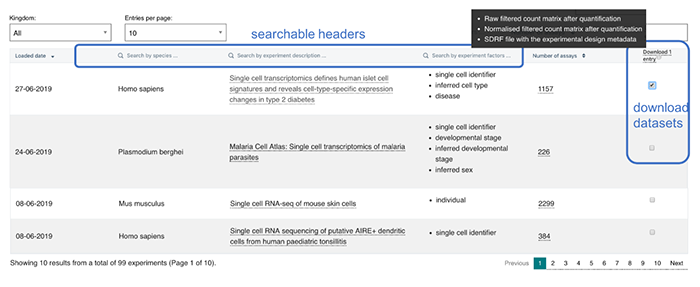
目前来看这个数据库包含了217个scRNA-seq研究、总计7,723,982个细胞,经过质控后得到来自18个不同组织共计5,312,183个细胞。
那么数据量介绍完毕后,下面开始介绍这个数据库的基本使用以及正文部分啦~
干货来袭预警
首先我们来到主页部分,看到下面这个界面就证明我们停留在主页部分
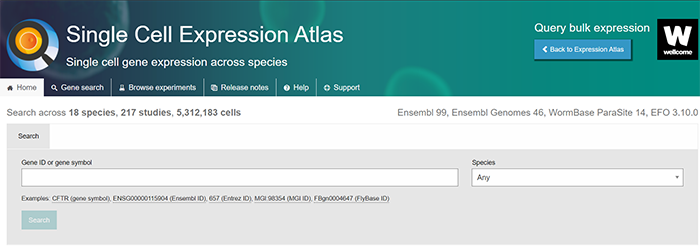
这里你可以使用Gene search box进行搜索,这里我们以Gene:CFTR为例子
搜索结果如下(搜索得到目标基因在某个细胞类型中的表达情况)
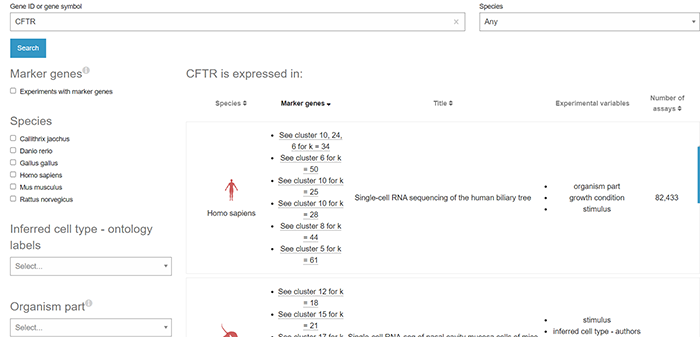
以上是加载的新界面,其中包含了目标基因表达的所有实验
EBI的单细胞数据库不像是panglaoDB数据库是以数据集为单位把数据存储起来,而是更像是按照实验或者研究为单位存储单细胞数据
回到正题
我们通过使用左边筛选栏对展现的结果进行筛选,比如说筛选条件从上到下分别是
1. 目标基因作为亚群的marker基因
2. 物种筛选
3. 限定目标基因所表达的细胞亚群类型
4. 限定目标基因所表达的组织类型
然后接下来我们进行结果正文部分的解读,我截取了我们检索结果的第一个
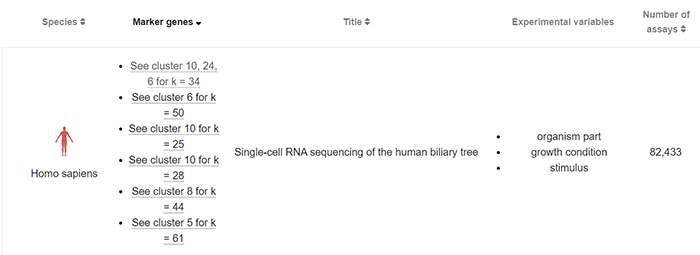
第一列为物种信息
第二列为目标基因作为marker基因所在的细胞亚群(K代表亚群数量,前面的数字则是代表在编号为多少的亚群中表达)
这里其实可以映射到scRNA-seq基本流程中的resolution参数
第三列为题目
这里再一次印证EBI数据库是以项目为单位整合的scRNA-seq数据
因为你可以在后续结果页面解读部分看到这部分信息(通过点击文章链接即可以达到文章界面)

第四列为该项目中的变量条件
第五列为数据的数量
然后我们点击进入结果展示页面,会出现下面这个展示效果(展示图的右上角可以提供下载链接)

这里我们分别解释一个画蓝色框选项卡里面的选项信息
t-SNE Perplexity
tSNE是一种2D图中可视化高度复杂数据的一种方法,尽管亚群和亚群之间大小和距离信息并不是十分有用的信息,但是你可以通过调节这个参数来达到重新展现可视化的目的
官网解释原文如下:
These are found in the t-SNE plots tab under Results. t-SNE plots are a useful way of visualising highly complex data in a 2D space. Although the size and distance between clusters is not very informative, manipulating the t-SNE perplexity value allows you to re-display the data in different ways.
Colour plot by
这里通过调整K值可以达到调整亚群数量的效果
这里需要注意一点,这里是基于Scanpy clustering algorithm算法来进行调整的
同时这个选项里还可以选择运用元数据集为集群着色,每组细胞的颜色图例显示在tSNE图层的底部,用于为集群着色的元数据集信息可以是:分组信息、前面提到的项目变量信息、组织来源信息等等。
提问:什么是scanpy?
回答:scanpy 是一个用于分析单细胞转录组数据的python库,文章2018发表在Genome Biology。其实它的许多分析思路借鉴了以seurat为中心的R语言单细胞转录数据分析生态的,scanpy以一己之力在python生态构建了单细胞转录组数据分析框架
小总结
这个数据库更像是把一个个单细胞研究,每一个研究弄成一个数据库,并且数据库与数据库之间的架构保持一致,这样做的好处在于,我们可以通过找到与我们研究相似的研究来获取数据,这里的相似可以是细胞亚群相似,也可以是组织来源相似。
回到正题,我们继续往下探索
我们将鼠标停在单个数据点上,会出现一个小弹框,如下:
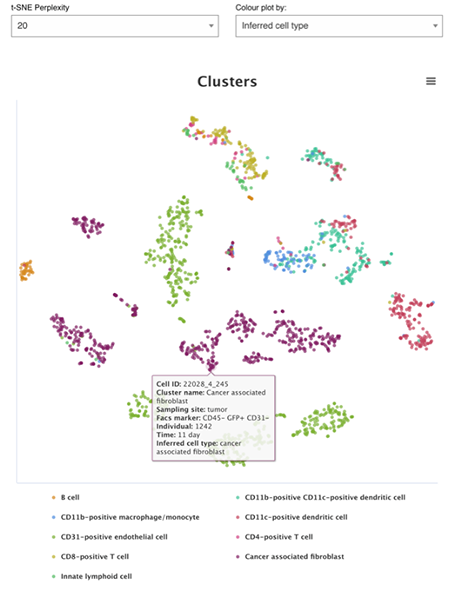
显示的相关信息会在弹框中显示~
都是很直接的概念,这里就不过多赘述啦~
然后让我们把目光聚焦到Gene expression,展示图如下

搜索框支持我们搜索Gene是否包含在细胞表达谱中及其表达情况
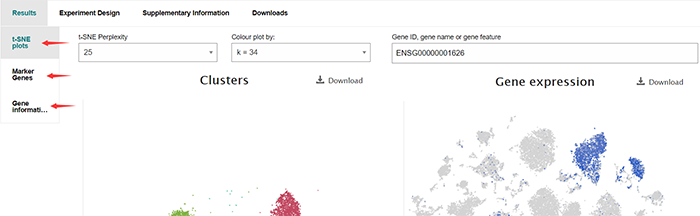
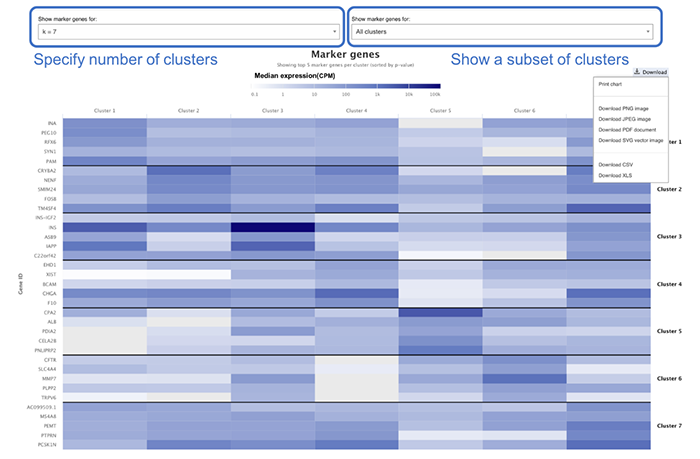
官网原文:Genes which comprise part of the specific expression profile for a cell population (cluster) are defined as marker genes. These are displayed - 5 per cluster in the Marker Genes tab under Results

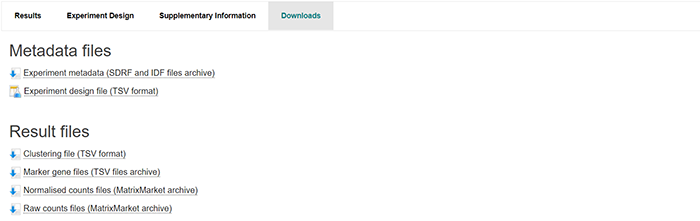
Metadata files: SDRF file (Sample to Data Relationship Format) - Creating an SDRF IDF file (Investigation Design Format) - Creating an IDF Experiment design file containing the sample characteristics and experiment variable summary table Results files: Clustering file: output of Scanpy analysis for clustering of filtered cells for the different k values (Scanpy clustering algorithm) Filtered quantification file: provides the measured gene expression in CPMs for each filtered cell alongside gene and cell ID Marker gene files: expression values for each marker gene per cluster Normalised counts files Raw quantification files
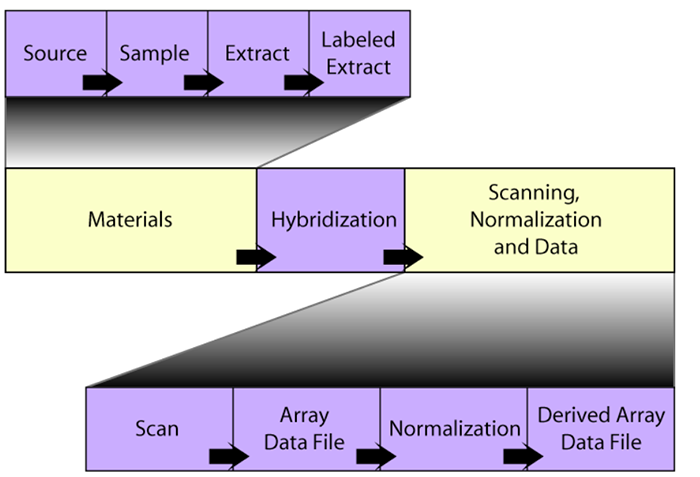
这里可能会有小伙伴对于SDRF文件有一些问题,我这里简单解释一下
这一部分讲解完毕~

本文来源:挑圈联盟














