在进行高通量测序的时候,我们在会得到很多的候选基因。但是对于基础实验而言,我们往往只需要寻找到这些基因当中重要的那些基因来进行后续的实验即可。但是这么多基因该怎么寻找的呢?
这个一般可以通过通路富集或者相互作用分析,来寻找一些关键的基因来进行后续的验证。但是的话,对于通路富集分析而言,由于是只是基于以往的研究结果来进行分析,所以对于一些新的基因或者目前研究较少的基因就容易造成分析的丢失。而对于相互作用分析而言,这个则是一种可以扩大候选基因选择的方法。因此对于后续基因的选择最好是结合这两种方法来综合性的分析。说了这么多,今天就给大家介绍一个综合了通路富集和蛋白相互作用分析的数据库:
SIGNAL https://signal.niaid.nih.gov
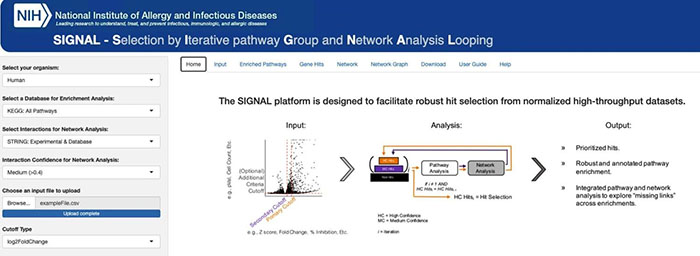
背景数据集介绍
SIGNAL是一个对候选的数据集进行通路和相互作用分析的数据库。综合两者综合性的结果,来确定在候选的一系列基因当中,那些是所谓的核心基因。
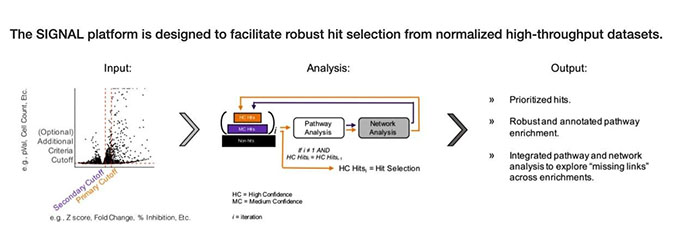
因此在这个数据库当中,内置了两个数据集,
通路相关数据库: 在SIGNAL当中主要是使用了KEGG数据库来进行通路富集分析
相互作用数据集: 这里主要是使用了STRING数据库进行分析。
数据库操作
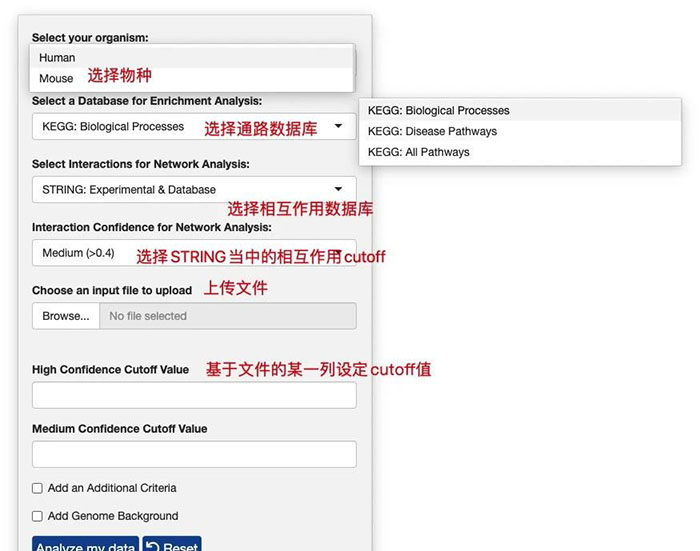
对于SIGNAL数据库而言,主要是通过左侧的选项栏,选择选择想要分析的参数,上传想要分析的文件同时选择cutoff值,最后点击Analyze my data即可。
数据上传
在数据上传方面,在SIGNAL当中,对于文件的格式有特定的要求。
上传的文件必须是: csv, txt 或者 xlsx格式的。
上传的文件的第一列必须是基因名(可以是GeneSymbol也可以是EntrezID),且列名也必须是GeneSymbol或者EntrezID。
上传的文件需要除了基因名这一列之外还需要包括一个或者或者多个数字信息。比如logFC, P值或者可信度这样的。
数值可以是连续值(例如 p 值范围或 Zscores 范围)或指定值(例如将值 1 分配给应被视为“高置信度”的所有 ID。值 0.5 到应视为“中等置信度”的所有 ID,对于应视为“未命中”的所有 ID,其值为 0)
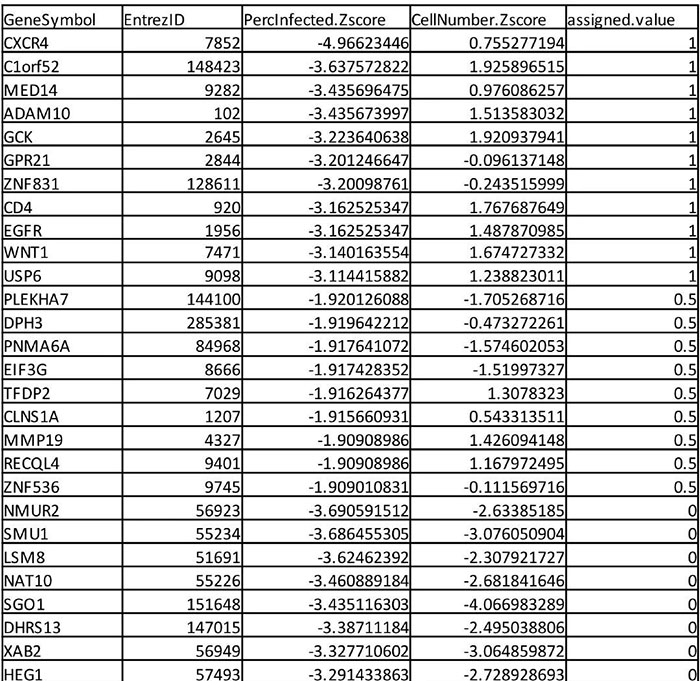
数据结果展示
在设定好cutoff值,点击分析之后,我们就可以获得相关的结果了。
首先在通路富集分析当中,我们可以看到通路分析的结果。同时点击相关的通路可以得到在KEGG当中具体的通路图和相关的基因注释。
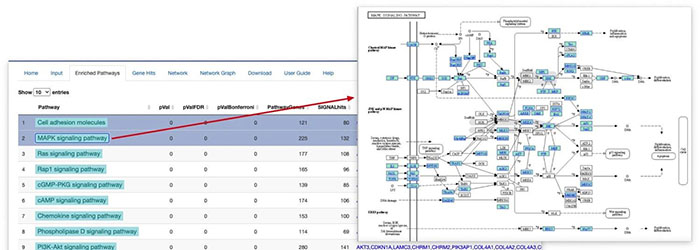
进一步,在Gene hits当中就可以了解核心基因是哪些了。
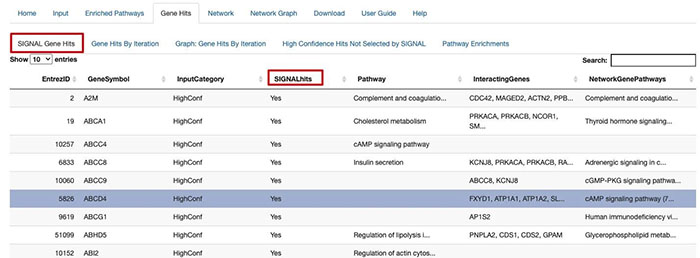
另外,这个数据库还有构建网络,下载所有数据这样的功能。这里就不一一介绍了
以上就是这个数据库的基本功能了。对于高通量测序得到的候选基因而言,如果精简一直是一个问题。在这个数据库当中,使用了两个方法来进行数据的分析,也算是一个新的方式。有需要的可以尝试一下这个数据库哈。














