数据库概览
cMap数据库(The Connectivity Map)由Broad研究所开发,最新版本于2017年发表在Cell杂志。该数据库基于164种药物/小分子化合物和过表达或基因敲除工具处理的细胞表达谱数据,借助L1000分析平台,探讨药物/小分子化合物、基因和疾病状态之间的交互网络关系。实际应用中,可将差异表达基因列表上传cMap与数据库参考基因集比对,根据差异表达基因在参考基因集富集情况获得相关性分数,数值在-100~100之间,正数表示差异表达基因与参考基因集正相关,负数差异表达基因与参考基因集负相关。
点击网址(http://www.broad.mit.edu/cmap)进入cMap旧版本Build2主页,普通注册即可使用,目前该版本不再更新,收录1300左右小分子相关的表达谱数据条目,检索框输入格式必须是GPL96探针号grp格式。
点击网址(https://clue.io/)进入cMap最新版本Clue主页,该版本目前收录约8000以上表达谱数据条目,包括小分子相关表达谱、基因敲除/过表达相关表达谱等。新版本需要以非盈利邮箱注册,比如学校edu邮箱。
以下以新版本为例进行介绍。新版本cMap功能众多,点击Tool可查看。
Query用于查询与表达差异基因集相似的基因表达谱; Command用于在数据库中快速查找感兴趣小分子和基因敲除/过表达相关的表达谱数据; Touchstone用于探索数据库中超过3000种药物和2000种基因信息及其相互间联系; Cell App用于探索数据库中超过3000个细胞系及其功能注释信息。
其他工具用法或有疑问可参考Help目录下Connectopedia页面。Developor目录下提供Python, Matlab, R和Java代码共学习使用。以下介绍以常见的Query和Touchstone功能为例。
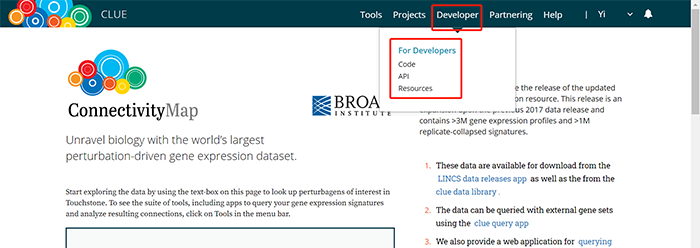
数据库功能及操作演示 1
Query功能模块
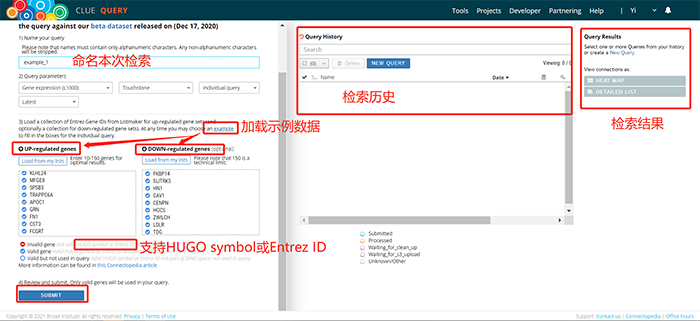
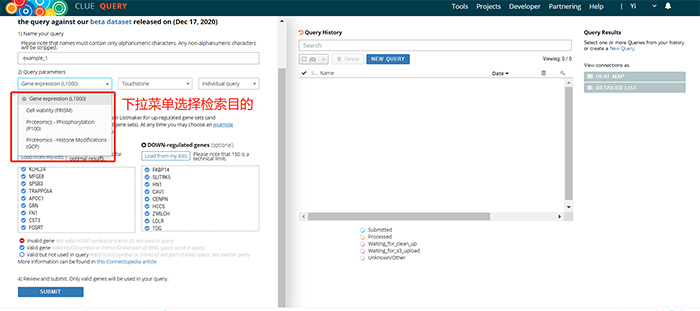
点击Tool目录下Query进入功能页面。首先,命名本次检索任务;然后,选择本次检索目标,可检索与上传表达差异基因集类似的基因表达谱,及细胞生存、磷酸化和组蛋白修饰相关的表达谱,此处以Gene expression为例;接下来,上传表达差异基因列表,可粘贴进输入框,或点击load from my files导入ListMaker工具构建的list数据,此处加载示例基因列表【(GSE33643) PI3K/MTOR inhibitor BEZ235 treated A2058 cells (3 doses at 24H) vs. DMSO treated】;其他参数默认,点击submit,等待分析结果。
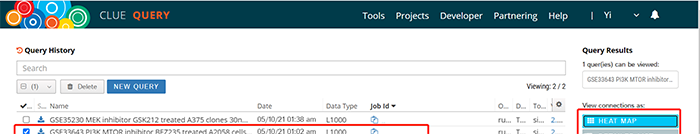
等待5~10min(甚至更久,取决于上传的基因集大小和网速)分析完成,点击Tools目录下History查看结果,可直接下载数据,或勾选后在右侧栏点击HEATMAP或DETAILED LIST查看详情。
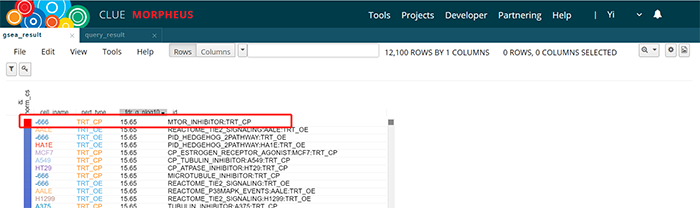
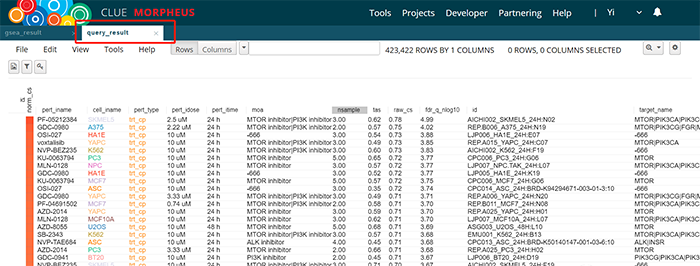
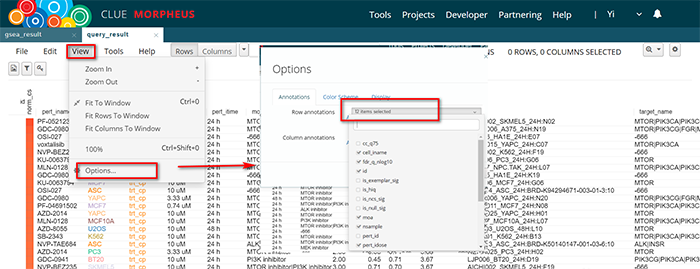
在GSEA结果栏,示例基因集在MTOR inhibitor这一药理功能富集分数高,与示例数据含义一致。在Query结果栏,展示更多信息。上述二者,均可在View目录下Option设置需要显示的列。
在ACE基因详情页面,Claim目录下同样是各个数据库来源中的基因基本信息和基因与药物交互信息。
2
Touchstone功能模块
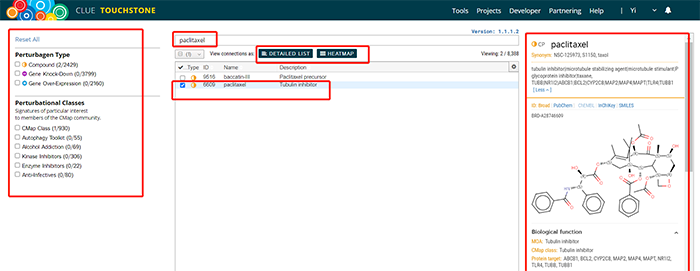
点击Tool目录下Touchstone进入功能页面,检索框输入紫杉醇(paclitaxel)为例。左侧显示数据库中,紫杉醇相关表达谱数据有2条,展示在检索框下方,描述信息为紫杉醇发挥Tublin inhibitor作用。右侧栏显示紫杉醇基本信息,包括药理学功能、化学结构式和特异性敏感的细胞系等信息。提供PubChem、ChEMBL、InChIKey和SMILES数据库链接。
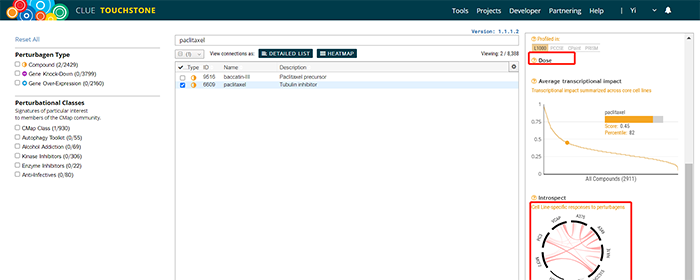
点击DETAILED LIST查看详细结果。左侧栏为结果总览,PHARMACOLOGIC展示富集分数绝对值大于90的药理学功能或基因组特征,排在第一个是Serotonin receptor agonist,排在第二个即是Tublin inhibitor。在右侧列表显示上述药理学功能或基因组特征对应的基因集信息,可检索感兴趣基因,并可下载该结果。
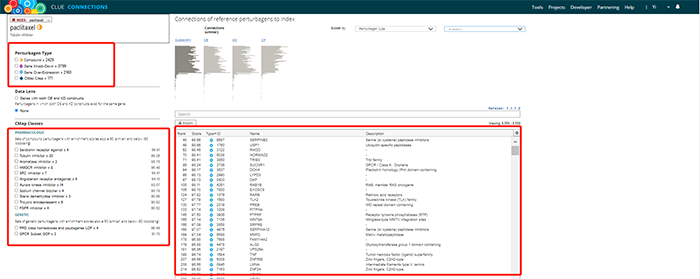
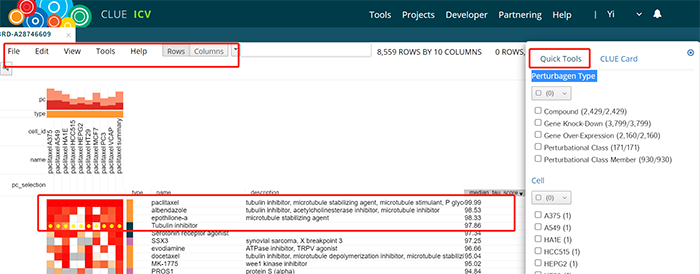
返回上一界面,点击HEATMAP,展示紫杉醇干预不同细胞的基因表达谱与其他药物/小分子和基因敲除/过表达相关表达谱的相似性热图。其中相似度较高的包括Serotonin receptor agonist和Tublin inhibitor,并提示紫杉醇与albendazole和epothilone-a干预细胞具有相似的下游机制。上方菜单栏有丰富选项,用于个性化设置和图片下载。右边Quick Tools中可分别以干预因素(Perturbagen Classes)和细胞系对结果进行筛选。
文献应用案例
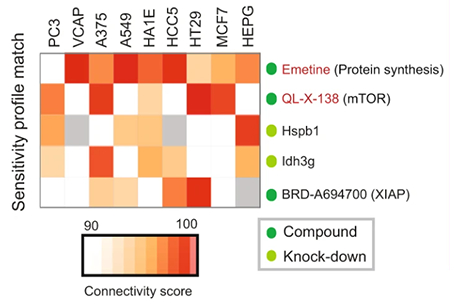
Notch1信号通路是T细胞急性淋巴细胞白血病(T-ALL)关键致癌因素,γ-分泌酶蛋白酶参与诱导Notch受体蛋白水解,使得Notch细胞内域(NICD)释放入核,启动下游靶基因转录。阻断Notch裂解和随后转录调控的靶向治疗为T-ALL患者带来希望,如γ-分泌酶抑制剂(GSI)的应用。本文探讨GSI抗性机制问题,基于蛋白质组学数据富集分析结果显示,GSI抗性涉及氨基酸合成和分解代谢、脂肪酸降解和活性氧(ROS)代谢过程。接下来,作者在cMap数据库检索得知,GSI敏感细胞基因表达谱与蛋白质合成抑制剂Emetine表达谱相似性最高,其次是mTOR抑制剂QL-X-138。进而推知,GSI抗性与mTOR信号或蛋白质合成抑制相关。
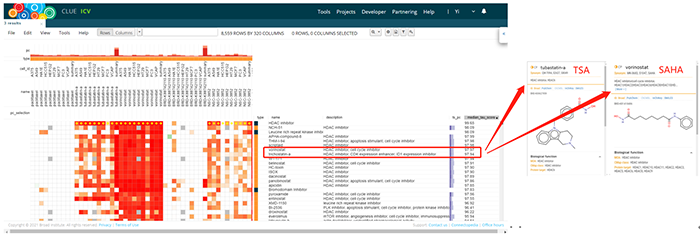
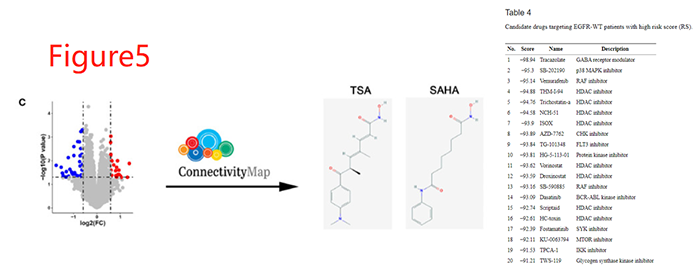
临床上,EGFR突变非小细胞肺癌(NSCLC)患者从EGFR酪氨酸激酶抑制剂(EGFR-TKIs)中受益匪浅,而缺乏EGFR敏感突变非小细胞肺癌(EGFR-WT NSCLC)患者预后很差。单因素和多因素Cox回归将CYP11B1和DNALI1确立为EGFR-WT NSCLC患者独立预后因素。基于此将EGFR-WT NSCLC患者分为高危组和低危组,组间基因表达谱的差异分析鉴定出25个上调基因和36个下调基因。接下来,将上述差异表达基因上传cMap数据库进行比较,获得24种候选药物,9种被归类为HDAC抑制剂。细胞实验检测两种常见HDACi抑制剂TSA和SAHA对EGFR-WT NSCLC细胞(A549和H1299)作用,结果显示TSA和SAHA抑制EGFR-WT NSCLC细胞增殖和迁移,提示TSA和SAHA作为EGFR-WT NSCLC治疗新策略的潜在价值。本文上述结果展示在Figure5C和Table4。
单图复现如下:
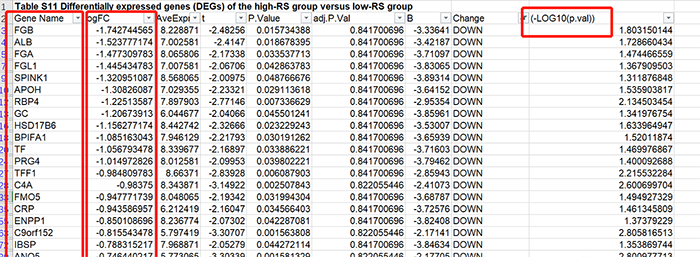
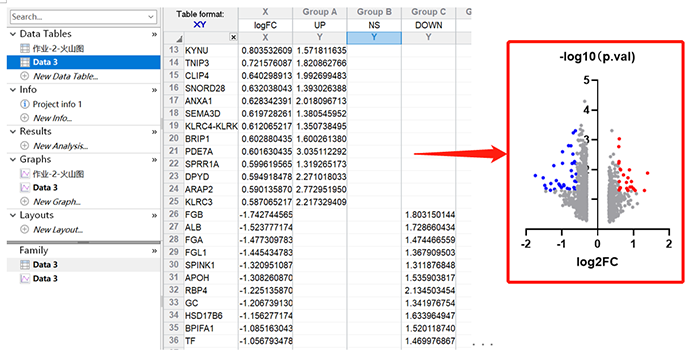
下载本文TableS12,为EGFR-WT NSCLC患者高危组和低危组之间基因表达谱差异分析结果,打开EXCEL筛选功能,将36个下调基因的基因名、logFC值和-log10(p.value)值,以及25个上调基因和无显著差异基因对应的数据,全部复制粘贴进GraphPad下XY数据框,如下图所示,绘制火山图。
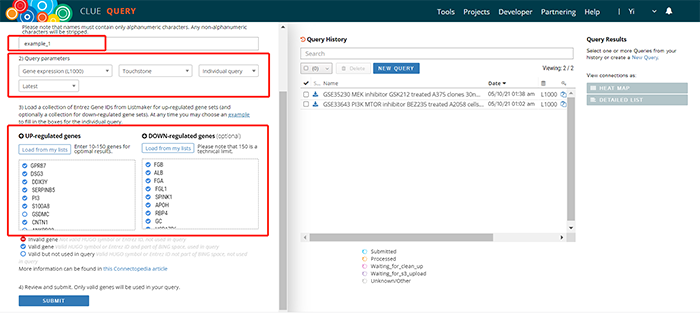
然后进入cMap数据库,点击Tool目录下Query进入功能页面。首先,命名本次检索任务;然后,选择本次检索目标Gene expression;接下来,将上述上、下调基因列表粘贴进输入框;其他参数默认,点击submit,等待分析结果。
分析完成,点击Tools目录下History查看结果,勾选目标任务,在右侧栏点击HEATMAP查看详情。热图展示320条结果,可根据评分排序,绝对值90以上的药物达30多种,与原文略有出入。分别点击trichostatin A (TSA)和vorinostat (SAHA)可查看药物结构式。
将上述火山图、药物结构式图拼图,即可得原位Figure5;将上述候选药物整理,即可得原文Table4。
以上就是cMap数据库的全部内容,开发并维护数据库不易,小伙伴们使用时别忘记引用以下参考文献!














