有一个数据库:FusionGDB 值得推荐给你!
https://ccsm.uth.edu/FusionGDB/
一、 融合基因
二、FusionGDB
1. 数据来源
2. 主要注释内容
3. 搜索方式
4. 具体内容
一、 融合基因
顾名思义,融合基因就是两个基因“融合”在一起。正常情况下,两个基因相隔很远互不干扰,但是由于病理的染色体重排,导致相隔很远甚至是不在同一条染色体的基因组合在一起。
一般,融合基因是由染色体重排而产生的,包括染色体的易位,插入,颠倒,缺失。
基因融合会产生基因融合转录本和嵌合蛋白产物,它们已被用作治疗的靶标。众所周知的例子是靶向BCR-ABL1基因融合的格列卫(Imantinib)和靶向EML4-ALK基因融合的克唑替尼
二、FusionGDB
现介绍一款能查询融合基因功能的数据库——FusionGDB
1. 数据来源
FusionGDB: Fusion Gene annotation DataBase (uth.edu) 
数据来源:包含三个数据库分别是ChiTaRS 3.1、TumorFusions以及一篇基于TCGA分析融合基因文章(PMID: 29617662)。最终收集到了48117个融合基因,同时也可进一步对这些融合基因进行肿瘤相关的功能注释。 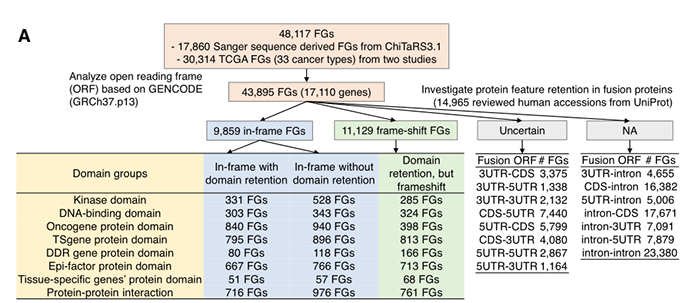
2. 主要注释内容
对于这些的融合基因,作者提供多种注释方式。
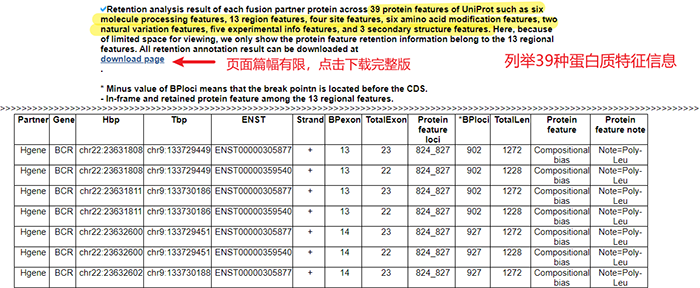
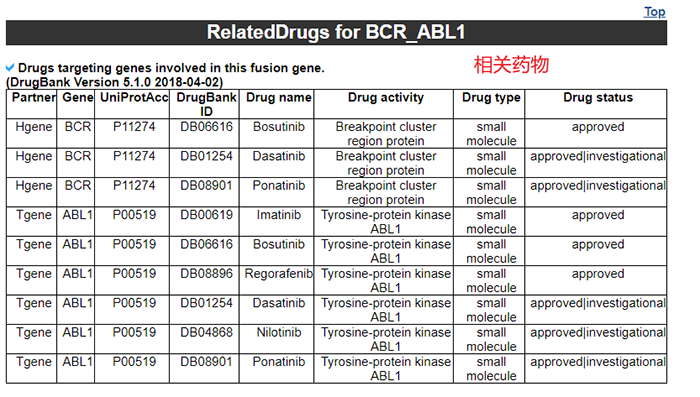
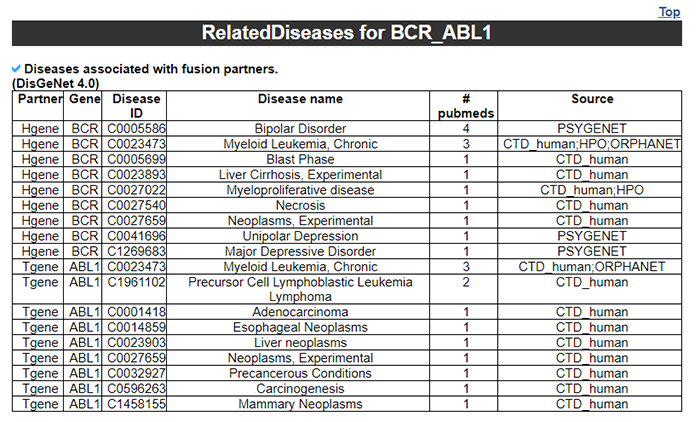
基因基本信息 癌症中的评估得分 生物过程基因本体论 功能描述 39个蛋白质特征(来自UniProt的六个加工特征,13个区域特征,四个位点特征,六个氨基酸修饰特征,两个自然变异特征,五个实验信息特征,三个二级结构特征)和PPI网络 相关药物及疾病
3. 搜索方式
页面的搜索方式有六种
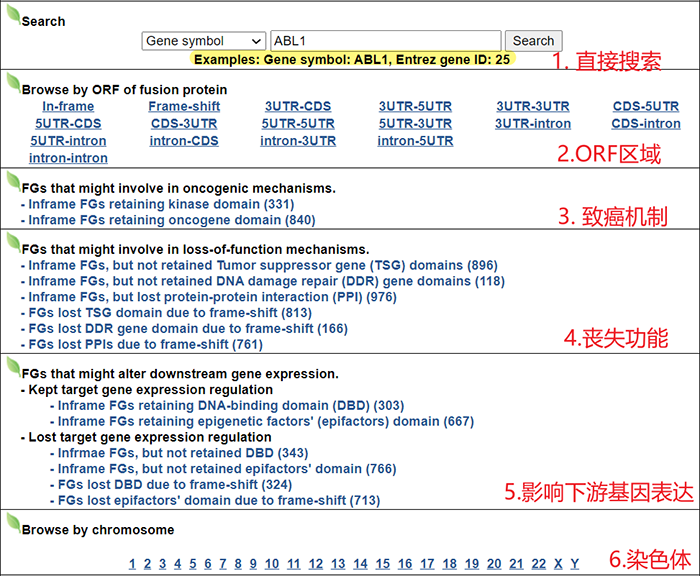
4. 具体内容
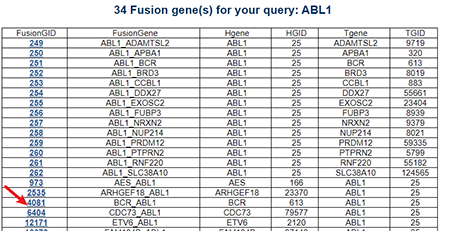
4.1 填写symbol或者entrez id

4.2 点击感兴趣融合基因
得到34个Fusion genes,点击感兴趣的基因
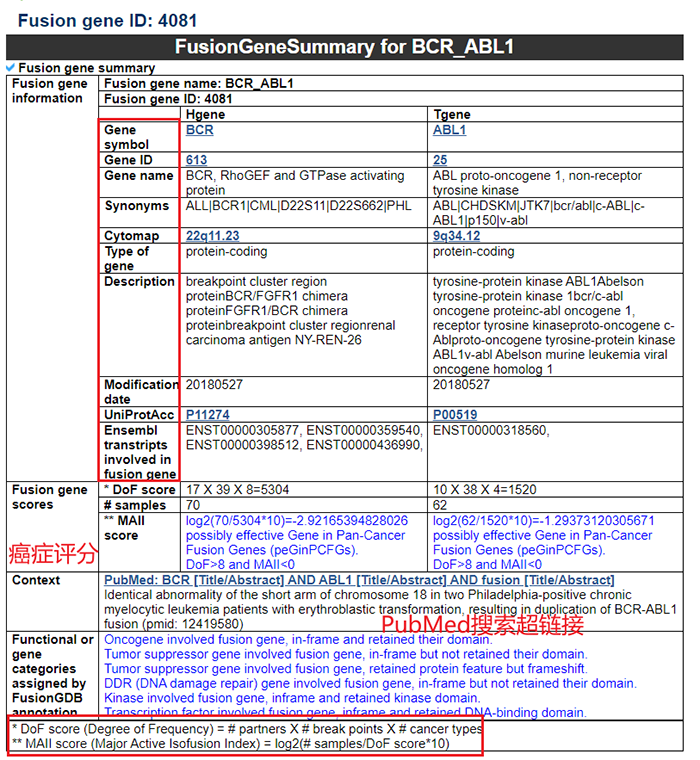
主要出现六个方面结果

4.3 基本信息
表格详细给出了对应symbol,gene ID,位置,癌症评分等信息,并附上对应数据库超链接,非常方便
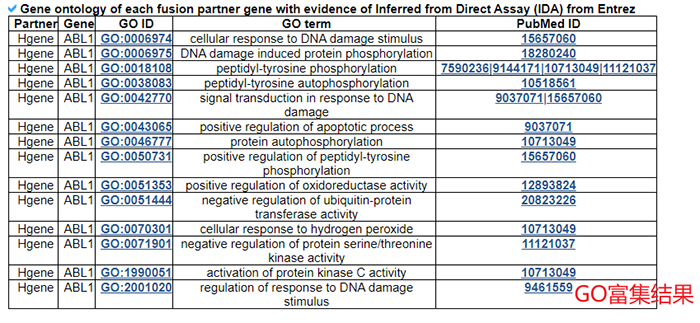
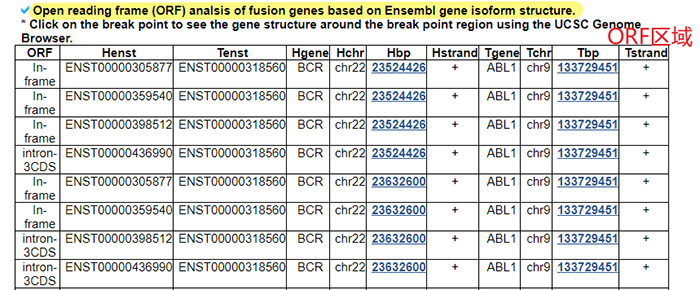
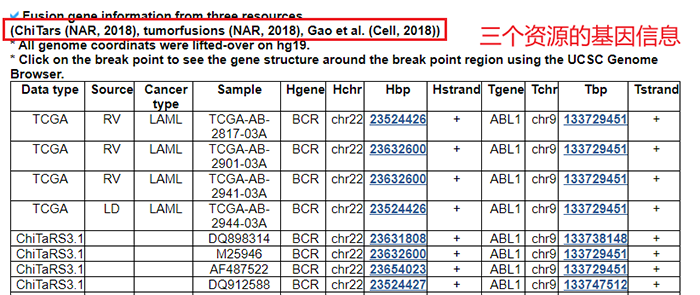
4.4 蛋白质特征
4.5 基因序列
4.6 PPI网络
4.7 相关药物
4.8 相关疾病
4.9 下载
下载页面有对应内容及数据库
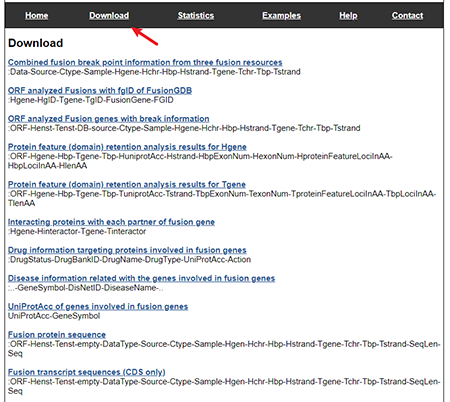
更多信息可查看:FusionGDB: fusion gene annotation DataBase - PubMed (nih.gov)