对于相互作用分析的而言,我们之前介绍过STRING、BioGRID和ConsensusPathDB两个数据库。其中STRING和ConsensusPathDB是一个综合性的查询蛋白相互作用的数据库;BioGRID是一个查询单基因相互作用的数据库。这些综合性的数据库往往没有限制疾病的检索。但是对于不同的疾病,基因内部的作用机制往往是不同的。所以传统的相互作用往往不一定适合具体的疾病。这个时候往往需要具体疾病的高通量测序的数据来进行进一步的验证。所以今天就来介绍一个可以对肿瘤相关疾病进行相互作用和验证的数据库:PINA(https://omics.bjcancer.org/pina/)

1.背景数据集介绍
对于基因之间的相互作用关系而言,PINA是整合了多个数据的结果来进行构建的。这个数据库整合了IntAct、BioGRID、MINT、DIP以及HPRD这些相互作用数据库的数据。
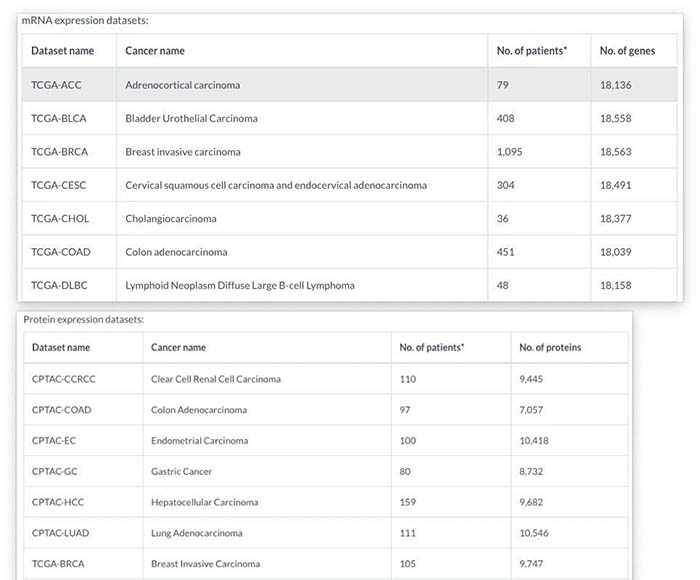
而对于后续相互作用在肿瘤当中的分析则是使用了TCGA的RNA-seq以及部分TCGA+CPTAC的蛋白质谱数据
对于整个蛋白网络数据库而言,主要包括两个使用的步骤,一个是构建一个蛋白相互作用数据库;另外一个对于这个网络进行进一步的分析
2. 相互作用网络构建
对于相互作用网络构建而言,数据库提供了四种构建网络的方法:1) 输入一个蛋白查找和这个基因相关的相互作用网络;2) 输入一系列的基因/蛋白,寻找和这一系列基因/蛋白相互作用的基因/蛋白;3) 有目的性的查询两个基因/蛋白之间的相互作用关系;4) 查找两组基因之间内部的相互作用关系
例如我们这里想要查找这三个蛋白相互作用的蛋白有哪些
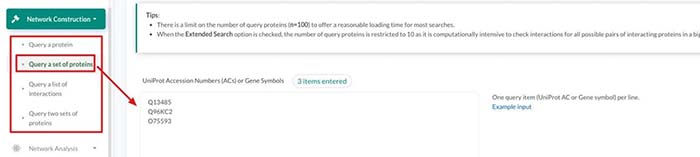
数据库可以输入基因名和uniprot的蛋白ID。关于两者之间的转换时通过biomart来进行转换的
输入之后,我们就可以获得和这三个蛋白存在相互作用的其他蛋白。整个结果界面包括两个部分,














