ChIPBase数据库网址为
http://rna.sysu.edu.cn/chipbase/,推荐大家使用的时候使用Google浏览器哦,会避免很多不必要的bug。
使用数据库的同时请记得引用参考文献:
ChIPBase v2.0: decoding transcriptional regulatory networks of non-coding RNAs and protein-coding genes from ChIP-seq data. Zhou KR, Liu S, Sun WJ, Zheng LL, Zhou H, Yang JH*, Qu LH*. Nucleic Acids Res. 2017 Jan 04;45(D1):D43-D50.
一、ChIPBase数据库基本介绍
输入网址http://rna.sysu.edu.cn/chipbase/,进入ChIPBase数据库主页面:
ChIPBase数据库是由中山大学生命科学院曲丽恒教授团队开发的一个在线工具,网页右上角标明了中山大学的校徽。数据库中还有一个中山大学开发的神器,即Encori数据库(原Starbase数据库)。需要提醒一下大家的就是,这两个数据库能不能使用全看缘分,有时会被禁止访问,如果大家进不去,说不定过段时间就好了。主页右侧的“Release notes”显示了该数据库的更新日志,最近一次更新时间是2016-11-23,目前是2.3.4版本。ChIPBase数据库收集了来自10个物种共10200个数据集的chip_seq数据,整理出了转录因子和各种基因之间的转录调控网络,还包括了组蛋白修饰数据。我们将页面下拉看看大致分析流程:
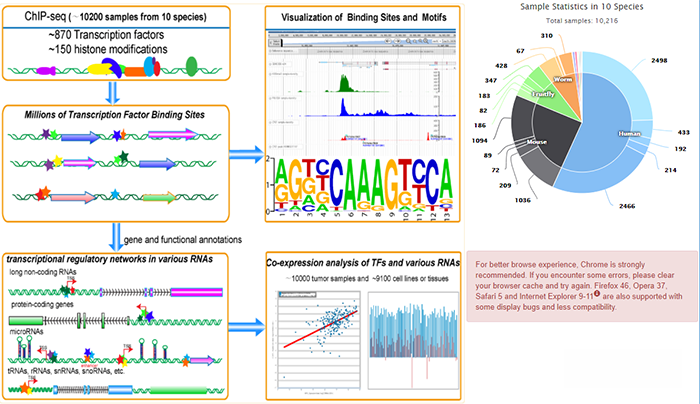
简单来说,就是从转录因子的chip_seq数据中分析得到转录因子结合位点的区域,称之为peak;通过peak可以进一步分析结合区域的motif;还可以通过基因注释得到转录因子和各种基因之间的调控关系;最后通过分析近10000个肿瘤样本及9100种细胞系和组织,得到转录因子和各种RNA之间的共表达关系。
二、ChIPBase数据库功能介绍
接下来我们看一下ChIPBase数据库的具体功能有哪些。
主页上方的菜单栏列出了网站的各功能模块,包括用于检索转录因子调控的lncRNA、miRNA、其他非编码RNA以及蛋白质的“LncRNA”, “miRNA”, “OtherNcRNA”, “Protein”。这些模块的使用极其类似,我们以LncRNA为例进行介绍,其他类型的非编码RNA大家可以举一反三。
1
已知转录因子,检索受其调控的LncRNA/miRNA/OtherNcRNA/Protein
点击菜单栏的“LncRNA”,进入以下界面:
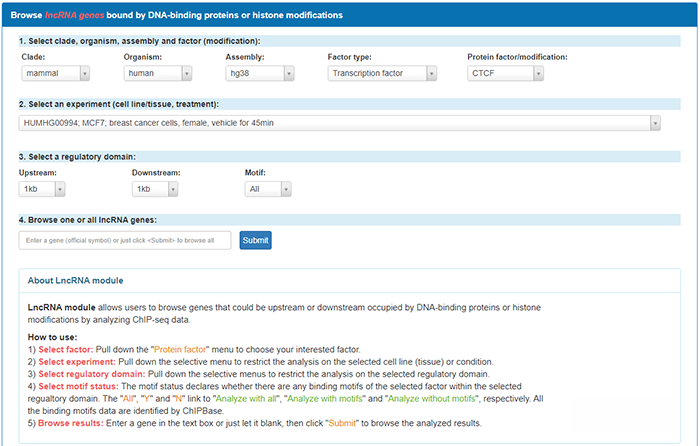
该页面需要用户在检索前进行多项选择,我们依次来看一下。
Clade只能选哺乳动物;
Organism可以选择人或小鼠,此处我们以人为例;
Assembly参考基因组版本选择hg38;
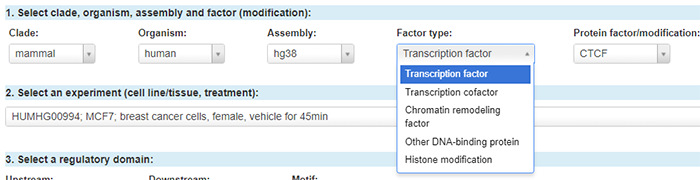
Factor type(因子类型),可以选转录因子、转录辅助因子、染色质重塑因子、DNA结合蛋白以及组蛋白修饰,可见网站的功能还是很丰富的。由于本次介绍的重点在于转录调控,此处我们选择转录因子。
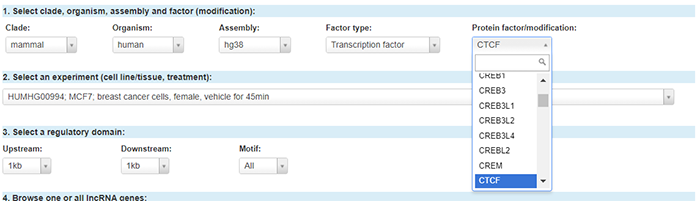
Protein factor/modifications选择目标转录因子。这里只能从已有ChIP-seq实验数据的转录因子中进行选择,此处我们以CTCF为例。
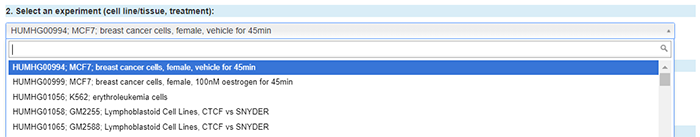
接下来选择细胞系,细胞系也是在有限的范围中进行选择。此处我们以第一条HUMHG00994细胞系为例;
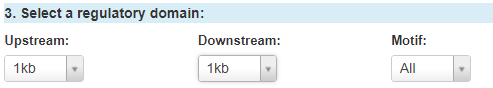
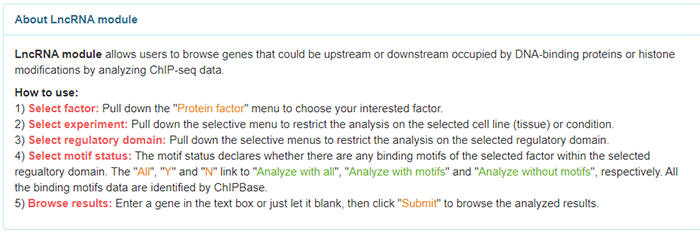
接着是选择分析的区域,默认是上下游1KB的区域,可选范围是转录起始位点上游30KB到下游10KB。Motif即DNA上结合转录因子的基序,可以选All、Yes或No,一般就选All。注意,上述这些选项的含义在网页下方About LncRNA module这块都有说明,小伙伴们若有疑问也可自行查看:
接下来到最后一步:
此处可以输入某一个特定LncRNA,检索其与转录因子的调控关系,也可以不输入任何分子,显示受该转录因子调控的所有LncRNA。这里我们就不输入了,直接点击右侧“Submit”,跳转到如下界面:
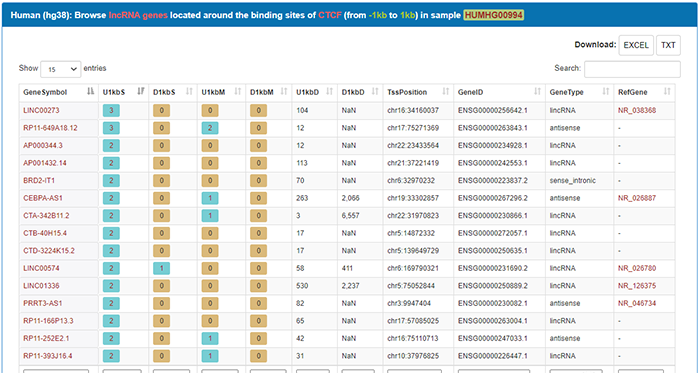
我们来阅读一下当前页面的内容。最上方的蓝色标签栏显示了人类hg38基因组上,在HUMHG00994细胞株中,转录因子CTCF调控的LncRNA,查看的是LncRNA宿主基因转录起始位点上下游1kb的区域。表格从左往右依次为LncRNA名称,位于上游1kb内的结合位点总数(U1kbS),位于下游1kb内的结合位点总数(D1kbS),位于上游1kb内的含有motif的结合位点的数量(U1kbM),位于下游1kb内的含有motif的结合位点的数量(D1kbM)。
结合位点与基因的转录起始位点(TSS)上游的最近距离(U1kbD),是结合位点与基因TSS下游的最近距离(D1kbD),转录起始位点在基因组中的位置(TssPosition),LncRNA的Ensembl ID和类型。右上角的Search框可以通过搜索过滤结果。
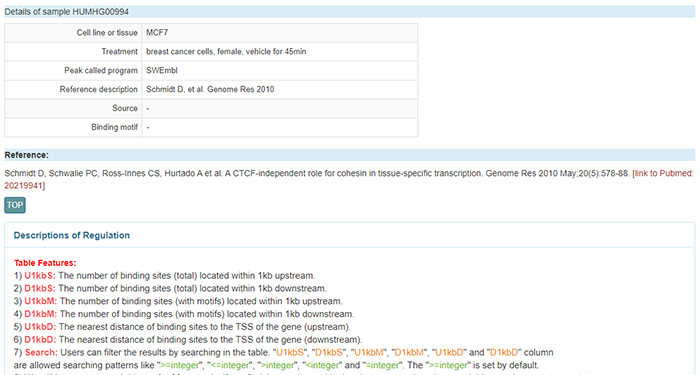
页面下拉,可以看到本次样本的详细信息,包括细胞株、处理情况、峰值程序、参考文献、GEO数据库来源等。最下方提供了上述表头的注释。
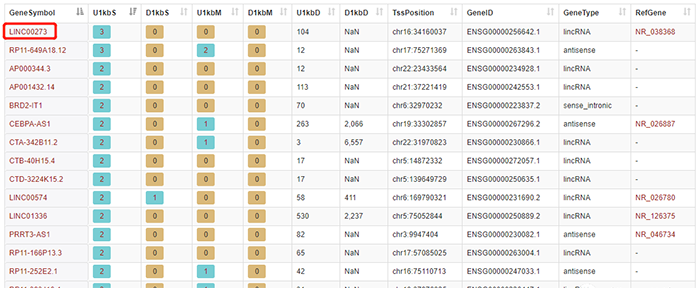
现在我们点击第一个LncRNA“LINC00273”查看一下结果:
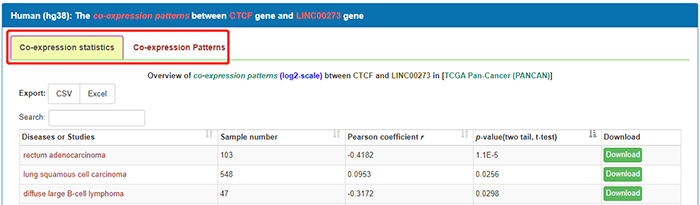
可以看到有共表达统计(Co-expression statistics)和共表达模式(Co-expression Patterns)两个菜单。在co-expression statistics菜单下可以看到该条lncRNA在不同肿瘤类型中的情况。包括样本数、Person相关系数和p值。点击download可以下载结果文件,有CSV和excel两种格式。
点击co-expression patterns,查看转录因子与我们选择的lncRNA在不同条件下的表达相关性:
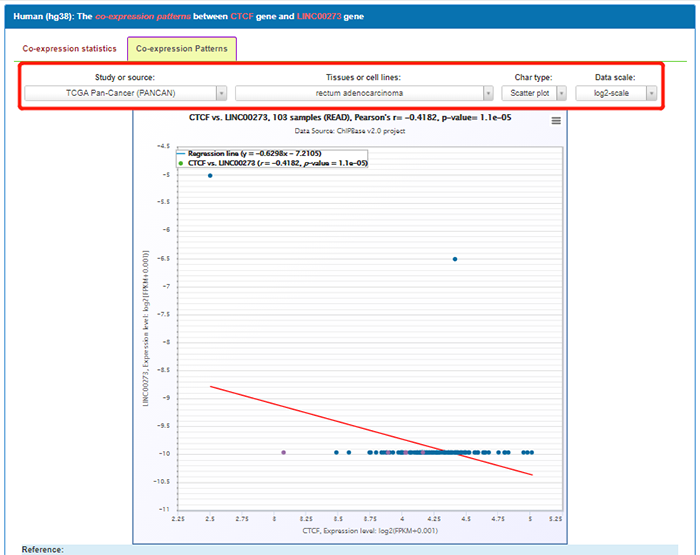
不同条件指可以通过下拉菜单选择研究来源,TCGA或GTEx项目;还可以选择不同的组织或细胞株。在chart type下,可以选择将数据以散点图、箱式图或柱状图的形式展现;Data Scale可以选择log2 scale或FPKM。
现在返回lncRNA列表:
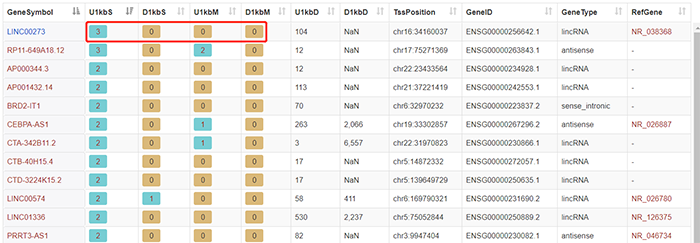
刚才我们查看了第一条lncRNA的信息,现在我们看第二列的“3”,这个3表明转录因
子CTCF在该lncRNA上游1kb范围内有三个结合位点。点击“3”。可以看到结合位点的具体信息,包括在染色体上的具体位置、长度等。数字“0”表示相应的区域没有结合位点:
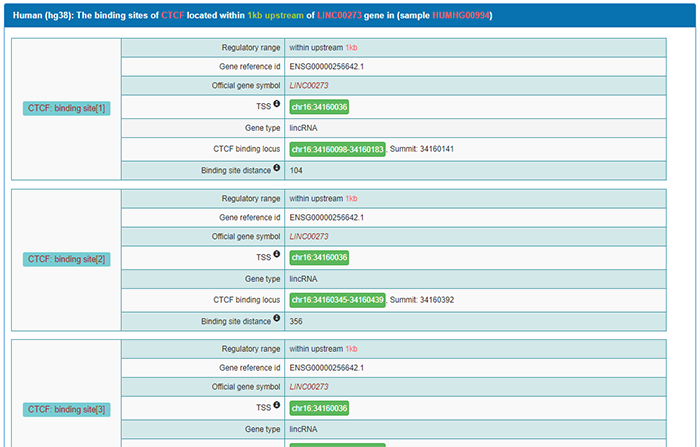
miRNA、otherNcRNA、Protein的检索方式与LncRNA一样,就不一一演示了。
2
已知目的基因,检索可以与之结合的转录因子
我们接下来看Regulator模块。该模块可以检索给定基因的转录起始位点附近-30KB到10kb区域内的DNA结合蛋白或组蛋白修饰,并提供相关结合位点的实验、文献证据。前面介绍的四个模板是通过转录因子找下游靶基因,这个模块就是找调控目的基因的转录因子了。
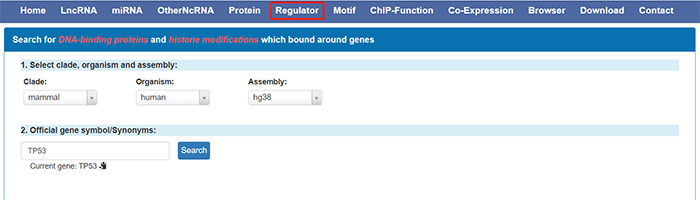
我们以TP53为例,点击search后显示结果:
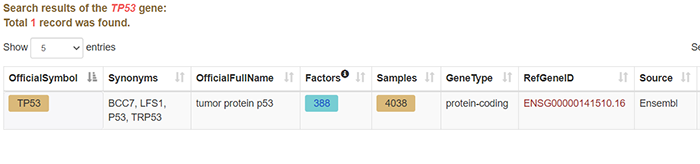
表格展示了TP53基因的一些基本信息,包括别名,各种官方命名。我们这里重点关注的是Factors,为TP53基因附近 DNA结合蛋白和组蛋白修饰的数量。共388个。我们点击看一下:
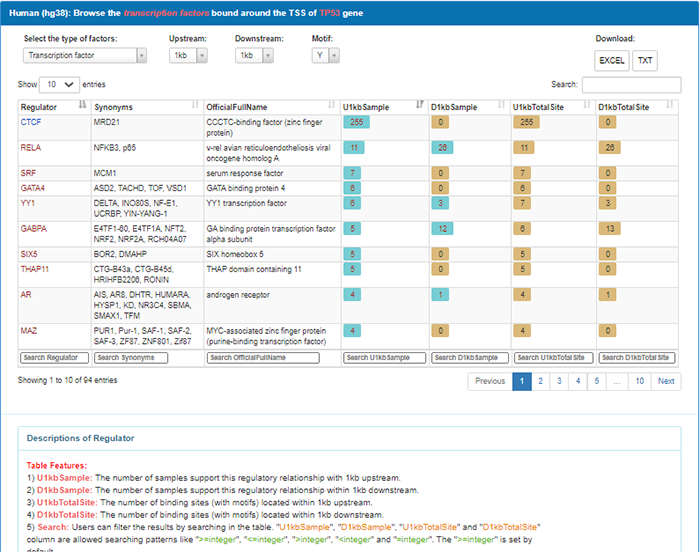
页面刷新后,可以通过上方的四个下拉菜单选择性地显示388个中的部分结果。如type of factors,默认的是transcription factor转录因子,还能选择转录辅助因子、组蛋白修饰等。旁边upstream、downstream用来界定查找的范围,最大范围是-30kb到10kb。Motif选择Y是提供motif序列的。下方的表格显示的是基于上面条件过滤后的结果。第一列为转录因子名称,第二列为别名,第三列为全称,第四第五列为支持相应区域结合关系的样本数量,第六第七列为相应区域的结合位点数量。页面最下方有对最后四列的注释。表格最后一行可通过search进一步过滤结果。
现在我们选择一个转录因子,如CTCF:
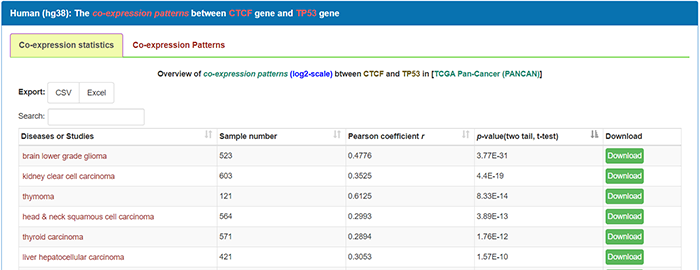
点击后显示CTCF与TP53在各肿瘤中的共表达情况,该页面同之前介绍的共表达页面,此处就不再赘述了。回到前面,点击255,显示结合位点的详细信息:
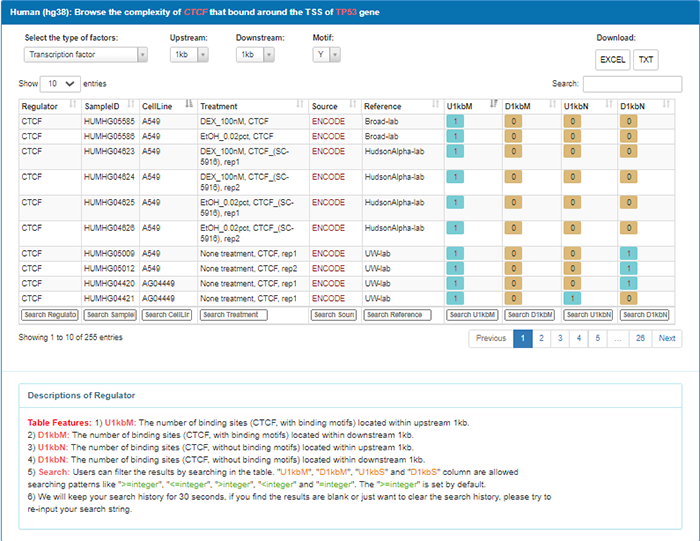
3
已知转录因子,检索其Motif分析结果
下面我们来看下motif版块,该版块可以检索转录因子的motif分析结果。比如搜索CREB1,回车后显示13条结果:
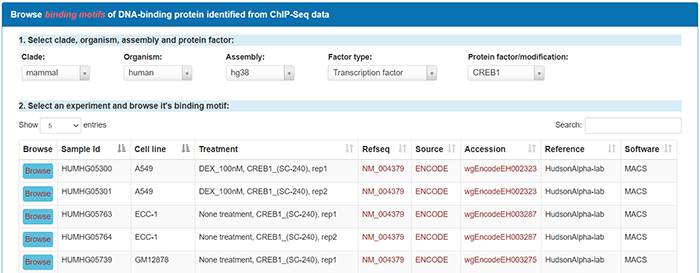
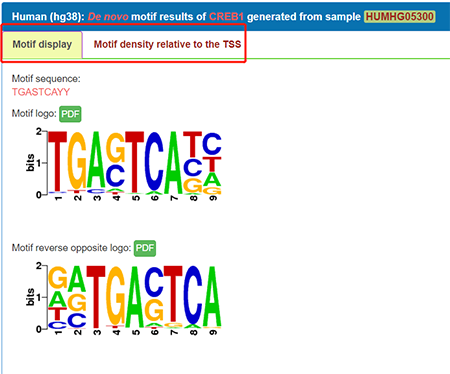
表格依次显示了细胞株、干预信息、Refseq、数据来源、文献支持以及分析软件。点击Browse后进入Motif display菜单,看到motif序列、序列图以及与motif序列反向互补的图,点击PDF可下载:
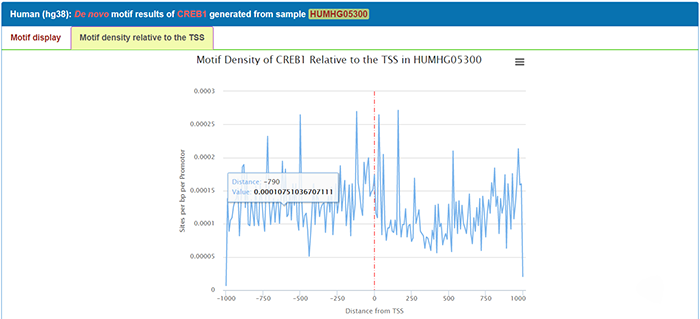
点击“motif density relative to the TSS”显示了在目的细胞中相对于转录起始位点的motif 密度,图中‘0’为转录起始位点:
4
已知转录因子,对靶基因进行富集分析
下面我们看下chip-function模块。该模块对转录因子调控的所有靶基因进行GO富集分析:
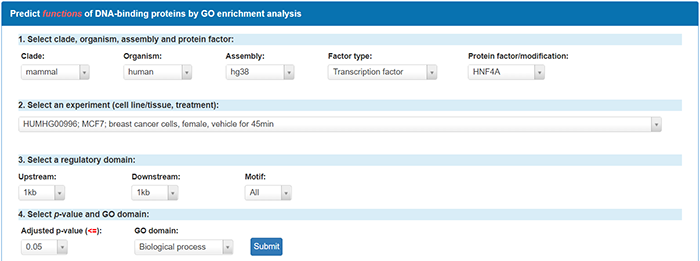
比如输入转录因子HNF4A,可以根据需要选择adjusted p value,GO domain可以选择biological process、molecular function或cellular component。我们以biological process为例,点击submit:
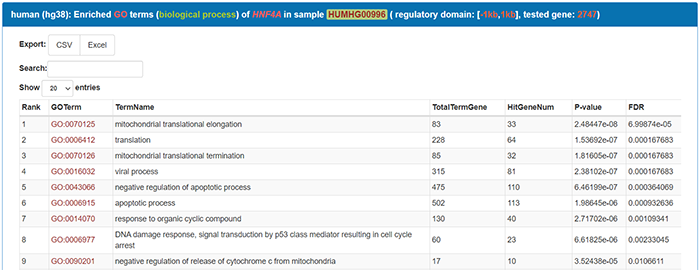
表格列出了HNF4A的靶基因可能参与的39个生物学过程,点击GO term可以查看每个term下的详细信息。molecular function及cellular component操作方式同上。
5
转录因子与靶基因的共表达分析
接下来,我们看一下co-Expression模块。该模块利用从TCGA等公共数据库中整理的约2000个RNA_seq的表达谱数据,分析指定基因间的共表达关系。通过计算pearson相关系数和线性回归来分析两个基因表达量之间的相关性。
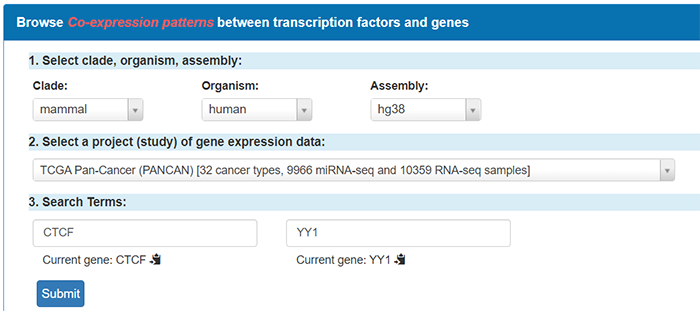
首先还是选择物种,基因组版本等信息,然后选择一个project,我们就选择来自TCGA泛癌分析(TCGA Pan-Cancer)的数据。接着输入需要分析相关性的两个基因,例如CTCF和YY1,点击submit:
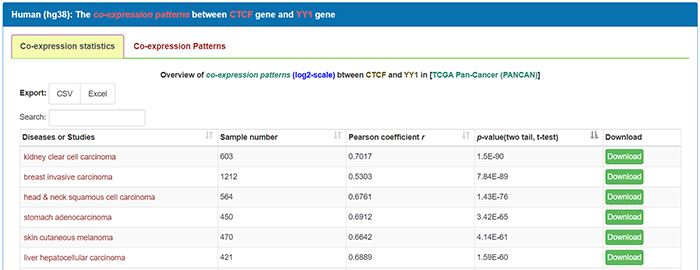
页面刷新后,出现了所有疾病或研究中,两个分子的表达相关性皮尔森系数和p值。点击co-expression patterns是结果的可视化呈现,前面介绍过,这里不再赘述。
三、文献案例实战
接下来我们以一篇文献来说明ChIPBase数据库的使用。文章题目为CDX2/mir-145-5p/SENP1 Pathways Affect LNCaP Cells Invasion and Migration,于2019年6月发表在Front Oncol杂志上,影响因子为4.848分:

文章中作者使用ChIPBase数据库来预测转录因子CDX2与miR-145-5p上游启动子区域的结合位点:

我们一起来看一下怎么操作。进入ChIPBase数据库,点击“miRNA”,转录因子选择CDX2,细胞系选择HUMHG01523,区域选择TSS上游30kb到下游10kb:
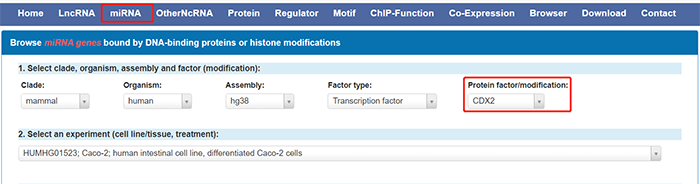
点击“Submit”,页面刷新后下拉到最后,对结果进行筛选:
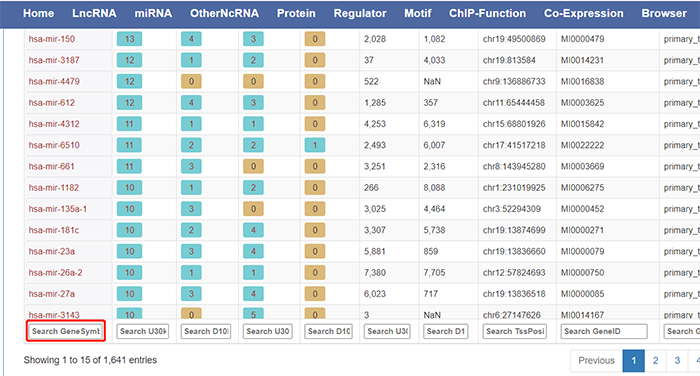
在“Search Gene Symbol”处输入“mir-145”,出现以下结果:
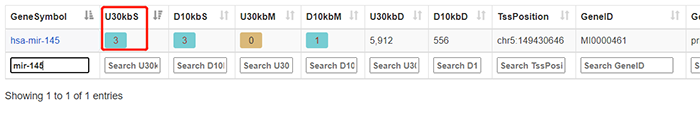
点击“3”:
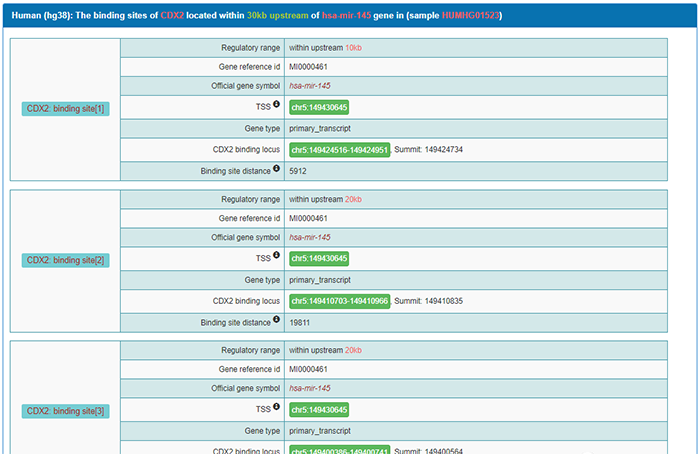
即可显示CDX2在上游区域的三个结合位点的详细信息。
好了,ChIPBase数据库的内容我们就介绍到这里了~ChIPBase数据库与Encori数据库都是由中大开发的神器,从操作页面到主要功能都有相似之处,希望大家可以好好掌握哦~














