目前文献报道的circRNA作用机制包括ceRNA,circRNA与蛋白结合,circRNA编码小分子多肽等,主要目光聚焦于前两个作用机制中。ceRNA涉及到circRNA与miRNA的结合,之前提到的circbank就可以预测circRNA上潜在的miRNA靶向结合序列,而今天介绍的主角circinteractome数据库则更进一步,除了可以预测circRNA的靶miRNA,同时可以预测某一特定circRNA可能结合的蛋白质有哪些,可以说集circRNA机制研究之大成者。
circinteratome数据库在一众研究circRNA的高分文章中举足轻重,运用得当可以成为大杀四方的利器,希望大家熟练运用~circinteractome数据库的网址为https://circinteractome.nia.nih.gov/ 。值得一提的是,circinteractome数据库的信息更新及收集与其他数据库相比稍显滞后,因此小伙伴们在查询circRNA相关信息的时候注意多个数据库交叉比对进行检索。
在使用circinteractome数据库时记得引用它的参考文献哟~
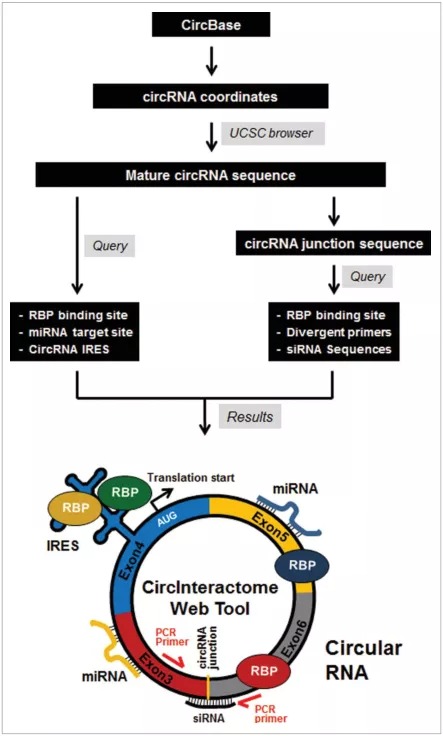
(circinteractome数据库数据处理流程图解)
接下来逐个介绍circinteractome数据库的几个重要板块:
一、circRNA与蛋白质的交互预测
输入网址https://circinteractome.nia.nih.gov/,进入circinteractome数据库主页面。在左侧导航栏依次可以看到
“Circular RNA”(用于查询circRNA基本信息以及可以互作的蛋白质), “RBP on CircRNA”(同样用于查询circRNA与蛋白的互作关系), “miRNA Target Sites”(查询circRNA与miRNA的互作关系), “Divergent Primers”(设计circRNA引物), “siRNA Design”(设计circRNA的siRNA)等
点击“Circular RNA”进入下一个界面。

页面刷新后,出现如下界面。在Step1中可以通过输入circRNA的ID,或者其host gene的gene symbol来进行检索。注意前者需要输入circRNA的circbase ID(circbase ID与circbank ID的区别请见之前的推文),而后者输入host gene的gene symbol出来的结果是某一个host gene序列可能编码出来的所有circRNA。Cell line/Tissue可以选择不同的细胞系。这里以hsa_circ_0000020为例,点击Step2下方的“circRNA Search”。
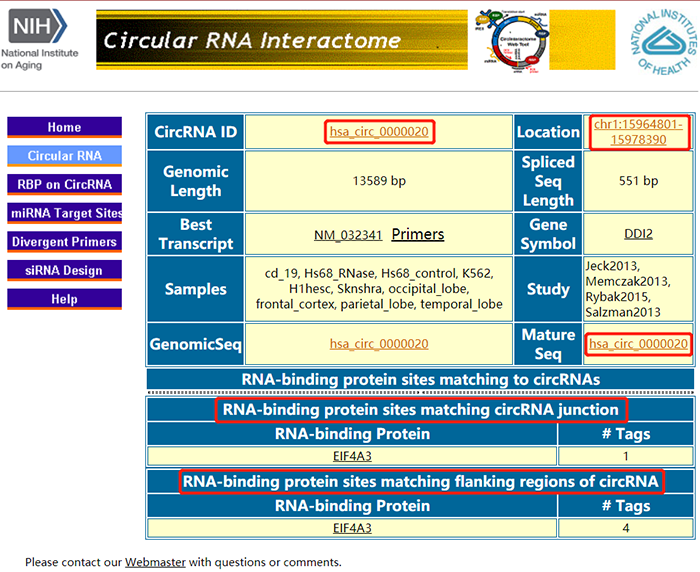
页面刷新后可以看到hsa_circ_0000020的具体信息。
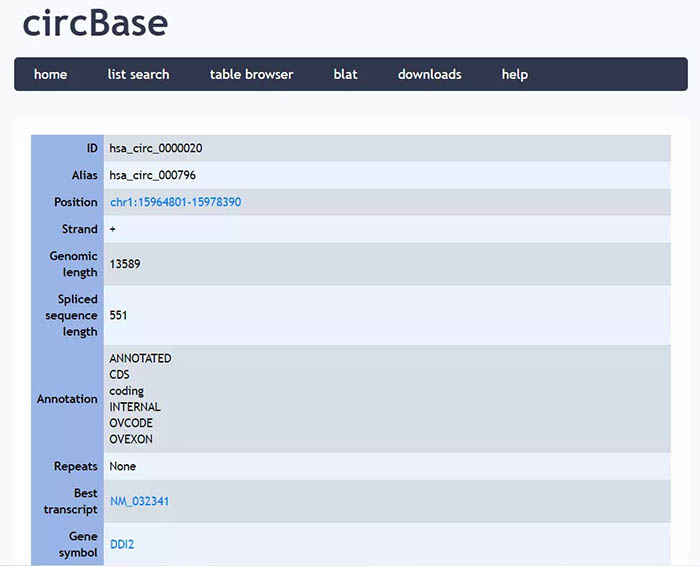
点击CircRNA ID旁的链接可以进入circbase数据库页面,展示了hsa_circ_0000020的染色体位置,长度等信息。
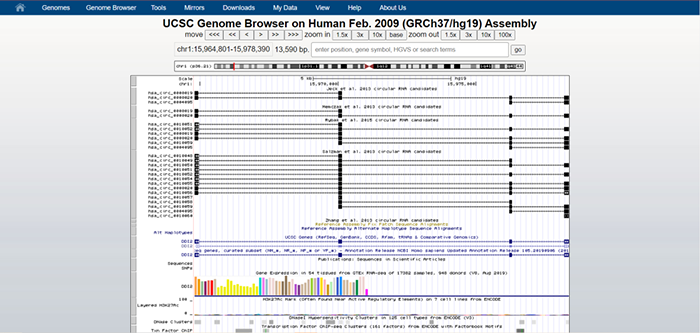
点击Location旁的链接进入USCS界面:
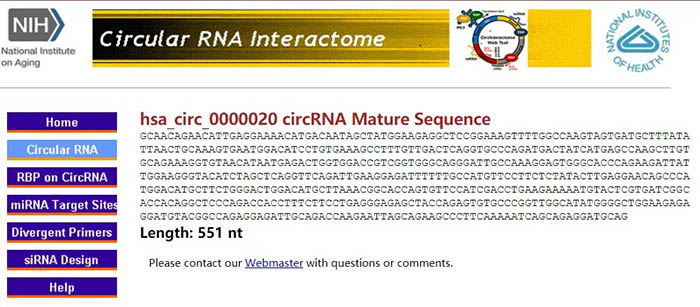
点击MatureSeq旁的链接可以查看该circRNA的序列:
下方的RNA-binding protein sites显示了可以与circRNA结合的蛋白质。注意这里的蛋白质分为结合circRNA反向剪切位点的蛋白质以及结合circRNA侧翼序列的蛋白质。前者可能参与circRNA的剪切、加工、折叠、稳定和定位,后者则可能参与细胞中一系列病理生理反应以及各种表型的发生。从图中可以看出EIF4A3蛋白可以同时结合在hsa_circ_0000020的侧翼序列以及反向剪切位点。注意并不是所有circRNA的RBP蛋白都可以结合在反向剪切位点,使用的时候注意区分。
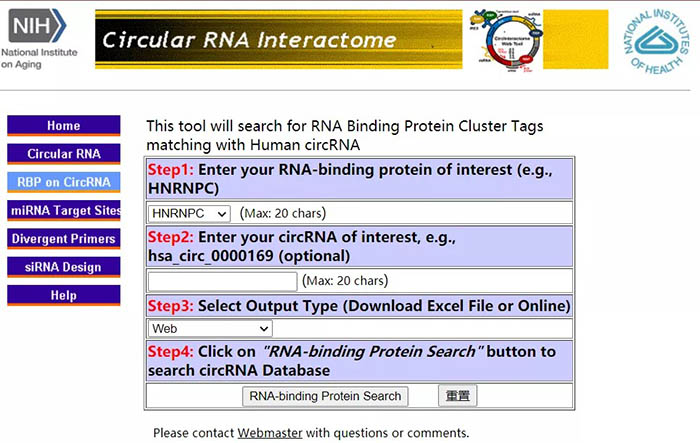
回到主页面,点击“RBP on CircRNA“,同样可以查询circRNA与蛋白的互作关系。以HNRNPC蛋白为例,在Step1中选择HNRNPC蛋白后,点击“RNA-binding Protein Search”。
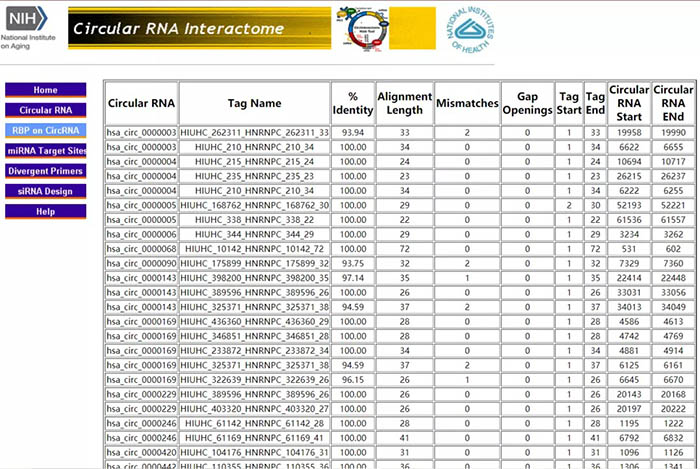
刷新后的页面展示的则是所有可以与HNRNPC蛋白结合的circRNA。
二、circRNA与miRNA的交互预测
点击左侧导航栏“miRNA Target Sites”进入circRNA-miRNA交互预测页面。注意这里的交互预测采用了Targetscan数据库算法,且具有双向性,即在Step1输入circRNA的ID可以预测与之结合的miRNA,在Step2输入miRNA的ID可以预测与之结合的circRNA,根据使用者的目的选择其一即可。这里以hsa_circ_0000020为例,在Step1中输入该分子后点击“miRNA Target Search”。
页面刷新后展示了所有与hsa_circ_0000020潜在结合的miRNA,并提供了两者的结合序列。再次提醒大家,circinteractome数据库的信息并不全面,使用的时候需要大家综合考虑。
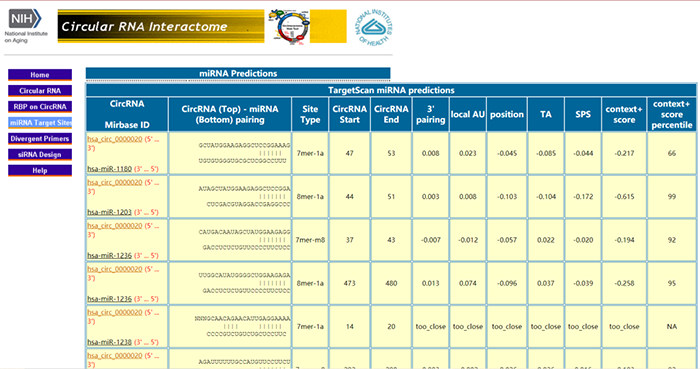
点击相应的miRNA ID进入miRBase数据库查询相关信息:
三、circRNA引物设计
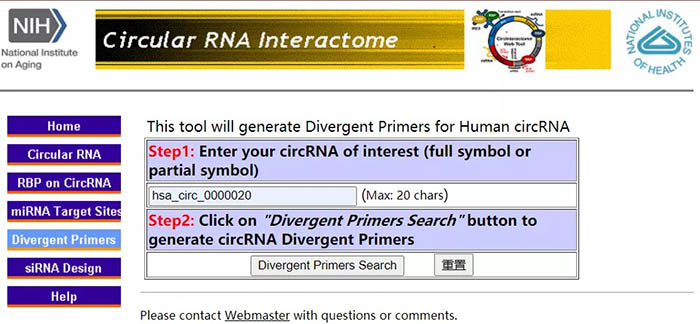
新手研究circRNA碰到的首要问题就是引物设计了。由于circRNA的5‘端与3’端反向成环,存在共价结合,因此circRNA的特异性引物应该能够扩增出包含circRNA反向剪切位点的一段序列,称为divergent primer,即背向引物。点击左侧导航栏的“Divergent Primers”,在Step1中输入需要设计引物的circRNA(以hsa_circ_0000020为例),点击“Divergent Primers Search”。
页面刷新后如图,circinteractome提供两种引物设计途径:Primer 3软件以及NCBI。点击Primer3。
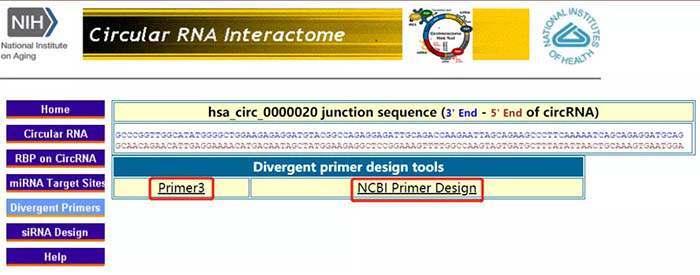
页面自动跳转到Primer3分析界面,可以看到circinteractome已经自动将hsa_circ_0000020的3‘端序列移至了5’端头部重新进行了排列。点击“Pick Primers”。
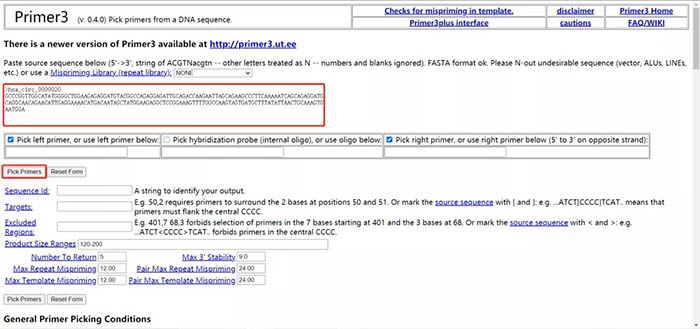
页面刷新后可以看到网站自动生成的hsa_circ_0000020引物有哪些,使用者可以挑选引物去NCBI进一步Blast。
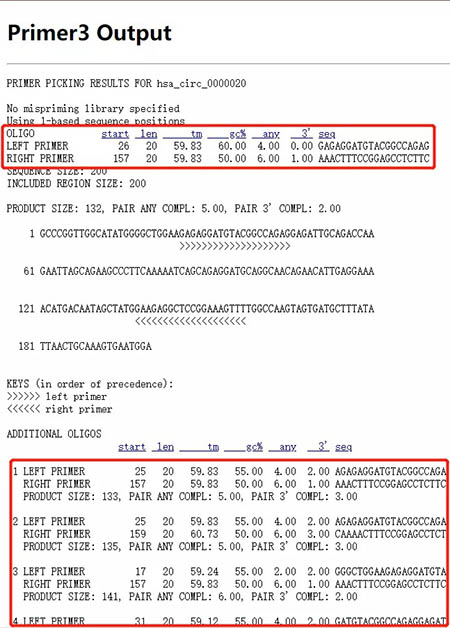
返回刚才的界面,点击“NCBI Primer Design”,页面下拉点击“Get Primers”同样可以得到hsa_circ_0000020的引物序列,此处不再赘述。
四、circRNA的siRNA设计
点击左侧导航栏“siRNA Design”,输入hsa_circ_0000020,点击“siRNA Search”。
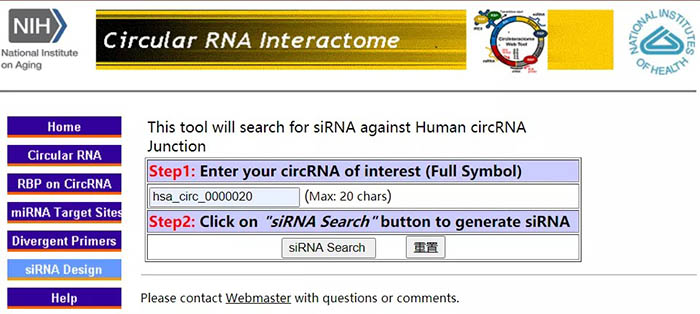
页面刷新后显示hsa_circ_0000020上可以设计的与siRNA结合的序列信息。

点击序列,可以链接至NCBI官网进行Blast。
好了,circinteractome数据库的主要功能我们就介绍到这里了,可以说它一手包办了circRNA研究的方方面面,从引物设计到机制探究到RNAi设计,全方位贴心服务。
最后我们再来总结一下circinteractome数据库的功能:
1. 检索circRNA名称 2. 检索circRNA的基因组位置以及相匹配的转录本信息 3. 检索成熟的circRNA序列 4. 检索与circRNA上游/下游序列结合的RBP 5. 检索与circRNA反向剪切位点结合的RBP 6. 预测靶向circRNA的miRNA 7. 设计circRNA的引物 8. 设计针对circRNA的siRNA














