RNA测序(RNA-Seq)技术是近年来发展和应用迅速的二代测序技术,它促进了基因研究,并被应用于多种癌症研究,为研究选择性剪接和量化基因/亚型表达水平提供了革命性的工具。
基于RNA-Seq的结果我们可以挖掘很多的信息,RNA-Seq-Nexus(CRN)数据库就能帮我们在线实现一些数据挖掘。
CRN数据库是第一个提供肿瘤细胞表型特异性编码转录物/lncRNA表达谱和lncRNA调控网络的公共数据库,网站链接为http://syslab4.nchu.edu.tw/CRN。

该数据库由中国台湾National Chung Hsing University國立中興大學的学者开发,相关文章(PMID: 26602695)于2016年初发表在Nucleic Acids Research杂志(2019IF=11.501)上,截至2021-01-19该文章已引用48次(数据来源:PubMed)。

在CRN数据库中,开发者们系统地收集了来自癌症基因组图谱(TCGA)、序列读取档案(SRA)和NCBI基因表达综合数据库(GEO)的RNA-Seq数据集。
该数据库囊括多种肿瘤RNA-Seq数据集,包括325个亚群和12167个样本。每个数据集都有几个特定表型的子集,每个子集包含一组具有特定表型特征或癌症状况(例如疾病状态、细胞系、细胞类型、组织、基因型)的RNA-seq样本。
另外,开发者们还手动创建子集,然后根据数据集和样本的描述将样本分配给子集。因为每个子集包含一组具有特定表型或基因型的RNA-seq样本,例如乳腺癌II期、ER+乳腺癌和Her2+乳腺癌。因此,CRN数据库可以为个性化医疗提供方便。
为了确定每个数据集中的表型特异性差异表达转录物(differentially expressed transcripts,DETs),开发者们选择了至少有3个样本的亚群,然后在两个没有重叠样本的亚群之间进行t检验。
共产生973个亚组对,包括822个“癌症与癌症”亚组对和151个“癌症与正常”亚组对。开发者们还将RNA-Seq reads与人类转录组(GENCODE release 21)进行比对来获得mRNA和lncRNA的表达谱数据。
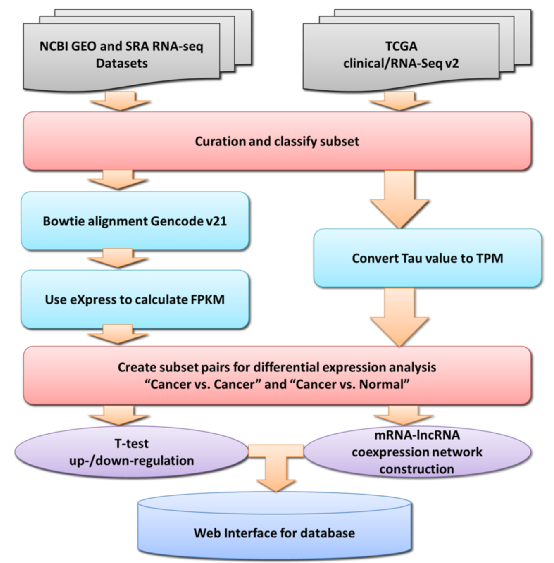
上图展示了该数据库的内部运算流程。下面,我们就一起来看一下这个好用的数据库吧!
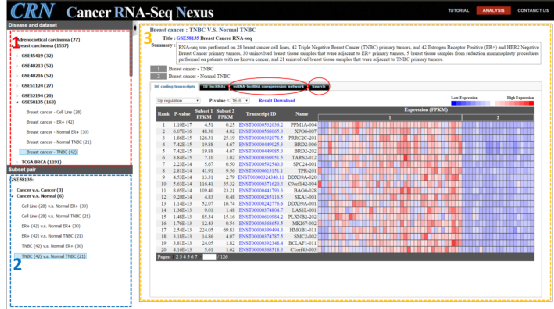
CRN数据库界面提供以下三个主要面板:
(1)疾病数据集面板(左上面板):使用癌症疾病和癌症子集构建层次结构。子集表示与特定表型或基因型相关的一组RNA-seq样本。
(2)子集对面板(左下面板):有两种类型的子集对“癌症与癌症”和“癌症与正常”。当用户单击癌症疾病或癌症子集时,数据库会在“子集对”面板中显示相关的子集对。
(3)表达谱面板(右面板):在子集对面板中单击子集对时,数据库显示数据集和子集的详细描述。
当用户选择子集对时,数据库会显示数据集和子集的详细描述。
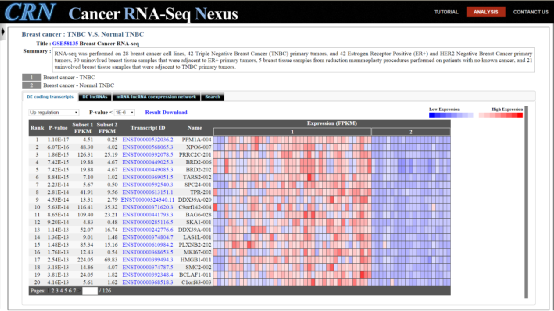
在表达谱页面中,有四个选项卡:
(1)DE coding transcripts:对差异mRNAs进行可视化。
(2)DE lncRNAs:对差异lncRNAs进行可视化。
(3)mRNA-lncRNA共表达网络:在这个标签面板中搜索mRNA和lncRNA时,会对二者之间最显著的负相关和正相关的共表达网络进行可视化。
(4)搜索:用户可以在此面板中搜索基因/转录本名称和转录本ID。
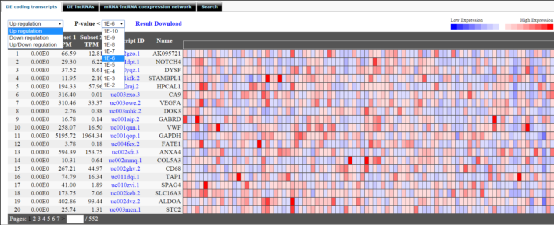
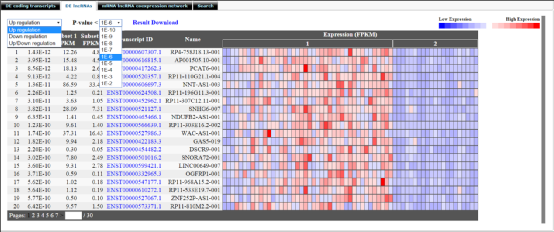
前两个选项卡类似,用户可以对差异的mRNAs和lncRNAs的上/下调和P值进行选择,也可以之间下载结果。
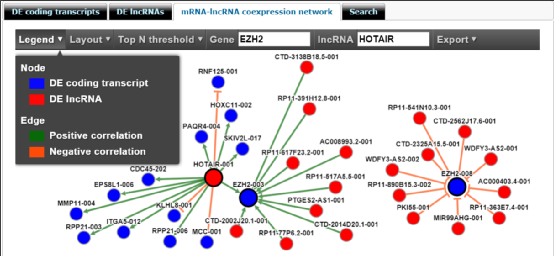
在第三个选项卡(共表达网络),用户可以输入mRNA和lncRNA名称,然后选择相关阈值。要选择mRNA和lncRNA之间的共表达网络,用户可以自定义前N个连接的选项(N=10、15、20或25)。这里也支持共表达网络的直接导出。
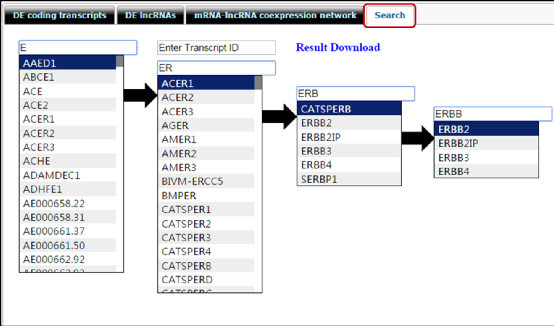
用户只需输入部分基因符号或转录本ID,数据库的自动完成功能提供了快速搜索和选择部分匹配项的功能。此外,通过输入更多字符,它将过滤列表以获得更好的匹配。
自动完成功能不仅可以帮助用户高效搜索,而且可以快速过滤。选择基因或转录本后,选项卡显示给定基因或转录本的表达谱。最后,用户也可以直接下载数据结果。
当然,如果用户对数据库的分析结果不满意,用户也可以直接在网站下载相关数据自行分析和进行可视化。















