今天的主角,circAtlas数据库,综合了我们既往讲过的所有circRNA数据库相关的内容,包括结合miRNA,结合RBP,circRNA编码小肽,疾病相关性,组织特异性,物种多样性,甚至还有ID转换的功能。更独树一帜的是,circAtlas数据库通过构建circRNA—miRNA/RBP/mRNA网络对circRNA的功能进行了GO/KEGG富集。要知道半个月前我才在组会上夸下海口,circRNA目前做不了富集分析,因为研究的比较少,此刻的我只感觉啪啪打脸,却甘之如饴。涵盖了方方面面,如此齐全的数据库,我愿称之为—“circRNA之宇宙的尽头”。
数据库网址为http://circatlas.biols.ac.cn/
使用数据库的时候记得引用参考文献:
Wu W, Ji P, Zhao F. CircAtlas: an integrated resource of one million highly accurate circular RNAs from 1070 vertebrate transcriptomes. Genome Biol. 2020 Apr 28;21(1):101. doi: 10.1186/s13059-020-02018-y. PMID: 32345360; PMCID: PMC7187532.
一、数据库主页介绍
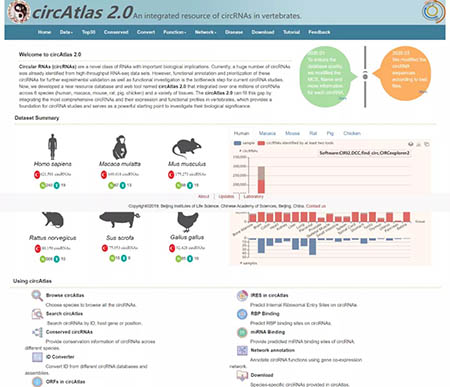
首先我们通过主页信息来简单了解一下这个数据库。输入网址http://circatlas.biols.ac.cn/ ,进入数据库主页面。该数据库由中国科学院北京生命科学研究院赵方庆研究员带领团队于去年4月份构建的,依旧是俺们中国人的杰作,因此可以不用挂梯子速度也很快。
左上角的数据库图标我很喜欢,由人类和猿猴的头像组成了太极八卦图,既包含了中国元素,又暗示了该数据库的一个重要特点----进行了多物种信息的汇总。
下方菜单栏链接至circAtlas数据库的各种功能,包括“Data”(检索某一个circRNA),“Top30”(展示不同物种和组织中表达量最高的30个circRNA),“Conserved”(对circRNA保守性进行分析),“Convert”(circRNA ID转换),“Function”(分析circRNA编码潜能),“Network”(构建circRNA互作网络),“Disease”(分析circRNA与疾病之间的关系),“Download”(下载数据库中的信息),“Tutorial”(数据库使用说明),“Feedback”(用户反馈)。这些功能我们将在之后一一介绍。
往下文字部分介绍了circAtlas数据库基本情况,一句话总结就是,这个优秀的数据库对6种脊椎动物(人类,猕猴,小鼠,大鼠,猪,鸡)的circRNA进行了功能注释。右侧的图片则展示了更新的日志。页面下拉展示了所收集的6个物种circRNA的信息,包括每个物种circRNA的数量,收集的样本数以及组织的数量。点击每个物种图标的超链接可以自动跳转到“Data”功能下的子目录“Browse circAtlas by species”,即通过物种来检索circRNA。
右侧以柱状图的形式来展示所收集的组织及样本数,柱状图右上角标注了circAtlas数据库鉴定circRNA所采用的算法—CIRI2,DCC,find_circ,CIRCexplorer2。看过前几期推文的小伙伴们应该能认出来,这都是各个circRNA数据库的老朋友了。点击右上角下载按钮可以下载柱状图的图片用于文章中。最下方罗列了circAtlas数据库的主要功能。
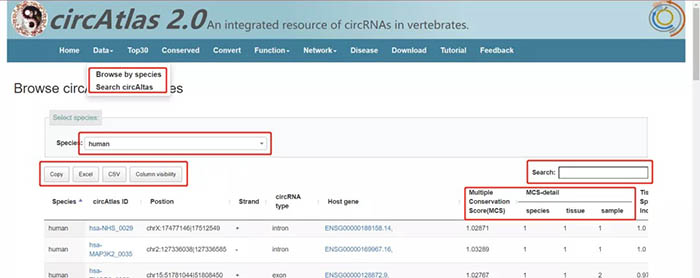
二、“Data”
“Data”主要用于在circAtlas数据库中进行基础检索,其下分为两个子目录:“Browse by species”与“Search circAltas”。点击“Browse by species”,页面跳转到如下界面,显示了circAtlas数据库收集的每个物种的所有circRNA。点击“Species”旁边的对话框可以对物种进行选择。下方还有“Copy”,“Excel”,“CSV”,“Column visibility”等可供使用。点击“Copy”可将当前页面的检索结果复制到粘贴板上,用户可以之后再粘贴到excel或者txt文档中。说到这不得不回过头来吐槽circbank数据库了,让用户自己一页一页地复制粘贴简直是反人类。点击“Excel”可将当前页面结果以excel的形式下载下来。划重点,是当前页面结果,也就是50行的内容,但是整个物种的信息显然不止有50行,所以还是得一页一页地进行下载。“CSV”功能与“Excel”功能相同,只不过文件保存的类型不同。点击“Column visibility”可以选择下方表格显示的内容,隐藏相应的列。下方表格罗列了每个物种的所有circRNA信息,包括物种、circAtlas ID(由circAtlas数据库自行命名)、染色体上的位置信息、正/反义链、circRNA类型、宿主基因的Ensemble ID、MCS评分及细则、组织特异性评分、circRNA长度以及鉴定所采用的算法。小声吐槽一下,表格右上角的检索框试了一下感觉没啥用,如果想进行检索不如直接把结果下下来在excel里面查找还来得快一些~
这里特别说明一下MCS评分是什么。MCS评分,即Multiple Conservation Score,由circAtlas数据库自行创建的评分标准,从物种、组织、个体三个层面来评估circRNA的保守性。具体算法如下图所示:
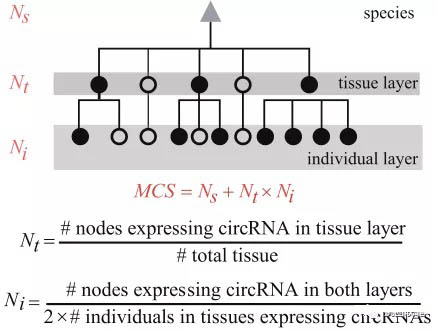
而MCS = Ns+ Nt×Ni。在表格中大家可以看到MCS都是以小数的形式呈现的,其中整数部分,即Ns,反映了circRNA跨物种的保守性,小数部分,即Nt×Ni,反映了circRNA跨组织及个体的保守性。MCS评分越高,说明circRNA保守性越好。
点击“Search circAtlas”,页面跳转到如下页面。在检索框中可以输入具体的circRNA信息进行检索,包括circAtlas ID,其他类型的circRNA ID(如circbase ID,芯片ID,CIRCpedia数据库ID等),宿主基因Ensemble ID,以及染色体位置。下方可以选择物种。我们以示例“has-ABCC2_0015”为例,点击“Run”。
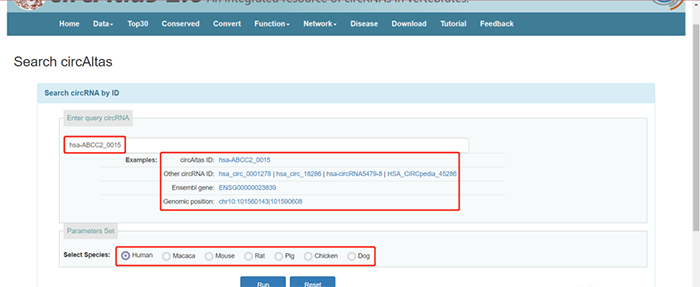
检索结果如下图所示,具体参数如上所述,此处不再赘述。
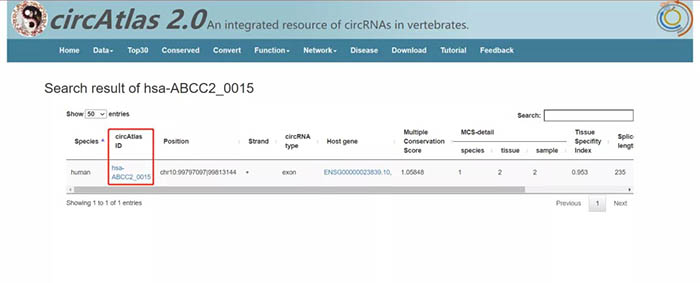
点击“circAltas ID”处的超链接,页面跳转到如下:
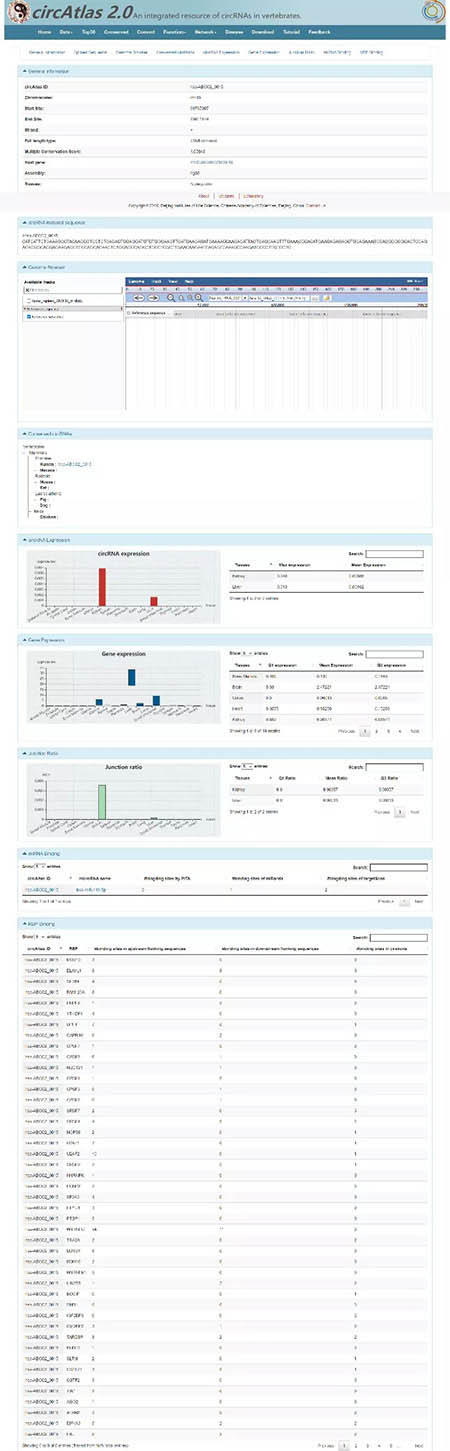
好家伙,出来了长长的一份检索报告,包括circRNA基本信息(如染色体位置,正/反义链,全长,MCS评分,宿主基因Ensemble ID,参考基因组版本,检测的组织及样本信息)、circRNA序列、基因组浏览器界面、circRNA保守性、circRNA在器官组织中的表达、宿主基因在器官组织中的表达、反向剪切位点的表达、预测的所能结合的miRNA以及RBP。是不是看的眼花缭乱?简直一波带走circRNA研究的绝大部分需要使用的信息。这里没有展示ORF和IRES,在circAtlas数据库其他地方可以查询。现在大家是不是对这个数据库的全面性和综合性有了更深的认识呢?
三、“Top30”
点击“Top30”,跳转到如下页面:
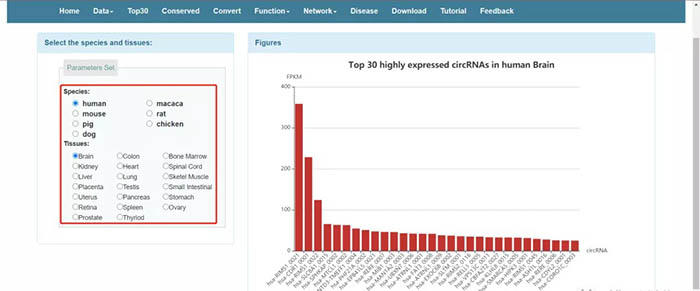
在左侧参数设置处勾选不同的物种和不同的组织可以查看前30个表达显著增高的circRNA,可供大家在设计课题的时候参考选择主变量分子。
四、“Conserved”
点击“Conserved”,跳转到如下页面。一共有两种方式可供使用,“Search Conserved circRNA groups”&“Analyze Conserved circRNAs”。
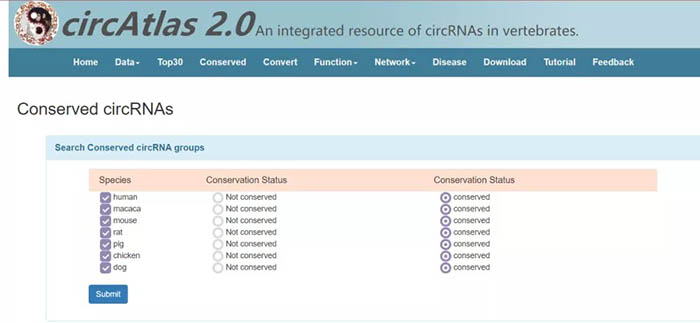
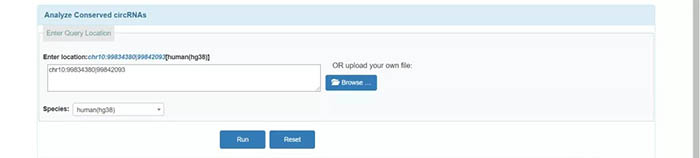
我们重点讲解第二种“Analyze Conserved circRNAs”。用户可以通过在对话框中输入自己的circRNA染色体位置信息或者上传文件进行保守性分析,可以输入一个分子,也可以输入一个list。我们以“chr10:99834380|99842093”为例,在“Species”旁选择物种“human”后,点击“Run”:
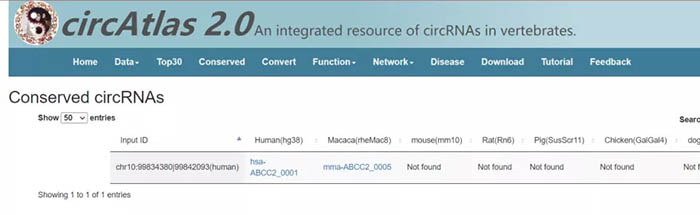
结果显示,该circRNA在人类和猕猴体内均有表达,在两个物种间具有保守性。
五、“Convert”
点击“Convert”,跳转到如下页面。一共有两种方式可供使用,“Search circRNA”&“Analyze circRNA”。我们重点讲解第一种“Search circRNA”。在“Search circRNA”版块中,我们以“hsa_circ_0064316”为例(circbas ID),输入后点击“Submit”:
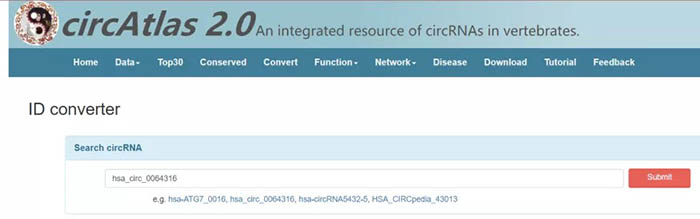
跳转到如下界面。可以发现circAtlas数据库提供了该circRNA相应的circAtlas ID,deepbase2 ID以及circpedia2 ID。换言之,circAtlas数据库可以在这几种ID之间进行转换。
六、“Function”
鼠标悬停在“Function”上可以看到这一部分主要检索的是ORF与IRES。我们以ORF为例,IRES的操作与ORF相同。点击“ORF”,跳转到如下页面。在“Search circRNA”版块中以 “hsa-ATG7_0015”为例输入,点击“Submit”。
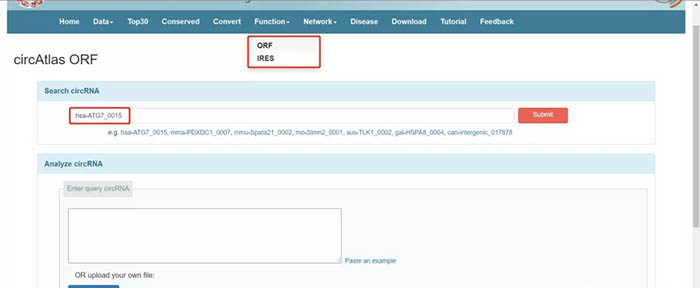
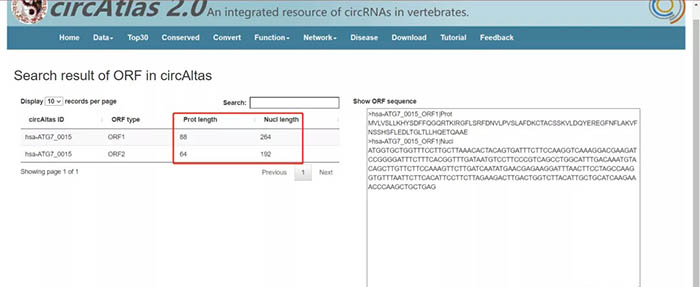
页面刷新后显示了hsa-ATG7_0015的ORF信息。点击每一行即可在右侧方框中显示该ORF的核酸和蛋白序列。
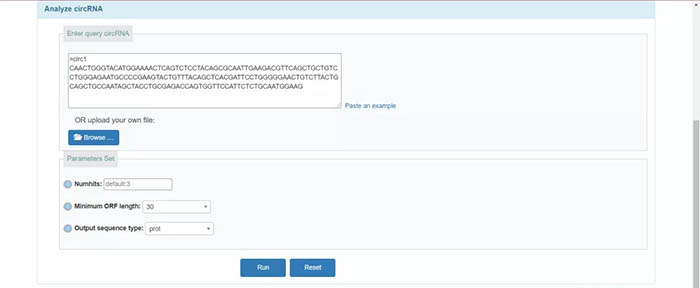
用户同样可以在“Analyze circRNA”中检索ORF。需要注意的是,这里输入的应该是circRNA的FASTA格式,包括:标头行(以“>”开头),序列行,如下图示例所示。用户同时可以设置参数,包括“Minimum ORF length”(最短ORF长度,默认为30),“Output sequence type”(输出的序列类型)。点击“Run”:
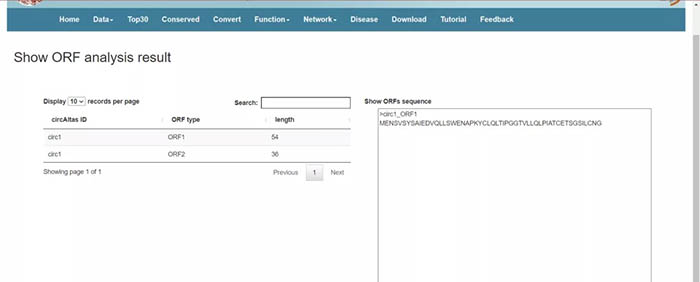
页面刷新后如下图所示,结果展示同上,不再赘述。
七、“Network”
鼠标悬停在“Network”上可以看到其下显示子目录“RBP”,“miRNA”,“Annotation”,提示该模块用于预测circRNA互作的蛋白,miRNA,并构建网络图。首先点击“RBP”,页面跳转如下:
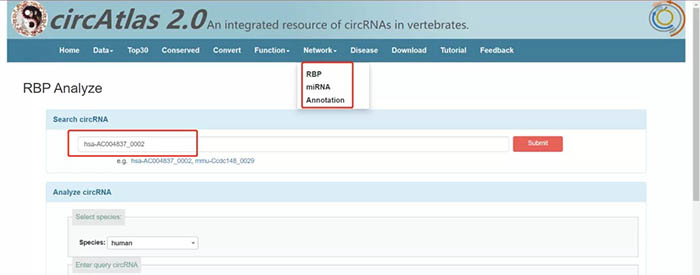
在“Search circRNA”中,用户可以通过输入circRNA ID进行检索。以hsa-AC004837_0002为例,点击“Submit”:
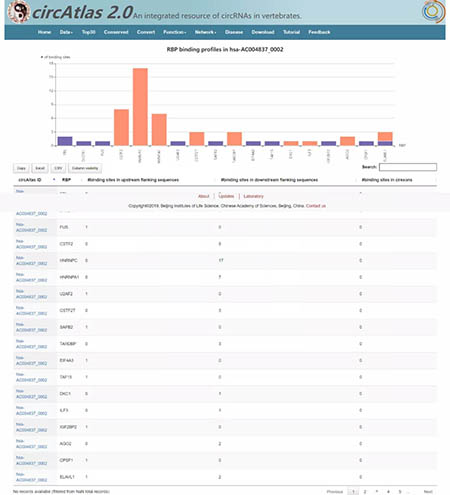
结果页面以柱状图及表格的形式展现了hsa-AC004837_0002可以结合的蛋白有哪些,同时标注了在上游侧翼序列、下游侧翼序列以及外显子区域部分结合的蛋白位点数量,其可视化的程度及信息数量比circInteractome数据库更胜一筹。
用户同样可以在“Analyze circRNA”版块通过输入circRNA的FASTA格式来进行检索。选择物种,输入FASTA格式序列/文件,选择反向剪接位点上下游结合的circRNA片段长度以及结合的蛋白种类,点击“Run”,即可出结果。结果页面解读同上及之前讲过的数据库,此处不再赘述。
点击“miRNA”,其操作流程及结果页面同RBP,不过多解释。需要注意的是,circAtlas数据库预测circRNA与miRNA结合的时候使用的数据库为Targetscan,miRanda以及Pita。
点击“Annotation”,即可看到circAtlas数据库对于circRNA构建的网络图和注释内容。在“Search circRNA”中以“hsa-ING1_0005”为例,点击“Submit”:

页面刷新后如下:
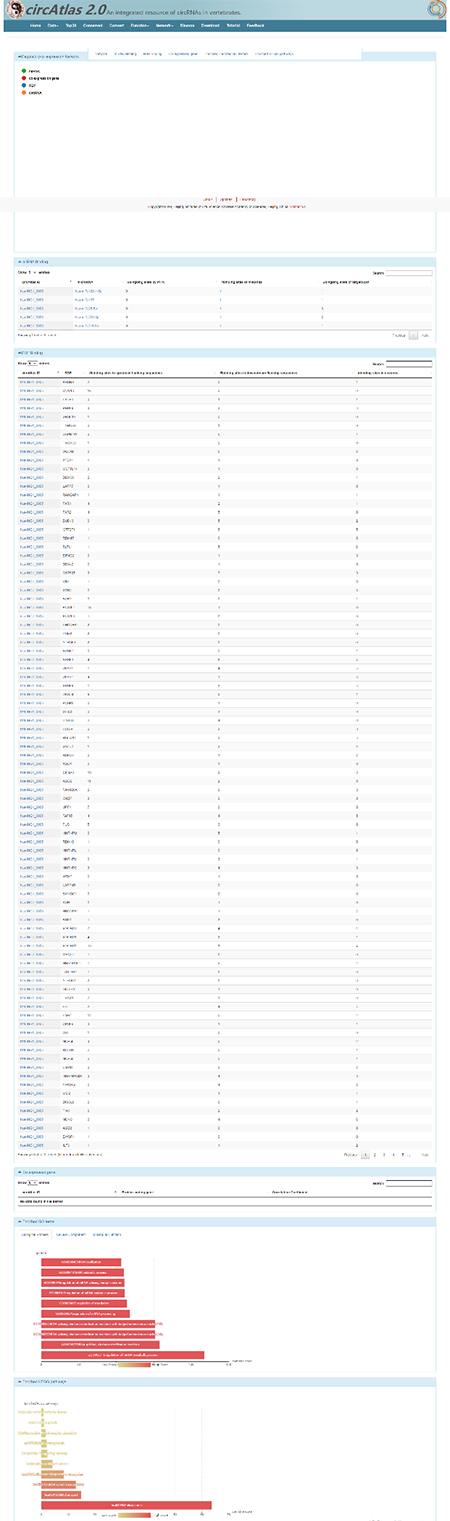
circAtlas数据库展示了该circRNA所能结合的miRNA及RBP信息,以及富集分析的结果,如GO/KEGG。
下方“Analyze circRNA”中可以输入自己的数据文件进行网络图绘制以及GO/KEGG分析,我们试试上传示例数据的matrix data来看看会有什么样的结果。示例数据可以通过点击“example”的超链接进行下载。数据上传后,点击“Run”:
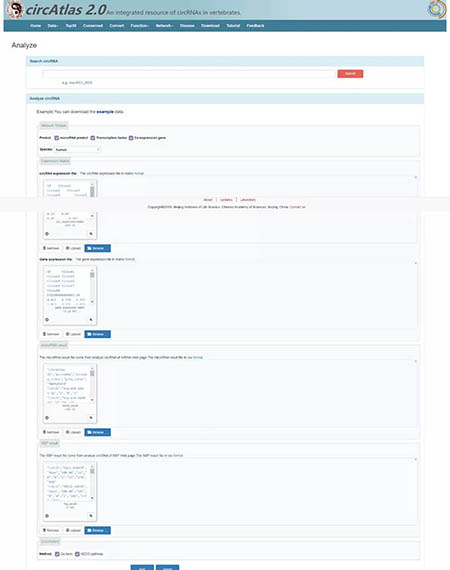
页面刷新后如下:
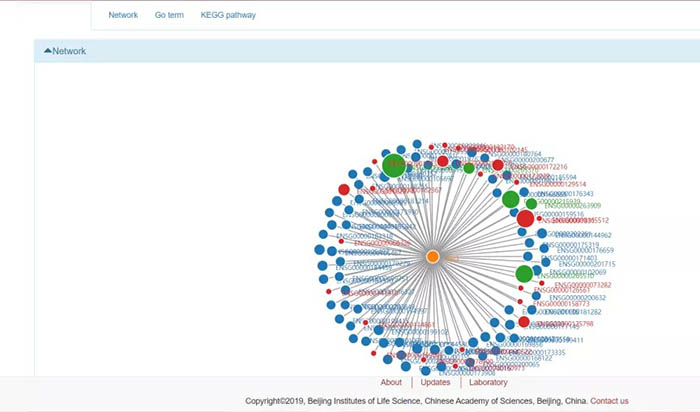
我们来说一下这个网络图和GO/KEGG分析是咋完成的。在数据上传时一共会上传4个文件:第一个是circRNA在每一个组织中的表达谱数据,第二个是基因在每一个组织中的表达谱数据,第三个是预测的circRNA与miRNA结合的文件,第四个是预测的circRNA与RBP结合的文件。circAtlas数据库通过分析circRNA与基因之间的相关性将其联系起来,再结合与该circRNA相关的所有基因、miRNA与蛋白的功能来推测circRNA的功能,并进行富集分析。换言之,富集实际上富的也是circRNA互作或者相关的一些分子,来侧面反应circRNA的功能。
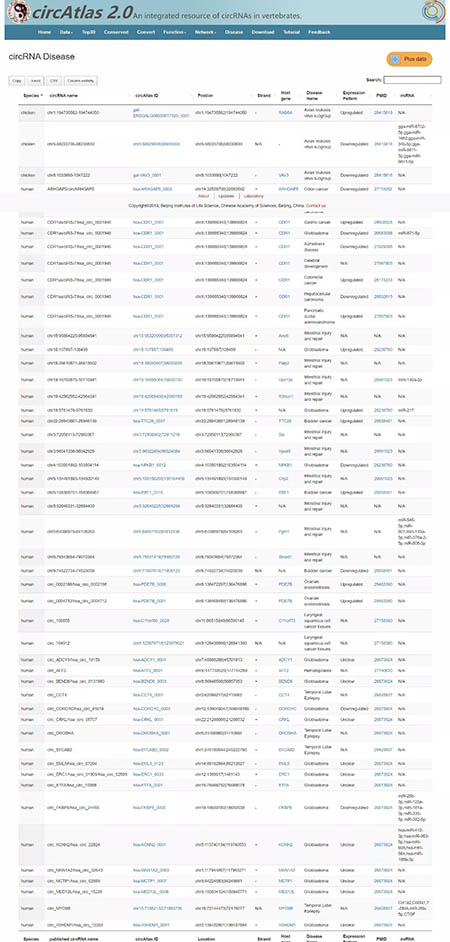
八、“Disease”
点击“Disease”可以查看circAtlas数据库收集的circRNA—疾病关系对,结果同之前介绍的circR2disease数据库等。在右上角的“Search”检索框中可以输入相应的染色体位置信息或者宿主基因名称等进行检索。“Plus data”可以使用户上传自己的数据。
circAtlas数据库的功能实在是太丰富了,涵盖了circRNA研究的方方面面,咱们今天的介绍到此为止,其实可以看到很多内容还是和之前介绍的一些数据库有异曲同工之妙,circAtlas数据库胜在广,但是深度可能比相关专业的数据库要差一丢丢。然鹅,一波带走所有问题,不香嘛?














