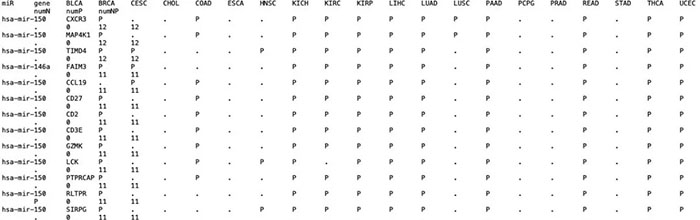
miRNA作为ncRNA的一类。其主要发挥功能的主要方式是通过和基因形成双向互补链进而来影响基因的表达。因此通过miRNA和基因序列是否互补就可以遇到miRNA可能的靶基因了。这个基本上就是经典的预测miRNA靶基因的原理了。类似的很多数据库例如targetscan, miRWalk都是基于这个原理来构建的。
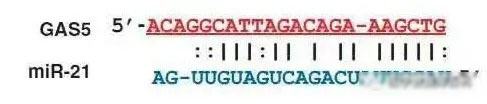
但是在不同疾病当中,miRNA和基因之间是否存在相互作用关系。也不能只是通过序列来判断。万一一个miRNA在这个疾病当中根本不表达,那也就不存在是否发挥作用了。随着高通量测序技术的发展。我们可以通过相同患者的miRNA-seq以及RNA-seq的相关分析,来进一步判断这miRNA和基因是否真的存在相互作用关系。对于相同患者进行miRNA和RNA测序的数据的话。目前只有肿瘤当中的TCGA数据库比较全了。因此,就有人用TCGA当中的miRNA和RNA-seq的数据来构建了一个通过测序数据预测多肿瘤miRNA靶点的数据库:miRactDB(https://ccsm.uth.edu/miRactDB/index.html)

背景数据库
由于是通过高通量测序技术来进行相关预测。所以选择的数据集是一定要是每一个样本都做了miRNA-seq以及RNA-seq的数据集。因此只有TCGA和GTEx这类的大型测序项目包括这些。同时这个数据库也包括了CCLE这种细胞系检测的数据
数据库使用
数据库的使用比较简单。我们只需要输入想要预测的miRNA以及想要预测的基因即可。如果只有其中一个预测目标。输入其中一个即可。例如,我们想要预测"CTLA4"所有具有相关的miRNA.
PS: 由于Mirna-Seq检测的Mirna前体。所以我们在输入的时候,需要输入的是前题,即没有5P/3P这样的Mirna。

结果解读
在点击搜索之后,就可以获得一个和CTLA4和所有miRNA在不同肿瘤当中相关性的性的结果。
PS: 作者把相关分析当中的Q < 0.05 且R > 0.1作为具有相关性。相关分析作用使用的是Spearman。
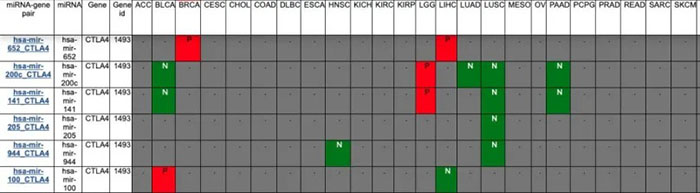
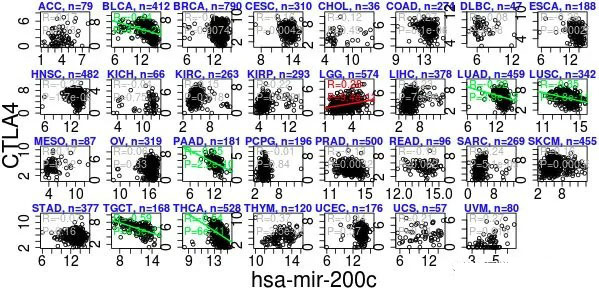
对于具体的一个组合。我们可以点击前面的链接查看具体的结果。在具体的结果当中,我们可以看到关于这个miRNA的总结、基因的总结以及具体的表达和相关性。同时还有其预后和用药等等信息。

例如,在其具体的相关分析当中,我们就可以看到这样的相关散点图
数据下载
以上就是这个数据的主要使用方法了。同时这个数据库提供了所有数据下载的链接。我们可以下载数据库当中的所有数据。可惜的是,在miRNA-gene相关分析的数据中。作者把具体的相关系数换成了。P/N。这样我们就不知道具体的结果了。
虽然说,结果分析起来不难,但是能偷懒还是可以偷一下懒