总的来说,上传数据分为三个步骤。如下:
①创建BioProject(用于描述项目的基本信息,里面可包含多个BioSample。BioProject 号以 PRJNA 开头,如 PRJNA*****)
②创建BioSample(主要是描述样本的基本属性,比如采样地点、采样时间、经纬度等。通常 BioSample 号以SAMN 开头,如 SAMN*****)
③上传数据
下面就是每步详细的sao操作
1 BioProject的创建
1.1 访问下面链接,New submission
https://submit.ncbi.nlm.nih.gov/subs/bioproject/
1.2 填写Submitter
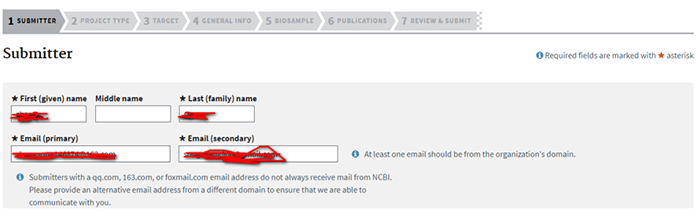
如实填写信息就行了
1.3 Project Type填写
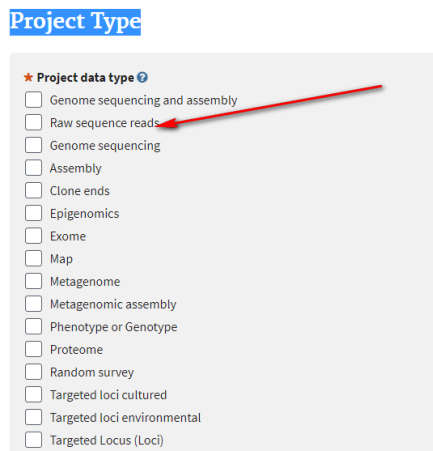
一般高通量测序数据可选择“Raw sequence reads”
根据自己的样本,选择Sample scope(通常来说,Sample scope是对实验物种的简洁描述或者说是一个题目)

1.4 Target填写
给自己的项目取个名字
1.5 General Info信息填写
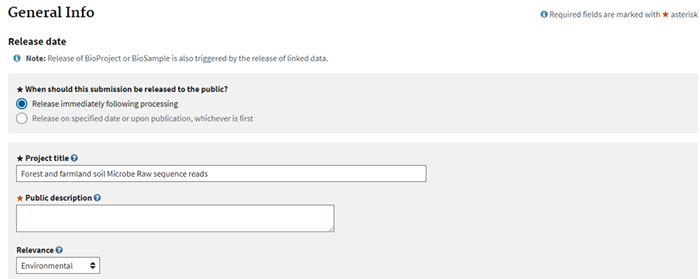
Release date:选择数据释放的时间
Project title :根据上一步的Target填写一个项目名称
下面就是否要选择关联其他数据,No就不用填写,Yes就必须要填写
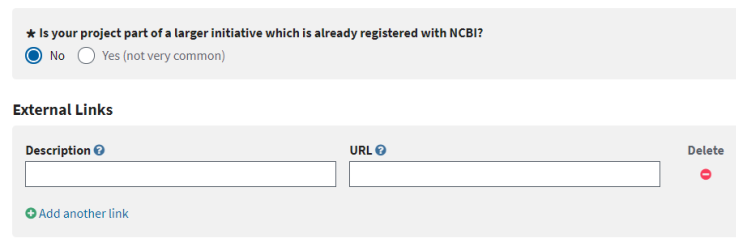
1.6 Biosample
这一步可以直接Continue,等创建好了BioProject再创建BioSample
1.7 Publications
填写PubMed ID 或DOI 号
这一步可以直接Continue
注:BioSample 和Publications 这两步可以省略,后期发邮件给NCBI 进行修改。
1.8 确认无误后,点击“Submit”按钮,创建该Project。经过以上步骤,经过批准会发送到邮箱里面,获得以 PRJNA 开头的BioProject ID。
2 BioSample的创建
访问下面的链接,New submission
https://submit.ncbi.nlm.nih.gov/subs/biosample/
2.1 Submitter的填写会自动填充
2.2 General Information
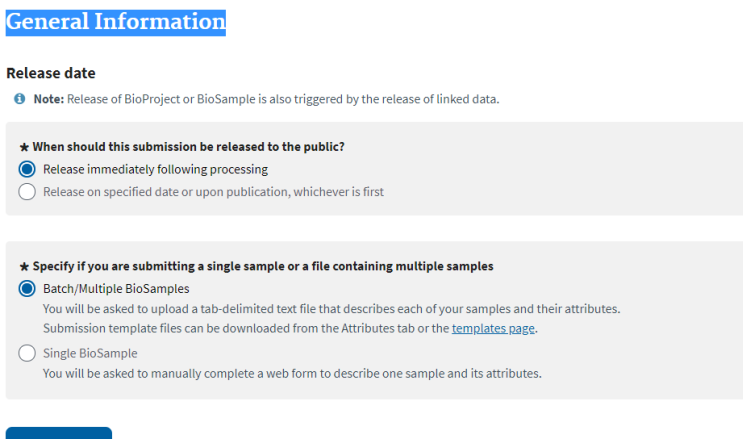
根据自己的需求选择释放样本的时间,这里选择的是立即释放。第二个是选择多样本还是单样本上传。
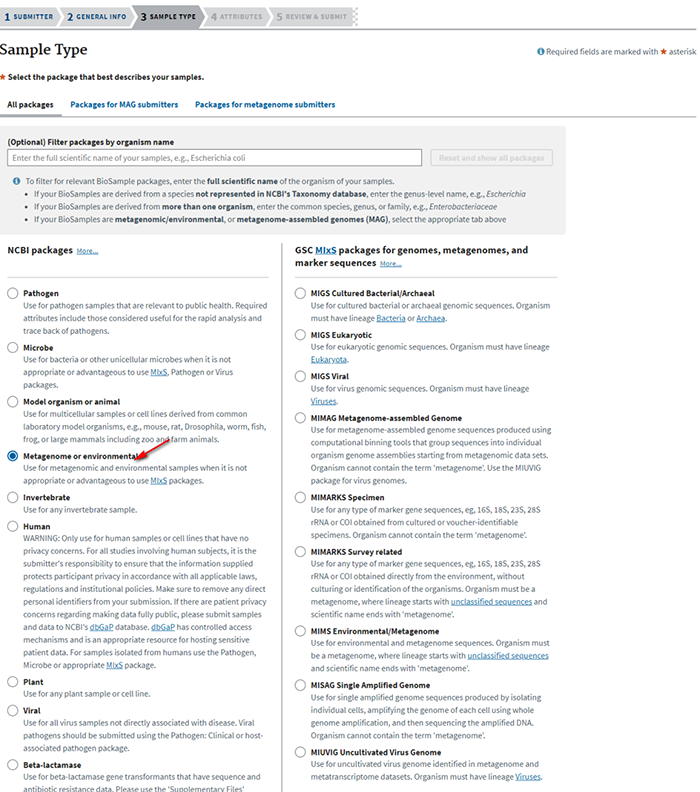
2.3 Sample Type
根据自己的项目进行选择,不过一般扩增子数据都选择Metagenome or environmental
2.4 Attributes
填写样本属性信息
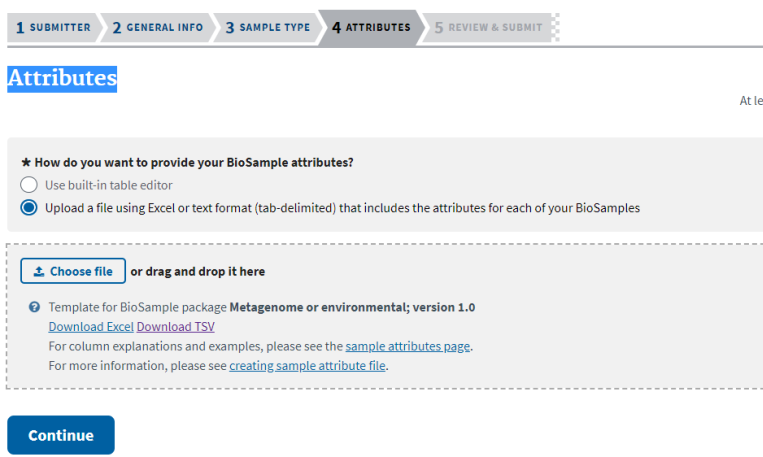
可以选择上传excel表格,表格信息填写如下
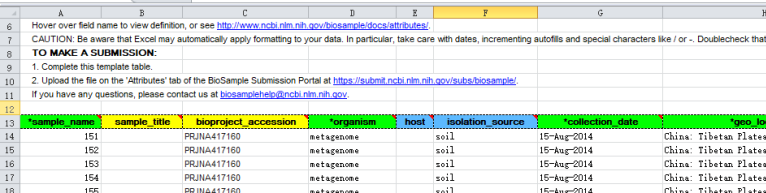
每列信息说明:
sample_name:样品名;
sample_title:每个处理可以写一个题目,可选;
description:处理的描述,可选;
organism:优势物种名,如:metagenome
collection_date:采样时间,如:2014-08-15和15-Aug-2014;
geo_loc_name:采样地,如:China:Beijing;
lat_lon:经纬度,如:39 N 116 E;
isolation_source:分离环境,如:Rhizosphere soil;
注:表格中的绿色是必填项,一定要保证至少有一列可以区分各个样本(名字除外)
表格提交后,一定要保证没有任何warnings,不然即使提交上去也不会通过,如果爆出“Warn”信息,则需要等待2 个工作日方能重新进行该步骤。
2.5 确认无误后,点击最后的“Submit”按钮。经过以上步骤,邮箱会收到以 SAMN 开头的BioSample ID。
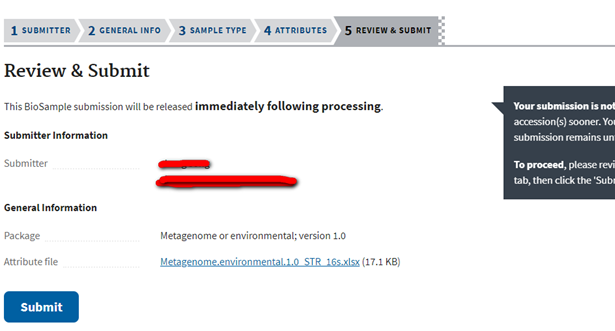
3 上传SRA数据
访问下列链接
https://submit.ncbi.nlm.nih.gov/subs/sra/
3.1 Submitter自动填充
3.2 General Information填写
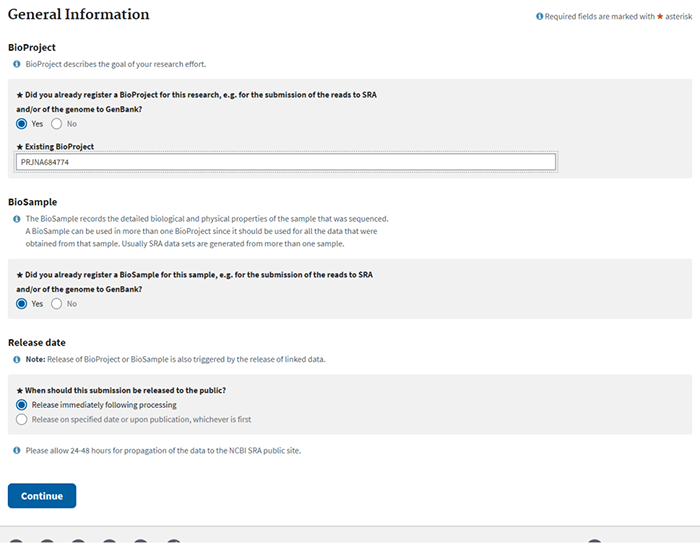
BioProject:填写已经创建好的BioProject号
BioSample:选择已经创建了BioSample
Release date: 选择释放数据的时间
3.3 SRA metadata信息上传
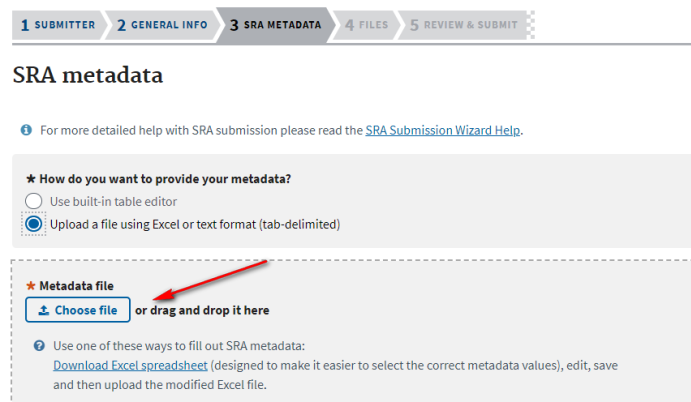
选择以excel的形式上传,表格内容如下:
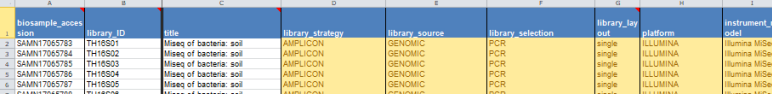
这里要根据自己的实际情况进行填写。
3.4 原始数据上传
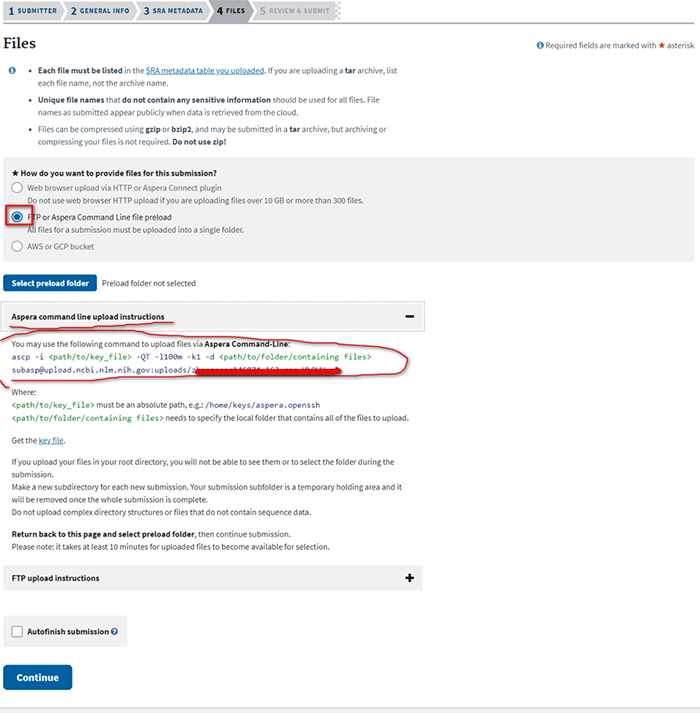
这里有FTP和Aspera Command-Line上传,在这里选择Aspera Command-Line上传(一是快,二是逼格高)
上传代码:
ascp -i ~/.aspera/connect/etc/asperaweb_id_dsa.openssh -QT -l100m -k1 -d ~/xx/16S/*.fastq subasp@upload.ncbi.nlm.nih.gov:uploads/xxxx_IAinE4W6/JSS_16S/
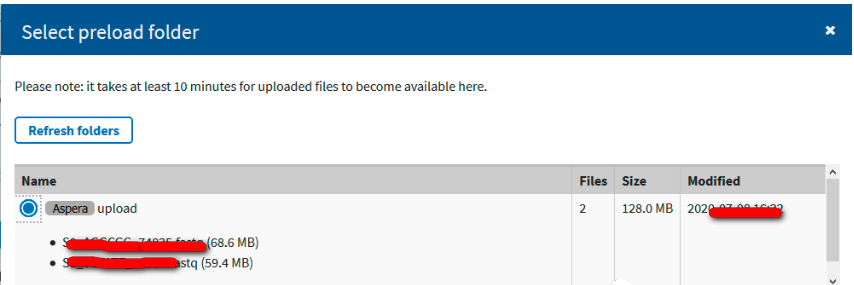
上传后选择select preload folder进入文件,刷新数据,需要点时间,文件也会慢慢的刷新出来:
3.5 提交

3.6 等待验证通过
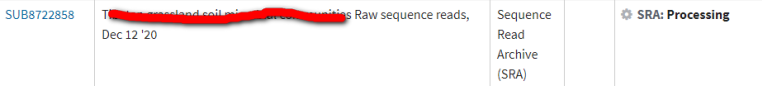
测序数据上传完成。NCBI会在后台审核数据,最后给出Accession Number,一般一天左右。
到此,整个数据的上传都已完成。
此文同步更新到github:https://github.com/songzhang-master/SRA_submit














