Lnc2Cancer 3.0由哈尔滨医科大学李霞老师和宁尚伟老师课题组开发,发表在2020年10月13日发表在Nucleic Acids Research杂志上。
数据库介绍 Lnc2Cancer 3.0 (http://www.bio-bigdata.net/lnc2cancer 或http://bio-bigdata.hrbmu.edu.cn/lnc2cancer ) 是更新版本,该数据库主要存储包括实验支持的人类癌症相关的长非编码RNA (lncRNAs)和环状RNA (circRNAs)与相关数据。也包括通过高通量RNA测序技术(RNA-seq)和单细胞RNA-seq(scRNA-seq)分析lncRNA表达的工具。 Lnc2Cancer 3.0更新了几个新的特征: (1)增加了肿瘤相关lncRNA数目,目前的版本包括9254个lncRNA-癌症关联条目,其中涉及2659个lncRNA和216个癌症亚型; (2)新增加了1049个实验支持的与癌症相关的circRNAs,其中涉及743个circRNAs和70个癌症亚型; (3)提供实验支持的癌相关lncRNAs和circRNAs调控机制,涉及microRNAs、转录因子(TF)、遗传变异、甲基化和增强子; (4)补充了实验支持的癌症相关lncRNAs和circRNAs的生物学功能,包括细胞生长、凋亡、自噬、上皮间充质转化(EMT)、免疫和编码能力。 (5)提供实验支持的癌症相关lncRNAs和circRNAs在转移、复发、循环、耐药性和预后方面的临床相关性。 此外,Lnc2Cancer 3.0还开发了两个在线工具,包括RNA-seq和scRNA-seq web工具,以实现对癌症中lncRNAs的快速、可定制的分析和可视化。Lnc2Cancer 3.0是一个对于阐明lncRNA、circRNA与癌症之间的关系有价值的资源。 Lnc2Cancer 3.0链接是http://www.bio-bigdata.net/lnc2cancer 或http://bio-bigdata.hrbmu.edu.cn/lnc2cancer ,在使用时小编建议使用第二个链接,因为第一个有事会遇到一些图片无法显示的情况,第二个链接的不存在这种问题。 使用方法
Browse浏览
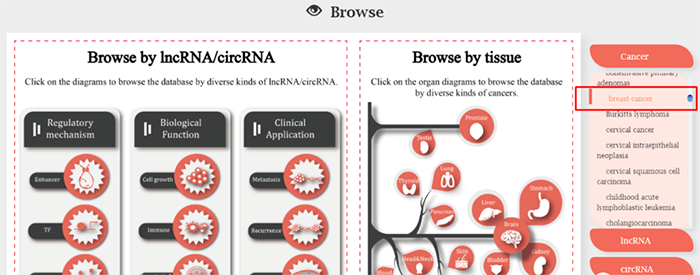
右侧侧边栏可以对癌型、lncRNA和circRNA随意点击浏览。

1. Cancer
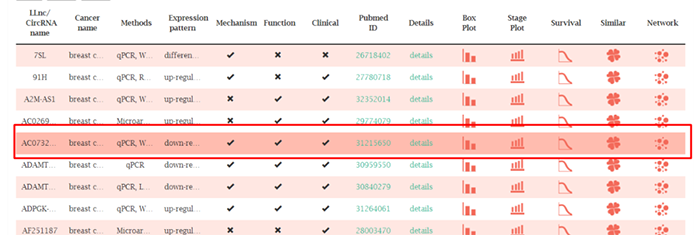
点击breast cancer
显示乳腺组织中的相关条目,比如该组织中涉及的lncRNA或circRNA,是以何种实验方法进行检测,在乳腺组织中是差异上调还是下调,是否提供关于其机制、功能和临床功能的相关信息,pubmed ID,是否提供详细说明,以及其相应的箱式图、stage图、生存曲线、相似性分析图和网络图
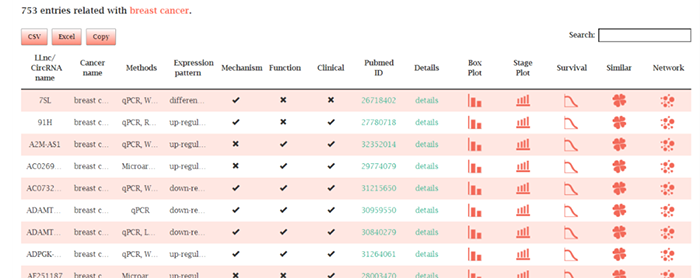
下面以该RNA为例进行展示:
(1) box plots :展示该癌型的肿瘤样本与正常样本比较所选的RNA的表达差异,使用wilcoxon秩和检验方法评估差异。
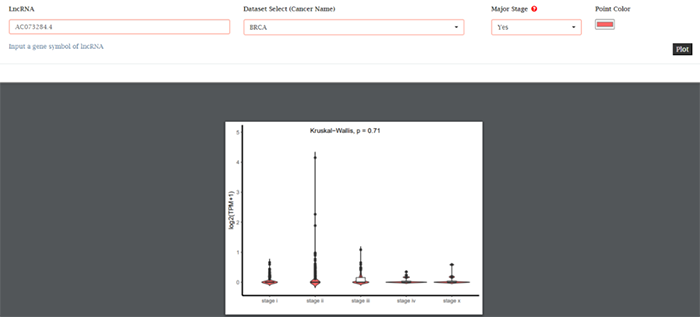
(2)Stage plot:该图展示不同临床病理学stage分组下所选的RNA的表达差异,使用Kruskal-Wallis test方法评估差异。
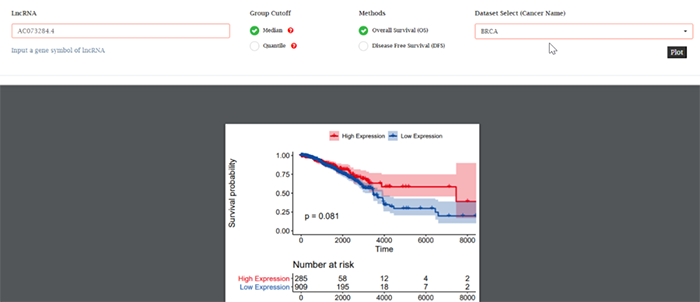
(3)Survival:基于所选RNA的表达均值将样本分成高低表达组, log-rank test检验两组样本OS或DFS差异
(4)Network:基于pearson相关性分析构建miRNA-lncRNA和mRNA-lncRNA 共表达网络
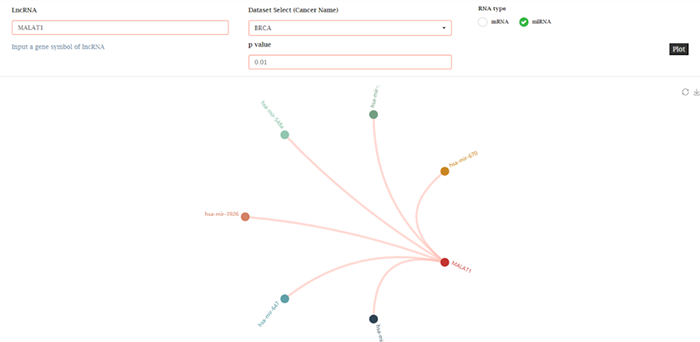
2. lncRNA
选择lncRNA浏览相关数据

3. circRNA
选择circRNA浏览相关数据
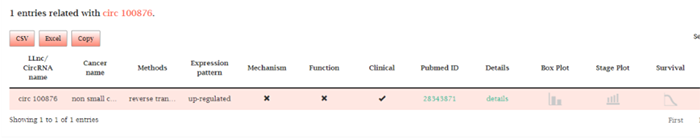
Search检索
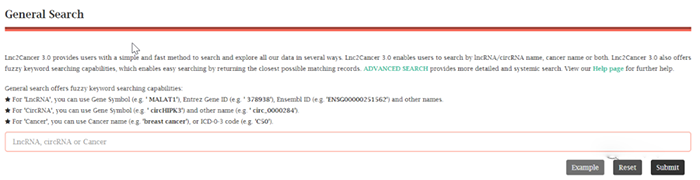
1. General Search通用检索
Lnc2Cancer 3.0可以通过多种方式搜索数据,可以通过lncRNA/circRNA名称、癌症名称或两者同时进行搜索。Lnc2Cancer 3.0还提供了模糊关键字搜索功能,通过返回最接近的可能匹配的记录,可以方便地进行搜索。
通用搜索提供模糊关键字搜索能力:(1)对于“LncRNA”,可以使用Gene Symbol(如MALAT1), Entrez Gene ID(如378938),Ensembl ID(如ENSG00000251562)等名称。(2)对于CircRNA,可以使用Gene Symbol (如 circHIPK3)和其他名称(如circ_0000284)。(3)对于癌型,可以使用癌症名称(如reast cancer)或ICD-0-3 code(如C50)。
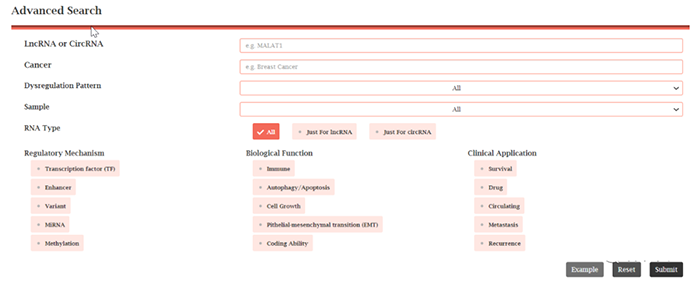
2. Advanced Search高级检索
高级搜索提供更详细和系统的搜索
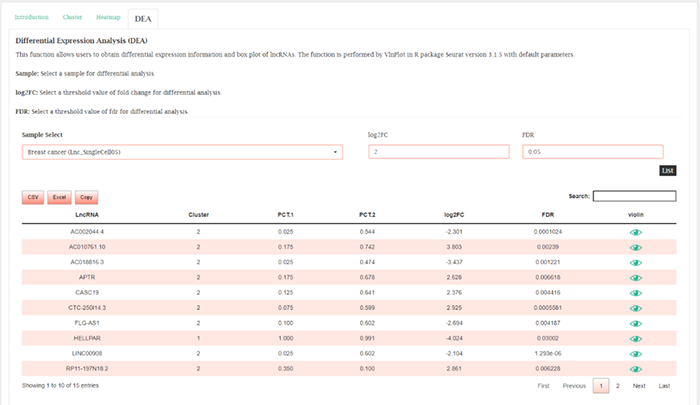
Single Cell Web Tools单细胞工具
single cell web工具提供了关键的交互式和可定制的功能,包括基于49个单细胞数据集的lncRNAs的一般信息、聚类、热图和差异表达分析。基于单细胞测序的49个与20种人类癌症相关的lncRNA表达数据集来自GEO和已发表的文献。
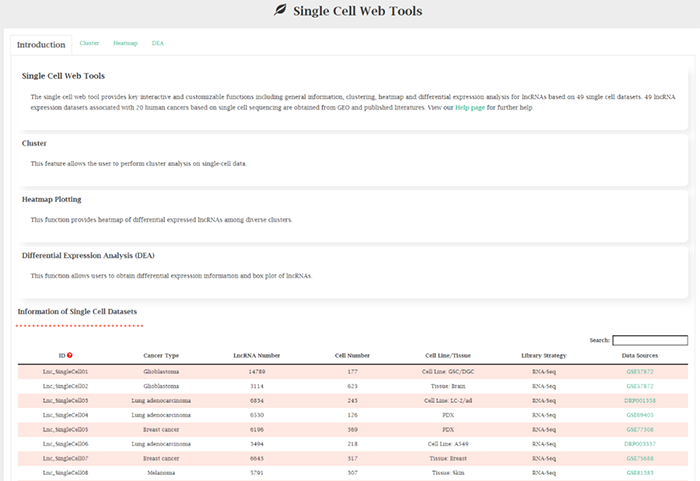
1. Cluster聚类
用户可以对单细胞数据进行聚类分析
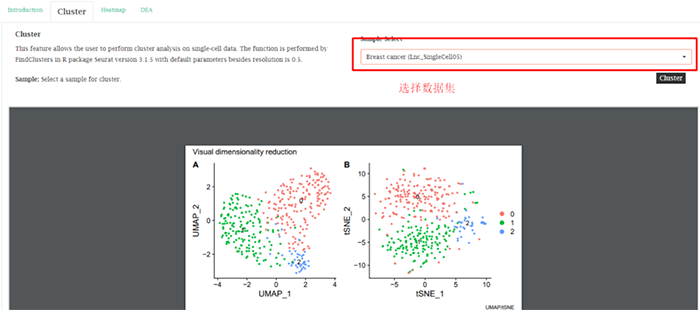
2. Heatmap Plotting绘制热图
对上述不同类之间识别差异表达lncRNAs,这里提供根据这些差异表达lncRNAs绘制的热图,使用R包Seurat中的DoHeatmap函数。
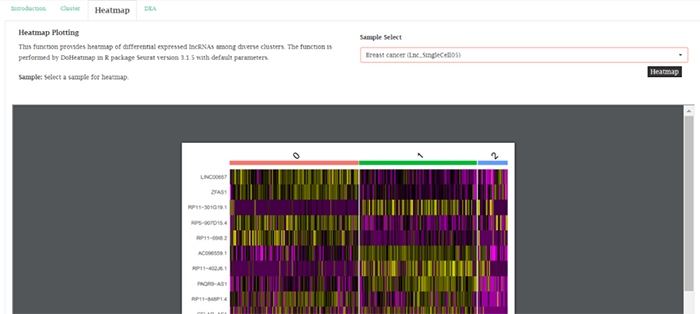
3. Differential Expression Analysis (DEA)差异表达分析
可以获得在自定义的log2FC和FDR阈值下差异表达的lncRNA信息和箱式图。
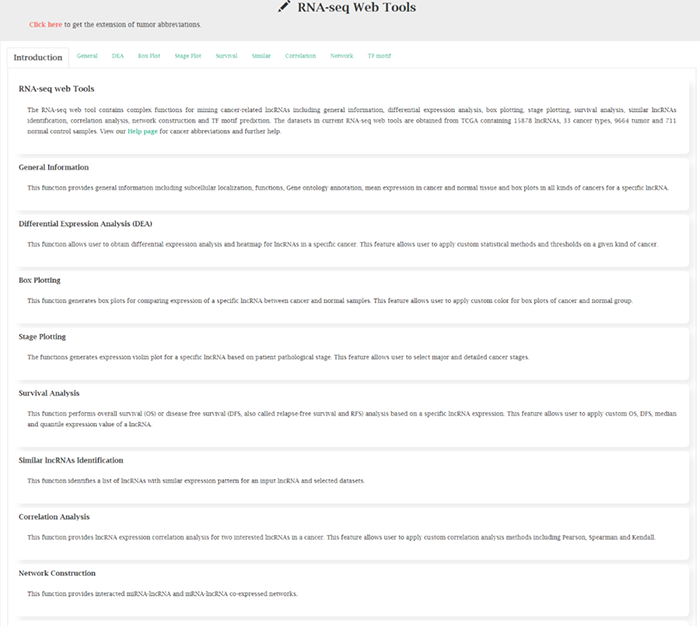
RNA-seq Web Tools RNA-seq工具
RNA-seq web工具包含挖掘癌症相关lncRNA等功能,包括一般信息、差异表达分析、box作图、stage作图、生存分析、识别相似lncRNA、相关性分析、网络构建和TF模体预测。目前RNA-seq web工具中的数据集来自TCGA,包含lncRNAs 15878个,癌症类型33个,肿瘤9664个,正常对照样本711个。
1.General Information一般信息
(前面介绍过了,这里不多介绍了)
2.Differential Expression Analysis (DEA) 差异表达分析
(前面介绍过了,这里不多介绍了)
3.Box Plotting 箱式图
(前面介绍过了,这里不多介绍了)
4.Stage Plotting临床stage绘图
(前面介绍过了,这里不多介绍了)
5.Survival Analysis生存分析
(前面介绍过了,这里不多介绍了)
6.Similar lncRNAs Identification识别相似lncRNA
这个功能能够识别出与输入的lucRNAs一组具有表达相似模式的lncRNA集合,限制相关系数>0.8,p<0.05。
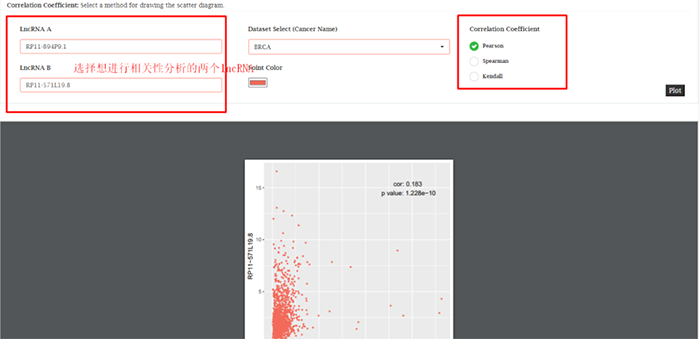
7.Correlation Analysis相关性分析
这里能够对感兴趣的两个lncRNA提供表达相关性分析。该功能允许用户应用自定义的相关分析方法,包括Pearson、Spearman和Kendall。
8. Network Construction网络构建
(前面介绍过了,这里不多介绍了)
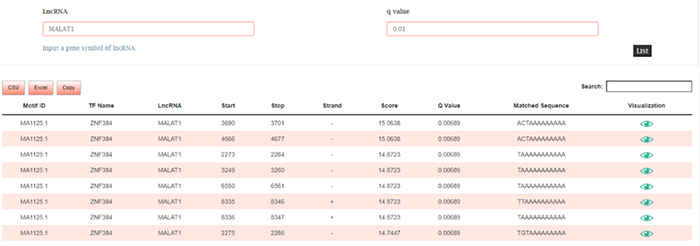
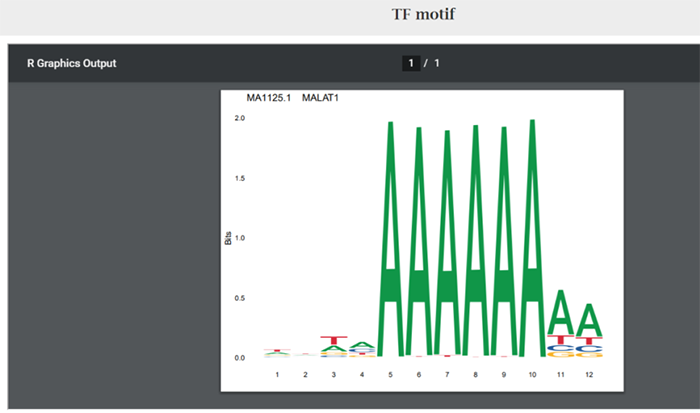
9. TF Motif PredictionTF模体预测
这个功能能够预测一个特定lncRNA的TF模体并提供由MEME Suite预测的TF和lncRNA的互作。提供了通过ggseqlogo包绘制的TF模体序列的LOGO图
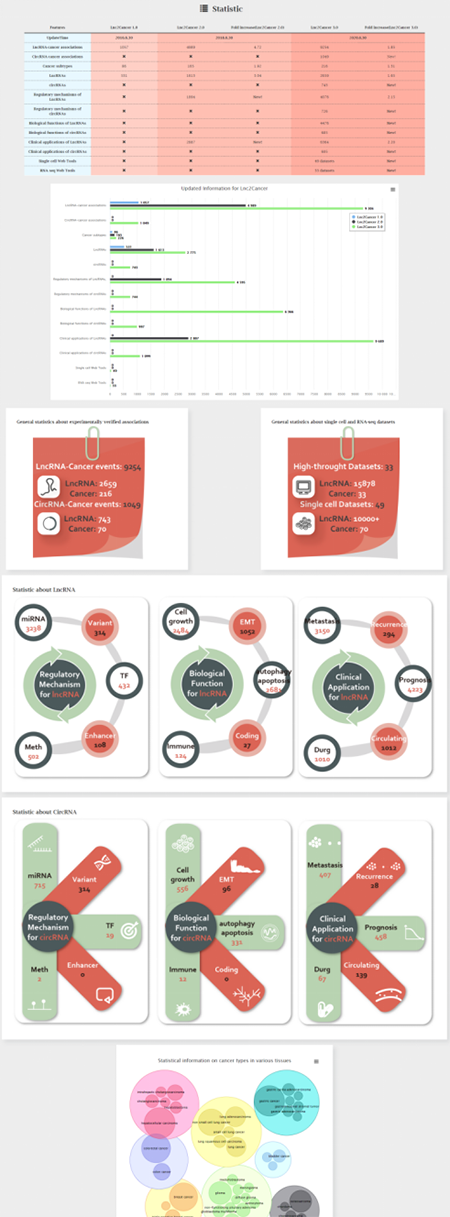
Statistic数据统计
这里提供了不同的Lnc2Cancer版本包含的数据量情况,以及各位数据更新量对比,实验验证的关联关系数目,单细胞测序数据和RNA-seq数据情况,不同作用机制、功能和临床关联下lncRNA的数目和circRNA数目,以及包括的癌型分类。
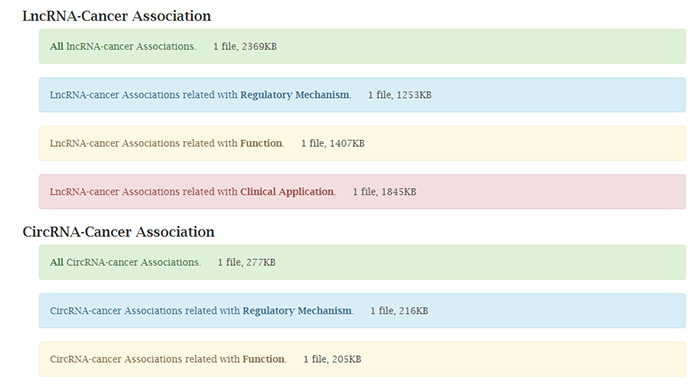
Download数据下载
可直接下载的数据如下: