1. 分析流程:
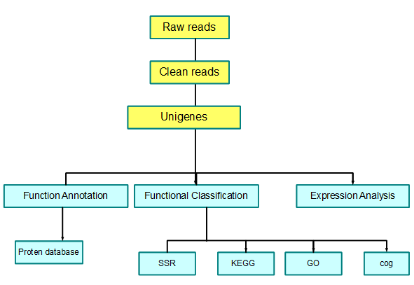
2. 分析步骤
2.1 数据预处理,质控过程,与有参考的转录组分析一致
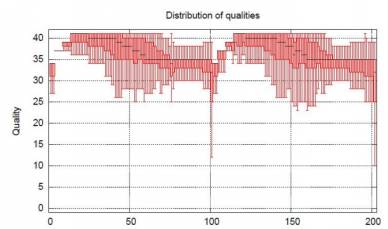
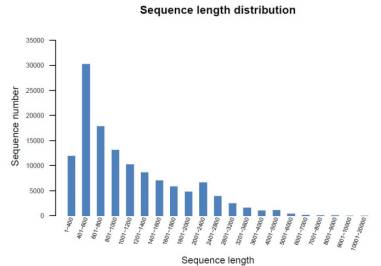
2.2 UniGene拼接目的:将预处理后reads进行拼接,得到拼接结果。
原理: 应用 de Bruijn graph path 算法对reads进行denovo拼接;对
上一步的拼接结果,再用Hamilton Path算法拼接。
结果:UniGene序列,UniGene统计信息,序列长度分布图
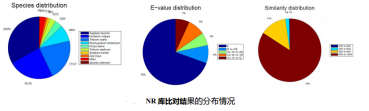
2.3 数据库注释
目的:对拼接得到的UniGene进行功能注释
原理:通过blast+算法将拼接得到的UniGene序列与数据库进行比对
结果:比对结果表格,物种分布统计和Evalue分布统计
2.4 UniGene表达分析
目的:UniGene定量分析。
原理:以UniGene为reference,分别将每个样本的reads进行reference mapping ,从而得到每个样本在每个UniGenes中的一个reads覆盖度,然后应用RPKM/FPKM标准化公式对富集片段的数量进行归一化。
RPKM:Reads Per Kilobase of exon model per Million mapped reads,公式下:
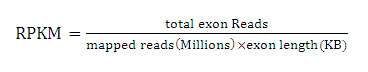
FPKM:Fragments Per Kilobase of exon model per Million mapped reads,公式下:
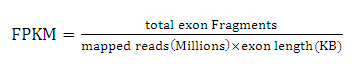
2.5 UniGene表达差异分析
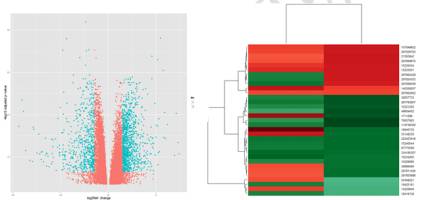
目的:对定量结果进行统计检验分析,找出差异表达UniGene
原理:双层过滤筛选差异基因
FC值筛选:采用Fold-change(FC),表达差异倍数进行第一层此的差异基因筛选
FDR检验:一般采用卡方检验中的fisher精确检验进行p值检验,采用Benjamini FDR(False discovery ratio)校验方法对p值进行假阳性检验,即,通过FDR显著性参数进行第二层次的差异基因筛选。
结果展示:
2.6 GO分析
利用数据库注释信息将 UniGene进行 GO 功能分类。
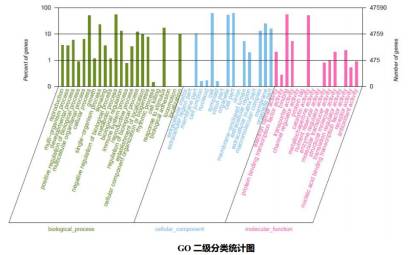
原理:利用数据库的注释结果,应用blast2GO算法进行GO功能分类,得到所 有序列在Gene Ontology 的三大类:molecular function, cellular component, biological process 的各个层次所占数目,一般取到14层。
结果:MF,BP,CC三大分类结果文件以及 UniGene2GO 关系列表,三大类别中第二层次上的柱状分布图和饼图,GO功能的层次分布图。
2.7 KEGG通路分析
对拼接得到 UniGene 进行 KEGG pathway 映射。
原理:应用KEGG KAAS在线 pathway比对分析工具对拼接得到的UniGene进行KEGG映射分析。
结果:标记的Pathway通路图。
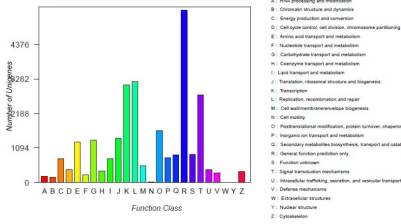
2.8 COG注释
对拼接得到 UniGene 进行 COG功能分类。
原理:利用blast+算法将拼接得到的UniGene与CDD库中的COG/KOG库进行比对,进行COG功能分类预测,将其映射到COG分类中。
结果: COG分类分布情况图。
2.9 SSR串联重复分析
对拼接得到 UniGene进行 SSR 简单重复序列的查找。
原理:筛选标准:单核苷酸重复的次数在10次或10次以上,二核苷酸重复的次数在 6次或6次以上,三至六核苷酸重复的次数在 5次或 5次以上。
同时,也筛选中间被少数碱基 (间隔小于100或等于100)打断的不完全重复的SSR。
结果:重复序列的信息文件以及统计文件。
2.10 lncRNA预测
对拼接得到的UniGene进行LncRNA(Long noncoding RNA)预测。
原理: 通过以下过程对UniGene进行过滤,最终得到候选LncRNA序列。
1) Unigene length > 200bp;
2) Unigene ORF(Open Reading Frame) length < 300;
3) 将满足长度条件的UniGene与多个近源物种进行进化分析,得到序列的保守性和进化特性;
4) 根据上述的特性和已知数据库中coding、noncoding区域的特性建立编码筛选模型;
5) 将符合noncoding模型的UniGene与Pfam等蛋白域数据库进行同源性比对,进一步去除可能的编码特性,最终得出LncRNA预测结果。














