目前对于人类微生物组研究已有大量体外培养和分子生物学技术,可以用来检测和分析微生物群落组成、物种多样性以及对人体细胞通路的影响。然而,在微生物生态学和流行病学的交叉领域,群体规模的微生物组研究设计和方法仍处于发展的早期阶段,需要充分考虑人类可变性、环境暴露和实验的可重复性。
现有微生物组研究技术与用于人类基因表达和全基因组关联研究技术有许多相似之处。人类相关微生物群通常根据其组成进行分析,例如,通过对16S rRNA基因进行测序,产生系统发育或分类谱。16S rRNA基因研究主要针对细菌,尽管高度敏感,但是容易受DNA提取和PCR扩增的影响。
宏基因组测序可以进一步描述整个群落的功能性遗传潜力,不能体现特定环境中这种遗传潜力的哪一部分被转录或翻译。宏转录组是使用群落所有RNA进行反转录建库测序,后续基本分析流程与宏基因组相似。宏代谢组则是提取群落总代谢物进行质谱检测。
宏转录组、代谢组学、和宏蛋白质组学技术可以将核苷酸序列图谱和生物活性产物联系起来。通过微生物rRNA基因测序、宏基因组学、宏转录组学和非靶向代谢组学等方法,结合“全微生物组关联分析(MWAS)”的核心策略,能精准解码菌群组成谱、功能谱和表达谱,挖掘关键生物标记物,进而阐明“菌群—宿主—环境(生态系统)”之间复杂的互作机制和因果关系。
许多不依赖培养的测序技术只能在属或种的水平上描述微生物群落,但并不是一个菌属/种内的所有菌株都具有同样的功能特性,特别是在致病性方面。例如E. coli有中性的,也有肠出血性大肠杆菌和益生菌,这种菌株间的变异对人类健康具有重要影响。传统认为无害的肠道共生菌Bacteroides vulgatus,也显示出很强的种内基因组变异性。
扩增子测序只会在高于核苷酸相似性阈值的类群之间进行区分(如97%),其鉴别菌株的能力是有限的,因为菌株关键的功能差异可能来自扩增基因之外的区域。
同样,宏基因组测序数据的组装策略也可能有意避免核苷酸水平变异。宏基因组测序分析精确识别菌株通常依赖于单核苷酸变异(SNVs,需要10倍以上测序深度)或识别可变区域两种方法。宏基因组测序通常只能准确鉴定复杂群落中任何一物种中的单一优势菌株,需要极高的测序深度才能区分次级菌株。
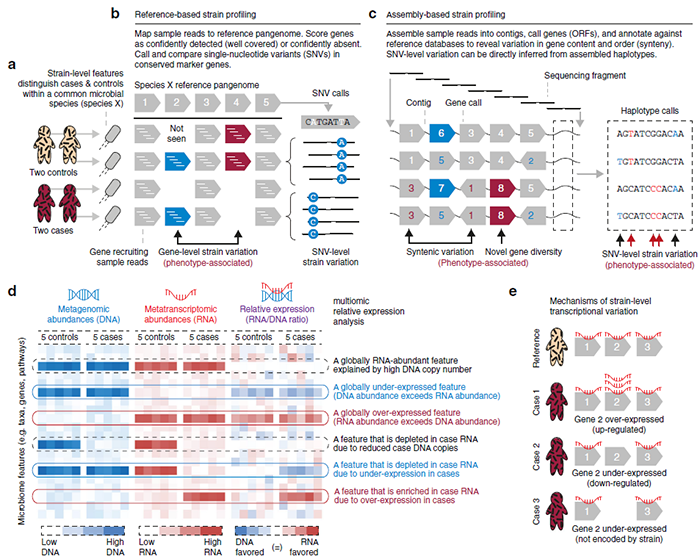
人类微生物组研究的菌株和功能的分析策略
分类学分析往往伴随着与群落组成及其基因和/或通路匹配的功能分析。宏基因组测序只能产生群落的潜在功能信息,如何种丰度的哪些生物可能执行怎样的生物过程(基因不一定是在当前条件下被转录)。
宏转录、蛋白是基于功能活性层面进行分析,更关注微生物群落的活性状态,可用于环境应激、药物处理等群落响应过程的时时研究。宏转录组还可以补充宏基因组研究RNA病毒的检测和罕见功能基因的量化等生物学信息。典型的宏转录组研究,如单微生物RNA-seq,包括转录本的定位组装、功能和分类信息的注释、数据标准化和差异表达分析等步骤。
但是宏转录组测序在流行病学规模上应用于人体微生物组研究仍然具有一定的困难。用于宏转录组的微生物组样本在收集时必须保留RNA,而且对于储存条件和时间更为敏感,RNA提取时对技术水平也有一定的要求。产生的宏转录组数据通常需要匹配的宏基因组数据来解释,否则DNA拷贝数的变化无法与转录活性的变化区分开来。
对于以扩增子为基础的宏转录组,16S rRNA基因的拷贝数变异、核糖体转录率差异以及16S rRNA转录丰度还没有确切的生物学解释。宏转录组表达谱的下游生物信息分析必须进一步解释RNA-seq实验相关分类组成变化和技术偏差。归一化后的差异基因表达分析也可以使用RNA-seq现有的分析工具。
非靶向代谢组学可能是目前可以解释生物活性机制最有效的方法之一,通过核磁共振(NMR)和质谱技术可以检测粪便、皮肤、循环代谢以及人类相关微生物群落中的小分子物质。在人体微生态中,超过10%的小分子可能是微生物来源的或经过微生物修饰的,因此人体微生物组研究需要将特定微生物菌株与代谢小分子相关联。目前相关研究设计在人口规模上的应用还很有限,哪些微生物群相关代谢物适合预测或调节人口健康还有待观察。
16S rRNA测序虽然可以方便地研究微生物群的组成,但目前对于菌群基因库认知不完全,限制了对有关代谢物影响的理解。宏基因组测序为现有的基因提供了更深入的了解,但这些基因中的大多数功能仍然未知。代谢物是最接近表型的组学,可以作为表型数据进行微生物群落的功能验证,微生物群落的特殊代谢产物还可能是疾病、药物治疗、环境治理等的分子标记。
如果想要进一步了解疾病相关微生物菌群的功能潜力,可以进行微生物组+代谢组多组学联合分析,在了解微生物多样性的同时,探寻微生物对机体生化和功能的影响。16S rDNA 测序、宏基因组测序与代谢组等多组学联用可在一定程度上克服单一组学研究的局限性, 在肠道微生物与健康疾病关系研究等方面取得众多进展,显示良好应用前景。
在所有基于扩增子、宏基因组测序等方法的微生物组流行病学研究中,目的都是确定微生物及分子特征丰度是否以及如何与样本特征(如供体健康、疾病状况或结果、饮食摄入、药物或环境)相关联。而微生物群落相关的数据通常由具有组成结构的计数(counts)组成,即微生物组数据通常表示为相对丰度,当进行经典的统计推断时,容易产生假阳性结果。
为了解决这些问题,几种分析方法被开发出来,这些方法都依赖于数据的归一化(normalization),主要区别在于计算p值时选择不同的数据转换和统计模型。归一化在差异丰度分析中至关重要,可以减少测序深度变化的影响。
宏基因组分析在输入原始计数(featureCounts)后,使用零膨胀高斯混合模型对取对数计数进行归一化和差异丰度分析,从而减少偏差。MaAsLin对微生物组数据进行反正弦平方根变换后,可以通过常规线性模型进行分析。一些RNA-seq数据差异表达分析工具,如edgeR、DESeq2和limma也常被用于微生物组研究。同时LEfSe、Metastats和ANCOM等非参数替代方法被广泛应用。
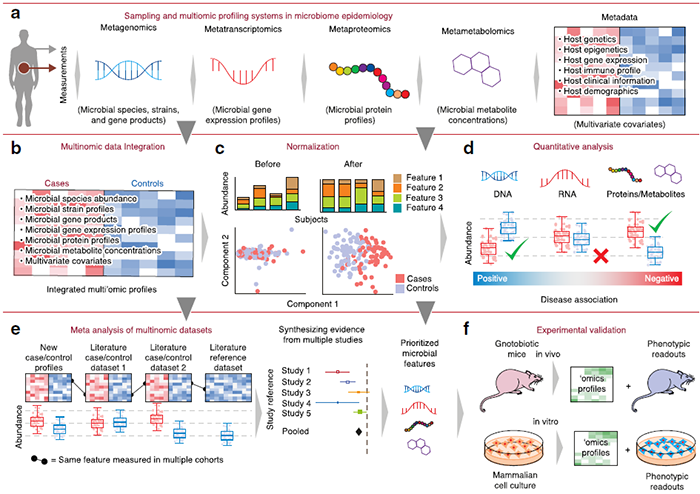
人类微生物组研究的菌株和分子功能分析策略
多组学整合关联的人体微生物组研究已经日益成为高分研究论文的首选:
(1)2019年发表在Cell Host & Microbe(IF 15.9)上的文章,研究通过“16S+宏基因测序+代谢组”学的方法,发现肥胖与微生物组组成、个别细菌类别造成的功能改变、以及与肠道微生物相关的血清代谢物的显著变化有关;T2D与肠道菌群的相关性并不明显;药物(如降压药和降糖药)以及膳食补充剂,与肠道菌群变化显著相关。总之,本研究将肥胖和D2T这两种相互关联的代谢疾病的微生物成分区分开来,确定了未来研究中需要考虑的饮食和药物用量。
(2)2020年发表在GeroScience(IF 6.4)上的文章,研究人员采用16S rRNA基因测序、代谢组检测、免疫因子检测等技术,对雄性Wistar大鼠的微生物组、代谢组、免疫因子与宿主寿命的相关性进行多组学整合关联研究,发现大鼠肠道微生物组和血清代谢组在衰老过程中发生了显著变化。
肠道菌群、血清代谢物、免疫因子的三者相互作用中存在重要的“关联对”,比如,与年龄相关的关键代谢物如4-羟脯氨酸、脯氨酸和赖氨酸聚类在一起,并与肠道菌群如Bifidobacterium、Lactobacillus、Akkermansia呈正相关。为了解衰老过程中肠道微生物组、血清代谢组、免疫因子之间的互作机制提供了新的见解。
(3)2020年发表在mSystems(IF 6.6)上的文章,通过“16S+宏基因组+靶标代谢组”检测运动员和不经常运动的人的粪便和尿液样本,比较两组样本之间代谢表型和肠道微生物的差异。发现,两组样本间表现出独特的代谢表型和微生物多样性差异。微生物多样性与高度坚持健康饮食习惯和身体锻炼相呼应,并与一系列不同的微生物衍生代谢物相关。
目前对微生物组的研究越来越复杂,已有多种基于序列、分子和培养的方法对群体规模的微生物组进行研究。为了将微生物数据与人类健康联系起来,必须考虑实验设计、数据分析和流行病学统计的问题。例如,将人群研究的数据转化为分子机制研究,以找出影响人体健康的菌群特性;菌群整合多组学研究中数据的获得、标准化和定量分析;了解实验设计因素、生物信息学数据分析和统计学的工具及其局限性。
总体来说,16S和宏基因组是针对DNA进行分析,探讨群落内的物种组成和潜在功能特征,分析的实验难度较低,比较容易实现,可作为基础探索,在微生物群落研究中,应用最为广泛。而多组学整合关联研究是将多种高通量检测研究策略,如(宏)基因组测序、(宏)转录组测序、蛋白质组定量检测和代谢组分析等,组合应用于对同一科学问题。
对于多组学测序数据,除了通过差异统计分析来筛选各组样本中的微生物标志物种或基因(Biomarker),还需要将转录组、代谢组、蛋白组、或者检测获得的理化指标、临床指标等各类数据,与微生物组数据进行关联分析,从而获得与各类指标变化相关联的特定微生物物种及其基因。通过多组学整合关联研究,我们能够在微生物物种/功能层面、以及宿主基因组/转录组/蛋白组/代谢组层面,同时全面筛选相关Biomarker,从而深入解析“微生物组—宿主—环境影响因素”的互相作用及其调控机制。
参考文献:
[1] Mallick H, Ma S, Franzosa EA, et al. Experimental design and quantitative analysis of microbial community multiomics. Genome Biol 2017;18:228.
[2] Aron-Wisnewsky J, Warmbrunn MV, Nieuwdorp M, et al. Metabolism and Metabolic Disorders and the Microbiome: The Intestinal Microbiota Associated With Obesity, Lipid Metabolism, and Metabolic Health: Pathophysiology and Therapeutic Strategies. Gastroenterology 2020.
文章来源:医学方














