最近又遇到的miRNA定量的问题,所以我又把之前的一些内容重新整理了一下,希望能对小伙伴有所帮助。
miRNA的实时定量PCR与普通mRNA的PCR有些不同,这主要是因为 miRNA的自身结构比较特殊。其最大的特点就是长度过于短小,仅仅二十几个碱基的核酸片段无法直接应用常规的 PCR 技术扩增。所以,在针对miRNA的PCR技术中,必须要延长待测miRNA 的长度,构建出一个足够长的PCR模板,才能进一步应用PCR技术 来定量分析。
目前常用的miRNA的定量的方法主要有两种。
颈环法:
茎环法的原理是,设计一种特殊的茎环结构的反转录引物,在反转录反应中直接完成模板链的延长。
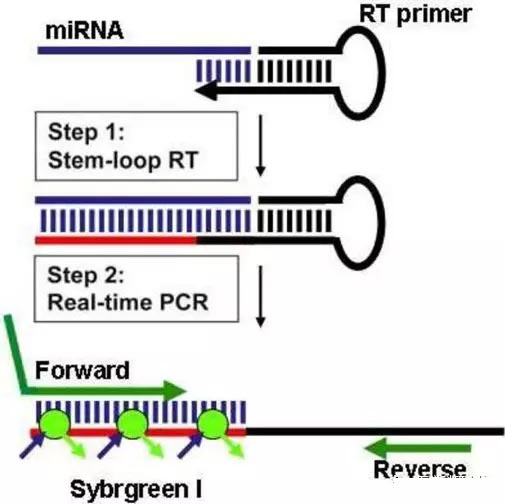
反转录引物设计:“茎环”即指反转录引物。茎环结构不但能有效地延长miRNA的长度,同时它自身互补的构象可以避免与其他同源基因结合,减少了非特异性扩增的几率。整个引物由2部分组成,一个通用的茎环结构和5~8个与目的miRNA的3'端反向互补的碱基。
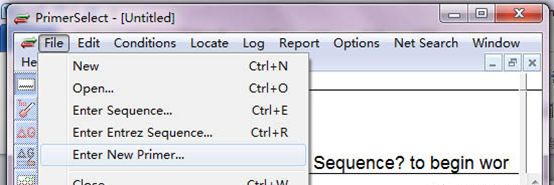
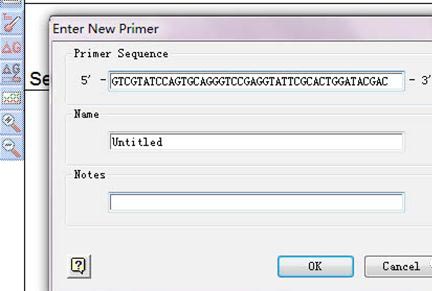
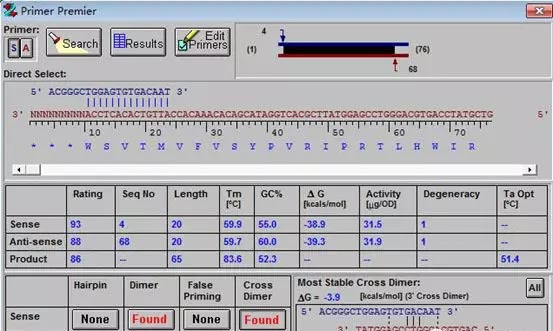
我们拿经典颈环序列GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGAC作介绍。打开DNAstar软件的PrimerSelect;file打开下拉菜单;打开Enter New Primer…;粘贴颈环序列;点击OK。
颈环结构已经输入,下面查看颈环结构会形成的那些发夹结构。
选择颈环结构(鼠标点一下);点击Report下拉菜单;选择Primer Hairpins。
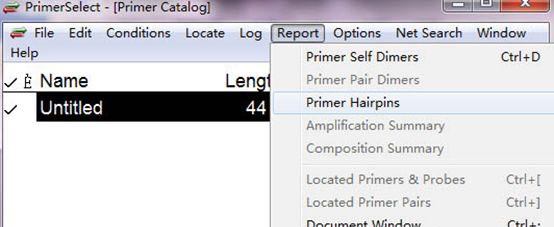
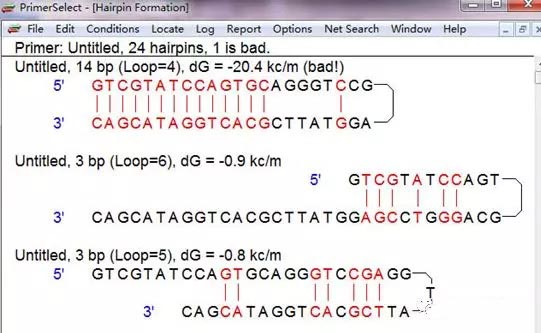
第一个发夹结构是实验所需的结构(dG=-20.4kc/m)。
下面结合has-miR-122-5P合成cDNA的具体过程讲解has-miR-122-5P:UGGAGUGUGACAAUGGUGUUUGU
将U转T,方便后面使用:TGGAGTGTGACAATGGTGTTTGT
miRNAs的颈环结构引物:是将miRNAs的3’端后6位碱基反向互补添加到经典颈环结构的3’端形成的结构。(自己根据后面图片想一下原因,加6个碱基为经验所授)
has-miR-122-5P的后6位:GTTTGT
has-miR-122-5P的后6位的反向互补序列:ACAAAC
颈环引物序列:
5’-GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACACAAAC-3’查看形成的发夹结构(方法前面有讲)
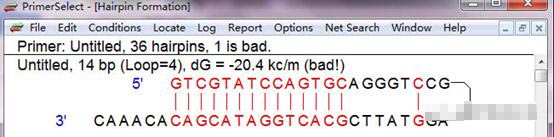
看一下miRNA和颈环引物在一起会是怎么样子:
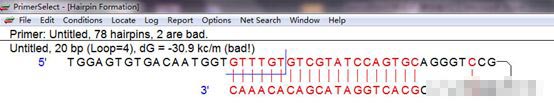
在含反转录酶及适当的条件下,miRNA和其颈环引物将会合成cDNA链:GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACACAAACACCATTGTCACACTCCA
查看cDNA结构:
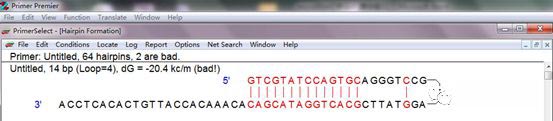
我们知道了cDNA的合成过程和序列,同时学习了miRNA的反转录过程。
下面进入重头戏,教大家如何进行miRNA引物设计。
首先如果你的前面实验是芯片的就在miRbase上再查看核对一遍,如果是测序实验的需要将miRNA的名字3p或5p及之前的部分复制到miRNA上查找完整序列。将得到的miRNA完整序列复制到一个excel表中,然后使用查找替换将U改为T(一定要记住改换,要不然primer5.0是不认U的)。
为了后面引物设计方便,需要以miRNA序列构建引物,所以需要将miRNA的cDNA的反向互补序列找出。使用primer5.0。
打开File下拉菜单;New;DNASequence。
由于正规设计过程中不会先把cDNA序列找出来,所以直接操作过程为先将miRNA序列(U改T)输入,再输入颈环结构序列的反向互补序列。
复制miRNA序列(U改T);点击primer5.0序列框,使用组合键“Ctrl+V”粘贴序列;选择正向序列。
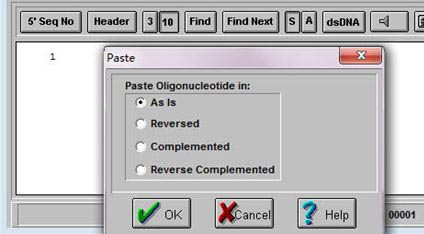
复制颈环结构序列;点击primer5.0序列框,使用组合键“Ctrl+V”粘贴序列;选择反向互补序列输入Reverse Complemented。
到这一歩cDNA的反向互补序列已经输入完毕,下一歩是在
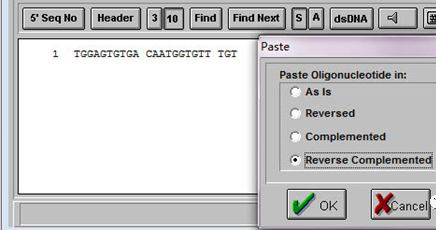
primer5.0上进行引物设计。
在序列前面输入9个N(这个是个人习惯,也有人是6个或以上的A,也有人是不输入就直接设计引物的),因为miRNA序列太短,很多时候不能设计出符合实验需要的miRNA,所以在设计正向引物时,一般需要加入3-6个碱基,最多时加入8个碱基,以满足正向引物的设计;
点击Function框下的Primer,开始设计上下游引物;
有经典颈环结构自然也就少不了常用的下游引物,一般使用CAGTGCAGGGTCCGAGGTAT;CGCAGGGTCCGAGGTATTC。同时还可以适当的在颈环序列中前后移动碱基设计下游引物;
自动跳出的显示框中默认选择的是下游引物,正好符合先将常用下游引物输入的操作。下游引物相当比较固定,先输入下游引物对整个引物设计能够提高设计效率。
点击框内的Edit Primers;选择所有引物序列;使用组合键“Ctrl+V”输入下游引物,输入方式为反向输入Reversed;点击OK;
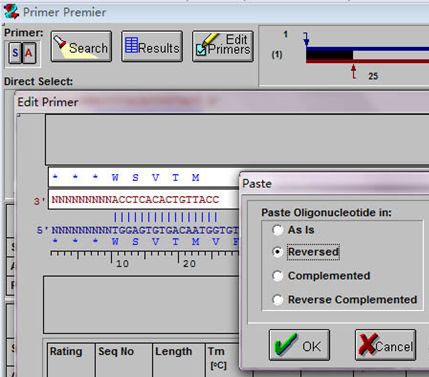
点击Analyze分析引物基本信息;点击Prime寻找引物在序列里结合部位;点击OK确定下游引物;
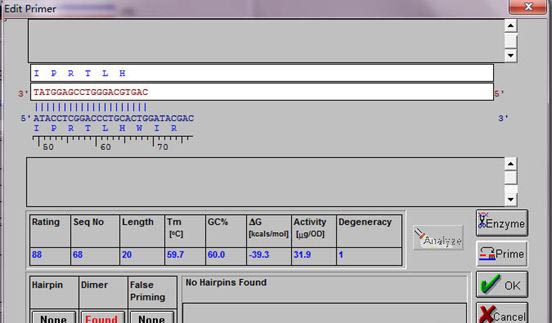
点击上下游转换键(图示),开始设计上游引物;点击Edit Primers;
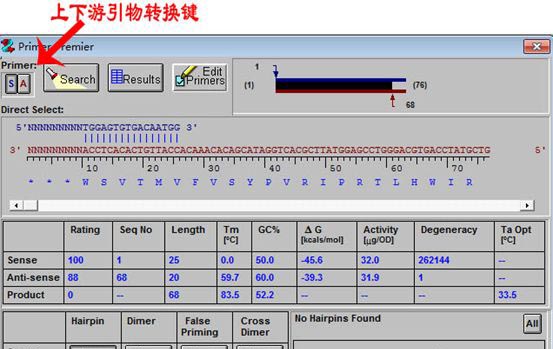
去除前面的9个N;点击Analyze;点击prime;查看前面不添加序列时的上游引物状况如何。
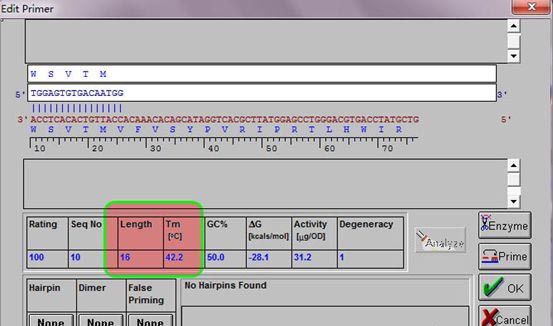
在这里的主要问题是Tm值太低,第二个问题是长度稍短。
绝大多数情况下在前边添加的序列以GC为主,且在加GC序列时如果序列中间不以AT隔开则TM值上升比值高,同理加AT序列。不要出现GCGC、ATAT、GCCG、或连续4个不同的核苷酸,这些失误会造成引物评分的降低。个人喜欢加CGGGC、GCGGGC、A/TGCCCG等。
例如加入ACGGGC(5’端加入,别在3’端);点击Analyze;点击primer;查看引物的长度和TM值差不多了就行。当然不是全行了,还要查看引物的自身颈环结构,自身引物配对,重要指标为dG。下游引物不用看了,因为是经典嘛。点击OK。
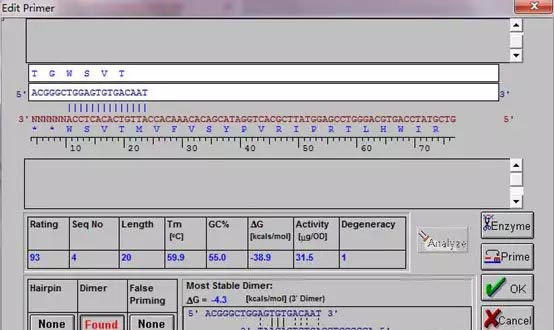
再查看一下引物对之间的匹对情况,加以对上游引物的修改,或者更换下游引物。有必要点击比对框下的All查看匹对情况。
加 PolyA 尾法
加尾法的原理则是先对miRNA进行加PolyA尾处理,改造成mRNA的结构,再采用mRNA常用的oligo(dT)反转录引物进行反转录,从而得到较长的模板链。
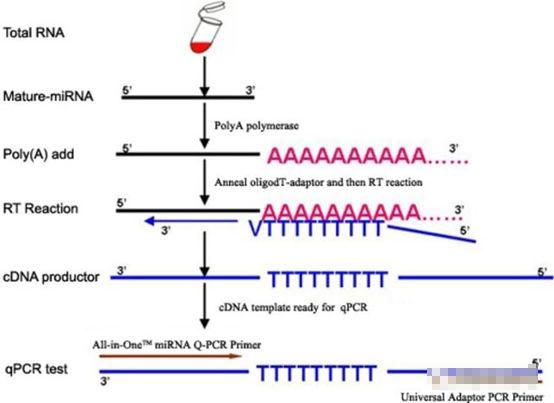
反转录引物:加尾法的反转录引物实际上就是处理mRNA所用的通用oligo(dT)引物在反转录前有一个步骤是为RNA样品加PolyA尾,然后反转录。所以它的反转录引物中包括:一段通用序列、一段多聚T和一个单碱基锚定。单碱基锚定的概念源于mRNA分析技术中的差异显示(Differential display)技术。其原理是借助mRNA的PolyA尾,对大量的mRNA进行分类。这种在多聚T后加上一个A或C或G的简并反转录引物,能将所有mRNA分类为3种,这就是单碱基锚定。给miRNA加上了PolyA尾后也可以用这种方法,设 计简并反转录引物。例如,5'-GCTGTCAACGATACGCTACGTAACGGCATGACAGTG( T)24 V(A\G\C)-3' 5'-GCTGTCAACGATACGCTACG-3'-对应的通用PCR下游引物或5'-GCGAGCACAGAATTAATACGACTCACTATAGG(T)12 V(A\G\C)N-3' 5'-GCGAGCACAGAATTAATACGAC-3'对应的通用PCR下游引物,V为锚定碱基。也就是说,研究者只需合成这3种反转录引物就足够了。在反转录时,将3种引物一同加入反应体系中,将所有序列反转录。
PCR扩增引物:加尾法的上游引物的设计相对简单,大多数只需直接将目的 miRNA的成熟序列中的U改为T。这时将模板序列上下游引物放到BeaconDesigner7中检验,在上游引物的两端适当地删减几个碱基,调整引物的GC比和Tm值,尽量保证不出现自身二聚体即可。加尾法不用考虑反转录引物与上游引物形成二聚体的问题。以hsa-mir-26a为例,可见上游引物设计只是将所有的U改为T而已,上游引物:5'-TTCAAGTAATCCAGGATAGGCT-3',成熟序列:5'-UUCAAGUAAUCCAGGAUAGGCU-3'。再以hsa-mir-21为例,不仅改变T外,还在3'端删去了一个A,上游引物:5'-TAGCTTATCAGACTGATGTTG-3',成熟序列:5'-UAGCUUAUCAGACUGAUGUUGA-3'。
两种方法的比较
无论是茎环法还是加尾法,其引物的设计最终要得到“一对半”,“一对”是指PCR扩增的上下游引物,“半”是反转录引物。这3个引物是完成整个实时定量实验所必须的。比较一下2种方法所设计出的引物,不难发现,加尾法的引物设计起来更加容易,当要测定的miRNA种类很多时,加尾 法的优势就更加明显了。在反转录引物上,加尾法应用的差异显示原理简并了3种引物,而茎环法则需要逐一设计相应的反转录引物,随着数量的增加,成本也会拉开差距。在PCR的扩增阶段,虽然两者都有通用的下游引物,但是,由于茎环法特殊的原理,设计上游引物时会比较麻烦,并且在真正的操作中难度还要大,因为目前的软件还不能自动设计超出模板链的引物,需要设计者将模板链与引物序列一同修改,操作繁琐。这一问题的出现,归根结底还是因为miRNA的长度过短所致。miRNA的序列和引物的序列都很短,几个碱基的变化就可能导致整个miRNA或引物的特性的改变,多数miRNA对2种方法都适用,但也有特殊情况。所以在大量检测miRNA时,加 尾法应是首选。另外,由于加尾法的特异性不如茎环法,故茎环法可作加尾法的候补方法,检测那些加尾法效果不理想的miRNA。
内参
除“一对半”的引物之外,实时定量还要设置内参。目前比较常用的是U6。U6是核小RNA,其序列较长,可直接进行扩增,反转录引物就是扩增的下游引物。如:
上游引物 5'-GCTTCGGCAGCACATATACTAAAAT-3'
下游引物 5'-CGCTTCACGAATTTGCGTGTCAT-3'或者
上游引物 5'-CTCGCTTCGGCAGCACA-3'
下游引物 5'-AACGCTTCACGAATTTGCGT-3'














