GO是Gene Ontology的简称,是基因功能国际标准分类体系。它旨在建立一个适用于各种物种的,对基因和蛋白质功能进行限定和描述的,并能随着研究不断深入而更新的语言词汇标准。GO分为分子功能(Molecular Function)、生物过程(Biological Process)、和细胞组成(Cellular Component)三个部分。
富集分析主要用于差异基因在GO term的富集程度。
GO term分为三大类,每一类从不同的层面解释基因的生物学功能,我们可以结合生物学问题的特殊性,有针对性的关注GO term。
GO富集分析的统计假设,并不能完全代基因功能的重要程度。要结合生物学问题、结合基因的功能注释,才能判断其中的基因变化是否有重要的生物学意义。
今天我们介绍我们在paper中经常看到的GO分析及可视化。(使用clusterProfiler包)。
代码如下:
第一步安装r包,对于3.6及以上的r的包安装方式参考(用高版本的R的人注意了)
BiocManager::install("clusterProfiler")BiocManager::install("org.Hs.eg.db")
library(org.Hs.eg.db)library(clusterProfiler)load("Desktop/go_genename.rda") ##加载数据gene <- mapIds(org.Hs.eg.db, geneNames, 'ENTREZID', 'SYMBOL') #转化id形式 all <- enrichGO(gene = gene, OrgDb = org.Hs.eg.db, ont = "ALL" , ##同时展示mf,bp和cc pAdjustMethod = "BH", pvalueCutoff = 0.01, qvalueCutoff = 0.05)
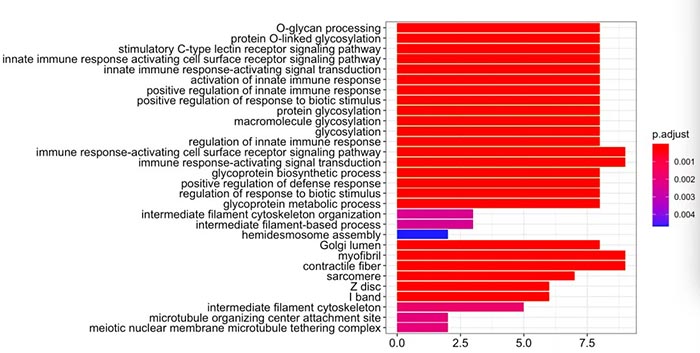
对结果做barplot展示,这个也是在paper中常看到的图。
barplot(all,showCategory=30)
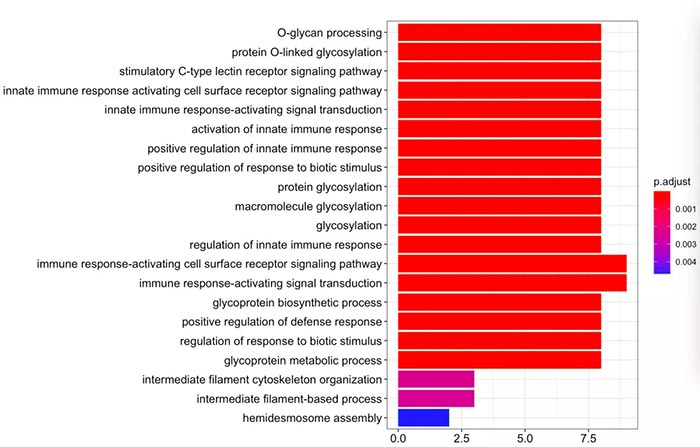
如果只展示bp,把ont参数给成BP即可。
BP <- enrichGO( gene = gene, OrgDb = org.Hs.eg.db, ont = "BP" , pAdjustMethod = "BH", pvalueCutoff = 0.01, qvalueCutoff = 0.05) barplot(BP, showCategory=30)
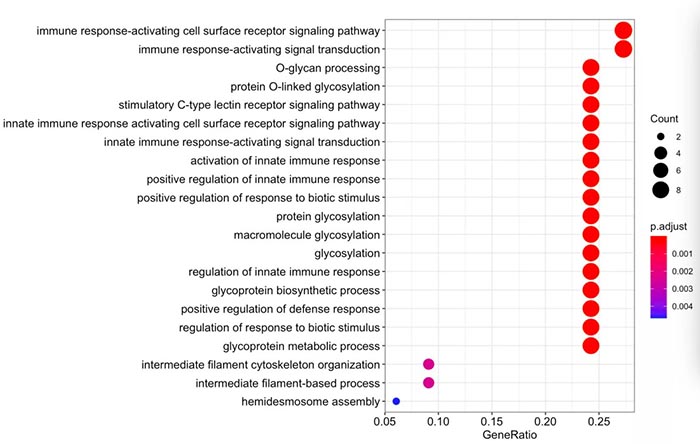
如果不喜欢barplot展示,也可以选择用dotplot展示。
dotplot(all,showCategory=30)
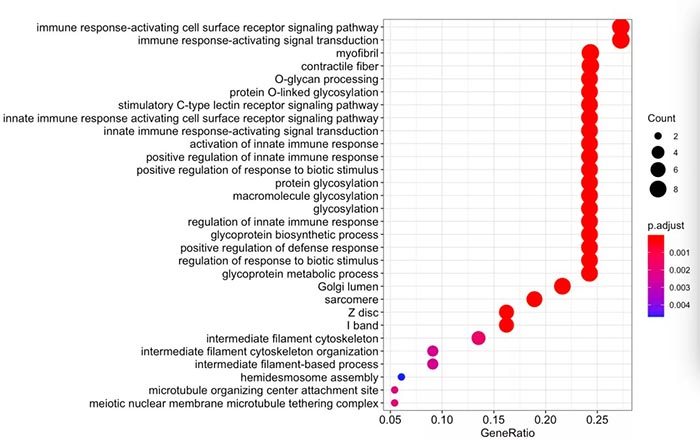
dotplot(BP, showCategory=30)